DAN BET CIPELANG
SKRIPSI
WIKE REISYA PERBA
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
BBIB Singosari, dan BET Cipelang. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D.
Salah satu gen yang diduga memiliki peranan penting terhadap sifat pertumbuhan, kualitas karkas, tingkat produksi, dan kualitas susu adalah gen Growth Hormone Receptor (GHR). Penelitian ini bertujuan untuk mengidentifikasi keragaman gen GHR pada sapi Friesian Holstein (FH) dan dibandingkan dengan sapi pedaging.
Sejumlah 126 sapi perah FH dan pedaging telah dikoleksi sampel darahnya. Sampel ini berasal dari 89 ekor sapi FH dari BIB Lembang (17), BBIB Singosari (32), dan BET Cipelang (40). Darah dari 37 ekor sapi pedaging berasal dari empat bangsa di BET Cipelang diambil sebagai pembanding, yaitu Simmental (13), Limousin (14), Brahman (5), dan Angus (5). Tingkat keragaman gen diidentifikasi dengan menggunakan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) menggunakan enzim restriksi AluI yang memotong pada situs AG|CT.
Panjang produk hasil amplifikasi fragmen GHR adalah 298 pb. Sapi FH dari ketiga lokasi bersifat polimorfik dengan dua alel, yaitu alel A dan G, serta memiliki tiga genotipe, yaitu genotipe AA, GG, dan AG. Genotipe AA memiliki tiga pita (50 pb, 81 pb, dan 167 pb), GG memiliki dua pita (131 pb dan 167 pb), dan genotipe gabungan AG (50 pb, 81 pb, 131 pb, dan 167 pb). Hasil yang sama diidentifikasi pada sapi pedaging. Kekecualian pada sapi Angus yang hanya memiliki genotipe AA dan AG, serta sapi Brahman yang bersifat monomorfik dengan genotipe AA (100%). Frekuensi alel A pada sapi FH dari ketiga lokasi lebih tinggi dibandingkan alel B dan genotipe tertinggi sapi FH adalah genotipe AA (0,719). Sapi FH BIB Lembang dan BBIB Singosari memiliki frekuensi genotipe AG dan GG yang seimbang (0,118 dan 0,125) sedangkan frekuensi genotipe AG pada sapi FH BET Cipelang sedikit lebih tinggi dibandingkan GG (0,200 dan 0,125). Frekuensi alel A tertinggi ditemukan juga pada sapi pedaging kecuali Simmental. Frekuensi genotipe AA tertinggi ditemukan pada sapi Limousin dan Brahman, namun frekuensi genotipe AG tertinggi ditemukan pada Simmental dan Angus.
Analisis Chi-Kuadrat menunjukkan bahwa sapi perah FH tidak berada dalam keseimbangan Hardy-Weinberg (χhit2>χ0,052). Hal ini berkebalikan dengan sapi
Simmental dan Limousin, sedangkan nilai Chi-Kuadrat sapi Brahman dan Angus tidak dapat dihitung. Tingkat heterozigositas pada sapi FH tergolong rendah (ho<he).
Sapi Angus memiliki nilai heterozigositas tertinggi (0,600) dan terendah sapi Brahman (0,000) pada sapi pedaging. Keragaman gen GHR|AluI dapat dijadikan informasi dasar untuk melakukan seleksi pada sifat pertumbuhan pada sapi FH dan sapi pedaging.
and BET Cipelang.
Perba, W. R., C. Sumantri, and A. Anggraeni
Growth hormone receptor (GHR) is one factor affecting animal growth. GHR is required by growth hormone (GH) to carry out its effect on target tissues. The objective of this study was to identify polymorphism of the Growth Hormone Receptor gene in Holstein Friesian (HF) dairy cattle and beef cattle. Genotyping was performed on 126 animals. These consisted of HF 89 heads from BIB Lembang (17), BBIB Singosari (32), and BET Cipelang (40); as well as for 37 heads of four breeds of beef cattle including for Simmental (13), Limousin (14), Brahman (5), and Angus (5) for a comparison. Varian genetic of the GHR gene was identified by PCR-RFLP method using AluI restriction enzyme cutting the base position at AG|CT. Single nucleotide polymorphism (SNP) had been found in exon 10, coding for the cytoplasmic domain of GHR, which was located at position 81 bp (A/G) induced amino acid substitutions Ser/Gly. The product of amplification of GHR was 298 bp. Genotyping the GHR gene produced two alleles (A and G) resulting three genotypes (AA, AG and GG). The genotype AA was dominant in HF cattle from all locations, and also in Brahman and Limousin cattle from BET Cipelang. All of HF dairy cattle were in Hardy-Weinberg disequilibrium (χhit2>χ0,052), in contrast, Simmental and
Limousin were in Hardy-Weinberg equilibrium. The Hardy-Weinberg in Brahman and Angus values could not be calculated. Heterozigosity values in HF and beef cattle were low (ho<he). In beef cattle, Angus had highest heterozigosity value
(0,600), while Brahman had the lowest heterozigosity value (0.000). The conclusion was that the variability of GHR|AluI gene can be used as a basic information for selection in growth caracteristic in the HF and beef cattle.
DAN BET CIPELANG
WIKE REISYA PERBA D14070081
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Nama : Wike Reisya Perba NIM : D14070081
Menyetujui,
Pembimbing Utama, Pembimbing Anggota,
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) (Ir. Anneke Anggraeni, M.Si, Ph.D.) NIP: 19591212 198603 1 004 NIP: 19630924 199803 2 001
Mengetahui: Ketua Departemen,
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) NIP: 19591212 198603 1 004
Penulis adalah anak kedua dari tiga bersaudara pasangan Bapak Idham Suhaimi,
S.ST dan Ibu Eti Setiawati.
Penulis mengawali pendidikan dasar tahun 1995 di Sekolah Dasar Negeri 4
Kepahiang, Bengkulu. Penulis pindah pada tahun 1996 ke Sekolah Dasar Negeri 1
Sembawa (sekarang SD Negeri 9 Sembawa), Palembang yang diselesaikan pada
tahun 2001. Pendidikan lanjutan tingkat pertama dimulai pada tahun 2001 dan
diselesaikan pada tahun 2004 di Sekolah Menengah Pertama Negeri 3 Banyuasin III.
Penulis melanjutkan pendidikan di Sekolah Menengah Atas Plus Negeri 2 Banyuasin
III pada tahun 2004 dan diselesaikan pada tahun 2007.
Penulis diterima di Institut Pertanian Bogor pada tahun 2007 melalui jalur
Undangan Seleksi Masuk IPB (USMI). Penulis diterima pada tahun 2008 di
Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan. Penulis
aktif dalam mengikuti kegiatan Himpunan Mahasiswa Ilmu Produksi dan Teknologi
Peternakan (HIMAPROTER). Penulis pernah menjadi panitia Dekan Cup 2009 serta
Kontes Bibit dan Seni Ketangkasan Domba Garut nasional 2010. Penulis aktif dalam
Organisasi Mahasiswa Daerah Ikatan Keluarga Mahasiswa Bumi Sriwijaya
(IKAMUSI) sebagai pengurus Departemen Informasi dan Komunikasi periode
2007-2008. Pernah mengikuti kegiatan magang di Balai Pembibitan Ternak Unggul
(BPTU) Sembawa pada tahun 2009. Penulis berkesempatan menjadi penerima
dan karunia yang dilimpahkan, sehingga penulis dapat menyelesaikan penelitian dan
penulisan skripsi ini. Salawat serta salam semoga senantiasa terlimpah dan tercurah
kepada Rasulullah Muhammad SAW, serta para sahabat, keluarga dan pengikutnya.
Skripsi yang berjudul Identifikasi Keragaman Gen Growth Hormone Receptor pada
Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang adalah
karya ilmiah yang merupakan salah satu syarat untuk memperoleh gelar sarjana pada
Fakultas Peternakan, Institut Pertanian Bogor.
Produktivitas sapi perah di dalam negeri masih berpotensi untuk terus
ditingkatkan. Beberapa upaya dapat dilakukan untuk meningkatkan produktivitas
ternak diantaranya dengan perbaikan pakan, manajemen pemeliharaan, seleksi, dan
persilangan. Seleksi pada ternak bisa dilakukan pada level DNA dan terhadap
kandidat gen (marka) terutama yang terkait dengan sifat yang bernilai ekonomis. Gen
GHR|AluI adalah gen yang berhubungan dengan ekspresi hormon pertumbuhan yang
berperan pada pertumbuhan dan perkembangan longitudinal pascanatal,
pertumbuhan jaringan, laktasi, reproduksi, serta metabolisme karbohidrat, protein,
dan lemak.
Tujuan dari penelitian ini adalah untuk memberikan informasi mengenai
keragaman gen GHR|AluI pada sapi perah FH yang dibandingkan dengan empat
bangsa sapi pedaging. Informasi keragaman genetik yang diperoleh dari gen
GHR|AluI diharapkan menjadi dasar seleksi berdasarkan penciri DNA terutama pada
sifat pertumbuhan dan produksi susu pada sapi FH dan sapi pedaging. Penulis
berharap semoga skripsi ini dapat bermanfaat dan memberikan sumbangan terhadap
kemajuan dunia peternakan di Indonesia. Amin.
Bogor, 3 Mei 2011
RINGKASAN ... i
ABSTRAK ... ii
LEMBAR PERNYATAAN ... iii
LEMBAR PENGESAHAN ... iv
RIWAYAT HIDUP ... v
KATA PENGANTAR ... vi
DAFTAR ISI ... vii
DAFTAR TABEL ... ix
DAFTAR GAMBAR ... x
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 2
TINJAUAN PUSTAKA ... 3
Sapi ... 3
Sapi Friesian Holstein ... 3
Sapi Pedaging ... 4
Simmental ... 4
Limousin ... 5
Brahman ... 5
Angus ... 5
Gen Pertumbuhan ... 6
Gen Growth Hormone Receptor ... 7
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) ... 10
Keragaman Genetik ... 11
MATERI DAN METODE ... 13
Lokasi dan Waktu ... 13
Materi ... 13
Sampel Darah ... 13
Ekstraksi DNA ... 14
Primer ... 14
Amplifikasi DNA ... 14
Analisis PCR-RFLP ... 14
viii
Ekstraksi DNA ... 15
Amplifikasi DNA ... 16
Analisis PCR-RFLP ... 16
Elektroforesis DNA Total dan Produk PCR-RFLP ... 16
Analisis Data ... 17
Frekuensi Genotipe dan Alel ... 17
Keseimbangan Hardy-Weinberg (HW) ... 17
Heterozigositas ... 18
HASIL DAN PEMBAHASAN ... 19
Amplifikasi Fragmen Gen GHR|AluI ... 19
Frekuensi Genotipe dan Alel Gen GHR|AluI ... 23
Keseimbangan Hardy-Weinberg ... 25
Heterozigositas ... 27
KESIMPULAN DAN SARAN ... 29
Kesimpulan ... 29
Saran ... 29
UCAPAN TERIMA KASIH ... 30
DAFTAR PUSTAKA ... 31
1. Jumlah DNA dan Asal Pengambilan Sampel Darah yang
Digunakan dalam Penelitian ... 13
2. Frekuensi Genotipe dan Alel ... 24
3. Keseimbangan Hardy-Weinberg ... 26
1. Sapi FriesianHolstein ... 4
2. Rekonstruksi Struktur Gen GHR Berdasarkan Sekuens Gen GHR
di Gen Bank (Kode Akses. EF207442) ... 7
3. Visualisasi Hasil Amplifkasi Ruas Gen GHR|AluI pada Gel
Agarose 1,5% ... 19
4. Fragmen Gen GHR|AluI Didasarkan pada Sekuens Gen GHR di
Gen Bank (Kode Akses. EF207442) ... 20
5. Visualisasi PCR-RFLP Ruas Gen GHR|AluI pada Gel Agarose 2% 21
6. Keragaman Gen GHR|AluI pada Sapi Friesian Holstein dan Sapi
1. Sekuens Gen Growth Hormone Receptor (GHR) pada Sapi yang
Diakses di Gen Bank (Kode Akses. EF207442) ... 37
2. Modifikasi Metode Ekstraksi DNA Menggunakan Metode
PENDAHULUAN Latar Belakang
Sapi merupakan ternak yang berperan penting sebagai sumber daging, susu,
tenaga kerja, dan kebutuhan manusia lainnya. Fluktuasi produksi dan kualitas susu
segar sapi perah masih menjadi kendala utama dalam memenuhi permintaan nasional
seperti tercermin dari angka impor susu dalam negeri yang tinggi, sekitar 70%.
Upaya untuk meningkatkan produksi susu sapi perah melalui perbaikan pakan,
manajemen pemeliharaan, seleksi, dan pemuliaan perlu dilakukan untuk menekan
nilai impor.
Ketersediaan bibit berkualitas akan meningkatkan produksi peternakan dalam
negeri yang dapat ditempuh melalui perbaikan genetik ternak yang ada. Pemerintah
berupaya untuk meningkatkan dan mengembangkan populasi dan produksi ternak
seperti yang telah dilakukan oleh berbagai institusi unit pelaksana teknis dari
Direktorat Jendral Peternakan. Salah satu upaya yang dilakukan adalah dengan
menyediakan semen beku dalam bentuk straw di BIB Lembang dan BBIB Singosari,
serta calon embrio dari BET Cipelang. Bibit yang disediakan dari balai-balai tersebut
berasal dari induk/tetua yang berkualitas dan merupakan hasil seleksi untuk
menghasilkan bibit yang unggul.
Sapi Friesian Holstein (FH) merupakan bangsa sapi perah yang paling banyak
dibudidayakan di dunia. Peternakan sapi perah FH di Indonesia masih terpusat di
pulau Jawa. Upaya untuk meningkatkan produksi susu sapi FH salah satunya dapat
dilakukan dengan perbaikan genetik melalui seleksi. Perkembangan teknologi
molekuler telah sangat membantu program seleksi. Teknologi dapat dilakukan
melalui aplikasi Marker Assisted Selection (MAS) melalui analisis keragaman DNA
dengan menggunakan metode PCR-RFLP, PCR-SSCP, DGGE, dan analisis sekuen.
Program seleksi konvensional yang biasanya dilakukan dengan mengembangbiakkan
ternak terlebih dahulu untuk mendapatkan keturunan, dan kemudian dapat
mengetahui sifat unggul yang dimiliki oleh tetua tersebut, membutuhkan waktu yang
lama. Penciri DNA yang banyak dikembangkan saat ini dapat digunakan untuk
mendeteksi sifat unggul seekor ternak dalam waktu yang relatif lebih cepat dan
Sifat pertumbuhan ternak dipengaruhi oleh beberapa gen pertumbuhan seperti
oleh gen growth hormone receptor (GHR). Gen GHR memiliki fungsi sebagai
mediasi gen pertumbuhan yang mempengaruhi sifat pertumbuhan karkas pada sapi
pedaging dan sifat produksi susu pada sapi perah. Keragaman gen GHR diharapkan
dapat dijadikan sebagai informasi dasar sebagai gen mayor yang mempengaruhi
tingkat produksi ternak.
Tujuan
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen GHR
menggunakan enzim restriksi AluI (GHR|AluI) dengan metode PCR-RFLP pada sapi
FHdi BIB Lembang, BBIB Singosari, dan BET Cipelang, dan dibandingkan dengan
TINJAUAN PUSTAKA Sapi
Sapi termasuk dalam filum chordata, yaitu hewan-hewan yang memiliki
tulang belakang, kelas Mamalia atau menyusui, ordo Artiodaktil, yaitu berkuku atau
berteracak genap, sub ordo Ruminansia atau pemamahbiak, family Bovidae dengan
tanduk berongga, dan genus Bos sebagai pemamah biak berkaki empat. Spesies sapi
terbagi dua, yaitu Bos taurus merupakan bangsa sapi yang ada dan tidak berpunuk
dan Bos indicus merupakan sapi-sapi yang memiliki punuk (Blakely dan Bade,
1998).
Tipe berpunuk (Zebu) tersebar di wilayah Asia dan Afrika Selatan, sedangkan
tipe tidak berpunuk (Taurine) termasuk dalam spesies Bos taurus yang tersebar di
wilayah Eropa dan Afrika Barat. Perbedaan antara Bos indicus dan Bos taurus telah
dibuktikan dari DNA mitokondria dari dua spesies tersebut lebih dari 1.000.000
tahun yang lalu (Loftus et al., 1994; Bradley et al., 1996.). Kedua spesies ini
kemungkinan besar didomestikasi secara bebas (Loftus et al. 1994) sekitar 10.000
tahun sebelum sekarang.
Sapi Perah Friesian Holstein
Sapi Friesian Holstein (FH) menurut Blakely dan Bade (1998)adalah bangsa
sapi perah yang menonjol di Amerika Serikat dengan jumlah cukup banyak, sekitar
80%-90% dari seluruh sapi perah di Amerika Serikat. Sapi ini berasal dari Negeri
Belanda yaitu di propinsi North Holand dan West Friesland, yang memiliki padang
rumput yang bagus. Produksi susu sapi FH yang tinggi dimanfaatkan untuk
pembuatan keju sehingga tujuan seleksi diutamakan untuk mendapatkan sapi FH
yang memiliki sifat produksi susu yang tinggi.
Bangsa sapi FH murni memiliki warna bulu hitam dan putih (blackHolstein)
atau merah dan putih (red Holstein) dengan batas-batas warna yang jelas, seperti
pada dahi umumnya terdapat warna putih berbentuk segitiga dan bulu kipas ekor,
bagian perut serta kaki dari taracak sampai lutut (knee) atau (hock) berwarna putih.
Sapi Friesian Holstein memiliki tanduk yang pendek dan mengarah ke depan
Keunggulan sapi FH adalah jinak, tidak tahan panas, tetapi sapi ini mudah
menyesuaikan diri dengan keadaan lingkungan, dan lambat dewasa (Blakely dan
Bade, 1998). Dwiyanto et al. (2000) menyatakan bahwa produksi susu sapi perah di
Indonesia untuk daerah dataran tinggi berkisar antara 3000-3900 liter per laktasi. Hal
ini menyamai performans sapi perah Bos taurus yang dipelihara di sejumlah daerah
tropis yang berkisar antara 3500-4500 liter perlaktasi, sementara produksi susu sapi
perah FH di daerah iklim sedang mencapai lebih 6000 liter per laktasi.
Gambar 1. Sapi Friesian Holstein
Sapi Friesian Holstein yang baik adalah sapi yang memiliki tubuh luas ke
belakang seperti gergaji, sistem dan bentuk perambingan baik, puting simetris, dan
efisiensi pakan yang dialihkan untuk produksi susu tinggi. Saat ini, di Indonesia sapi
Friesian Holstein paling banyak dimanfaatkan sebagai sapi perah. Selain pemakaian
sapi perah sebagai jenis murni, di Indonesia juga terdapat jenis baru yang disebut
sapi Grati yaitu hasil persilangan antara FriesianHolstein dan sapi lokal (peranakan
Ongole) (Blakely dan Bade, 1998).
Sapi Pedaging
Sapi pedaging atau sapi potong memiliki keunggulan dalam menghasilkan
karkas berkualitas dan tingkat pertumbuhan yang tinggi. Beberapa jenis sapi yang
memiliki produksi daging yang tinggi, yaitu sapi Simmental, Limousin, Brahman,
dan Angus.
Simmental
Sapi Simmental berasal dari lembah Simme Swis. Sapi ini menjadi sapi yang
di Jerman diberi nama ‘Fleckvieh’. Sapi Simmental berwarna totol dan mukanya
berwarna putih. Sapi ini terkenal mampu menyusui anak dengan baik, pertumbuhan
cepat, serta badan padat dan panjang. Sapi Simmental termasuk ukuran berat, baik
pada melahirkan (Blakely dan Bade, 1998), serta memiliki karakter berat sapih dan
pertambahan berat badan pasca sapih yang tinggi (Williamson dan Payne, 1993)
Limousin
Sapi Limousin berasal dari suatu propinsi di Perancis yang banyak berbukit
batu. Warna bulunya mulai dari kuning hingga merah keemasan dengan tanduk
berwarna cerah. Keunggulan sapi Limousin adalah fertilitas cukup tinggi yang
mampu melahirkan, menyusui, dan mengasuh anak (Blakely dan Bade, 1998).
Limousin memiliki perdagingan yang bagus dengan laju pertumbuhan yang tinggi
(Phillips, 2001) yang dapat dilihat dari bobot lahirnya yang tergolong kecil hingga
medium yang berkembang menjadi golongan besar saat dewasa. Bobot badan betina
dewasa sekitar 575 kg, sedangkan pejantan sekitar 1100 kg (Blakely dan Bade,
1998).
Brahman
Sapi Brahman dikembangkan di Amerika Serikat yang merupakan hasil
perkawinan tiga bangsa sapi India, yaitu Gir, Guzerat, dan Nellore. Sapi Zebu
pertama dimasukkan ke Amerika Serikat pada tahun 1849 (Blakely dan Bade, 1998).
Sapi ini memiliki ciri khas, yaitu berpunuk di bagian punggungnya, berambut pendek
dan halus, serta sebagian besar berwarna putih (Phillips, 2001). Sapi Brahman
termasuk spesies Bos indicus yang memiliki kemampuan beradaptasi dengan
lingkungan panas, tahan terhadap penyakit caplak (Phillips, 2001), dan parasit
(Blakely dan Bade, 1998). Sapi ini sering digunakan untuk persilangan dengan sapi
lain untuk meningkatkan hybrid vigor. Berat badan betina dewasa mencapai 585 kg
dan pada pejantan mencapai 900 kg atau lebih (Blakely dan Bade, 1998).
Angus
Sapi Angus berasal dari Skotlandia Timur Laut dan diimpor ke Amerika
Serikat pada tahun 1873 dan disilangkan dengan sapi Longhorn untuk mendukung
halus, tetapi tidak bertanduk. Ukuran badan relatif kecil, pejantan memiliki berat
badan sekitar 850 kg, sedangkan betina sekitar 675 kg. Salah satu sifat yang sangat
penting adalah memiliki kualitas karkas yang istimewa dengan tulang-tulang yang
kecil, perdagingan yang baik, serta persentase lemak subkutan yang rendah (Blakely
dan Bade, 1998).
Gen Pertumbuhan
Sifat utama dari makhluk hidup diantaranya adalah tumbuh dan berkembang.
Pertumbuhan secara umum memiliki aspek yang luas, seperti pertumbuhan sel,
organ, fetus, tulang, dan beberapa aspek lain yang terkait dengan pertumbuhan
individu. Lawrence dan Fowler (2002) menyatakan bahwa pertumbuhan merupakan
suatu proses deposisi, pemindahan substansi sel-sel, serta peningkatan ukuran dan
jumlah sel pada tingkat dan titik berbeda dalam suatu waktu tertentu.
Pertumbuhan individu dipengaruhi oleh gen-gen pertumbuhan. Hormon
pertumbuhan atau growth hormone (GH) merupakan hormon anabolik yang
disintesis dan disekresikan oleh sel somatotrof pada lobus anterior pituitary (Ayuk
dan Sheppard, 2006). Gen GH memiliki fungsi penting karena mempengaruhi
pengaturan sifat-sifat yang bernilai ekonomi tinggi. Oleh sebab itu, gen GH menjadi
kandidat gen untuk program Marker Assisted Selection pada sapi (Beauchemin et al.,
2006). Gen GH merupakan salah satu gen esensial yang diperlukan untuk
pertumbuhan jaringan, metabolisme lemak, dan pertumbuhan tubuh normal (Burton
et al., 1994).
Protein GH terdiri atas 191 asam amino dengan berat molekul 22 kDa (Frago
dan Chowen 2005). Menurut Woychick et al. (1982) gen GH memiliki panjang 1800
pb dengan 5 ekson dan 4 intron. Gen GH disintesis dan disekresikan oleh sel
somatotrop anterior pada kelenjar pituitary (Ayuk and Sheppard, 2006). Gen GH
memiliki peranan terhadap pertumbuhan berbagai jaringan, seperti pada tulang, otot
dan jaringan adiposa, organ dan sistem tubuh, serta berperan dalam perkembangan
longitudinal pascanatal, laktasi, reproduksi, metabolisme protein, lemak, dan
karbohidrat (Ayuk and Sheppard, 2006; Akers et al., 2006). GH berperan dalam
pengaturan perkembangan kelenjar mamae pada ternak ruminansia (Akers et al.,
2006). Sumantran et al. (1992) telah membuktikan bahwa gen GH menjadi pengatur
pertumbuhan, serta mengatur produksi susu, karkas, dan respon imun (Ge et al.,
2000).
Gen Growth Hormone Receptor
Reseptor hormon pertumbuhan atau growth hormone receptor (GHR)
merupakan protein transmembran yang mengikat GH dengan afinitas dan spesifitas
yang tinggi (Di Stasio et al., 2005). Gen GHR merupakan gen yang menjadi faktor
penentu dari efektivitas kerja gen pertumbuhan pada ternak disebabkan gen GH
membutuhkan reseptor dalam mekanisme ekspresinya ke target jaringan. Menurut
Zhou dan Jiang (2005), aksi biologis gen GH dimediasi oleh gen GHR pada
tingkatan jaringan. Gen GHR pada sapi dipetakan sebagai gen tunggal yang terletak
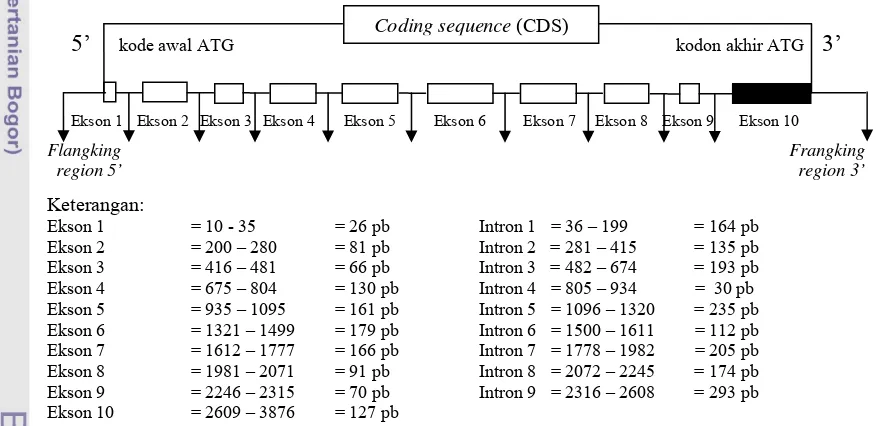
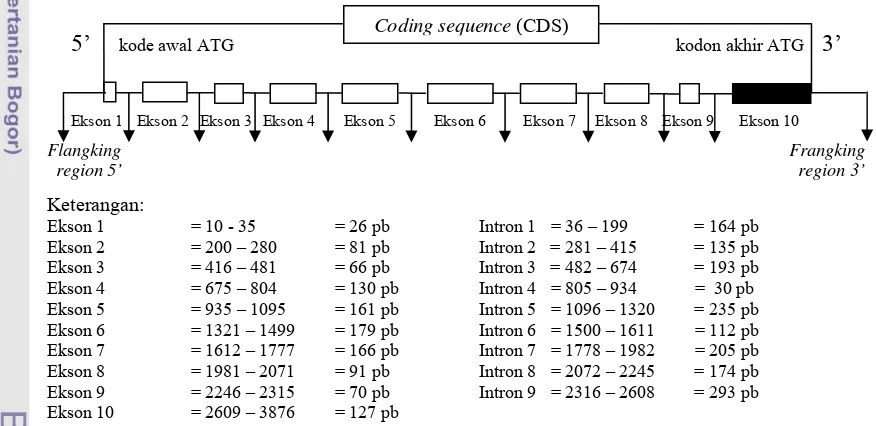
pada kromoson 20 (Moody et al., 1995), terdiri atas 10 ekson dan 9 intron, dengan
panjang 25.688 pb (Lucy et al., 1998; Jiang dan Lucy, 2001). Susunan fisik gen GHR
ditunjukkan pada Gambar 2.
5’ kode awal ATG kodon akhir ATG 3’
Ekson 1 Ekson 2 Ekson 3 Ekson 4 Ekson 5 Ekson 6 Ekson 7 Ekson 8 Ekson 9 Ekson 10
Flangking Frangking region 5’ region 3’
Keterangan:
Ekson 1 = 10 - 35 = 26 pb Intron 1 = 36 – 199 = 164 pb Ekson 2 = 200 – 280 = 81 pb Intron 2 = 281 – 415 = 135 pb Ekson 3 = 416 – 481 = 66 pb Intron 3 = 482 – 674 = 193 pb Ekson 4 = 675 – 804 = 130 pb Intron 4 = 805 – 934 = 30 pb Ekson 5 = 935 – 1095 = 161 pb Intron 5 = 1096 – 1320 = 235 pb Ekson 6 = 1321 – 1499 = 179 pb Intron 6 = 1500 – 1611 = 112 pb Ekson 7 = 1612 – 1777 = 166 pb Intron 7 = 1778 – 1982 = 205 pb Ekson 8 = 1981 – 2071 = 91 pb Intron 8 = 2072 – 2245 = 174 pb Ekson 9 = 2246 – 2315 = 70 pb Intron 9 = 2316 – 2608 = 293 pb Ekson 10 = 2609 – 3876 = 127 pb
Gambar 2. Rekonstruksi Struktur Gen GHR Berdasarkan Sekuens Gen GHR di GenBank (Kode Akses. EF207442)
Menurut Moody et al. (1995) gen GHR adalah sel permukaan reseptor untuk
GH dan dibutuhkan oleh GH untuk membawa pengaruhnya ke target jaringan. GH
memiliki berat molekul yang tinggi sehingga sulit memasuki sel yang harus melewati
membran sel. Efek pertama dari GH adalah pada reseptor yang terdapat dalam
membran sel, sehingga GH dapat memberikan efeknya pada target sel tanpa merusak
membran (Djojosoebagio, 1996).
Gen GHRsangat penting dalam proses pertumbuhan ternak. Mutasi pada gen
GHR telah diasosiasikan sebagai Larontype dwarfism pada manusia (Godowski et
al., 1989). Dwarfism dihubungkan dengan rendahnya peredaran konsentrasi gen
IGF-I dan dengan ketiadaan atau rendahnya ekspresi gen GHR (Vandeerpooten et
al., 1991). Gen GH, insulin-like growth factors 1 dan 2 (IGF1 dan IGF2), dan
hubungan keduanya pada pengikatan protein dan reseptor transmembran (GHR,
IGF1R dan IGF2R) memberi pengatur penting pada fisiologis pertumbuhan mamalia
(Curi et al., 2006).
Gen GHR pada sapi pedaging diketahui bertanggung jawab pada sifat
pertumbuhan (Hale et al., 2000), lemak karkas (Tatsuda et al., 2008), lemak intra
muscular (Han et al., 2009), serta komposisi otot (lemak intra muskular, protein, dan
kadar air) (Reardon et al., 2010). Gen GHR pada sapi perah diketahui dapat
mempengaruhi sifat produksi susu (Aggrey et al., 1999). Keragaman gen GHR
berhubungan dengan keragaman produksi daging yang terjadi pada liver-specific
promoter sapi (Ohkubo et al.,2006). Gen GHR memiliki ekspresi pada tubuh dengan
level yang tinggi pada hati (Lucy et al., 1998).
Aggrey et al. (1999) meneliti gen GHR|AluI menggunakan metode
PCR-RFLP dengan suhu annealing 66 oC selama 80 detik pada bull Holstein. Runutan
primer yang digunakan, yaitu primer forward 5’-TGCGTGCACAGCA
GCTCAACC-3’ dan primer reverse ‘5-AGCAACCCCACTGCTGGGCAT-3’.
Panjang fragmen hasil amplifikasi gen yang diperoleh sepanjang 836 pb (1036-1871,
ekson 5-intron 7). Hasil pemotongan gen GHR dengan enzim restriksi AluI
menghasilkan dua alel yang diberi nama AluI(+) dan AluI(-), serta tiga genotipe
AluI(+/+), AluI(+/-), dan Alu(-/-). Genotipe AluI(+/+) memiliki panjang fragmen 747
pb (terbagi menjadi dua fragmen yaitu 602 pb dan 145 pb), AluI(+/-) (747 pb, 602
pb, 145pb,75 pb, dan 14 pb), AluI(-/-) (747 pb, 75 pb, dan 14 pb (tidak terdeteksi
pada gel). Titik mutasi yang ditemukan pada runutan basa nukleotida 1182 pb
dimana A (Adenin) menjadi T (Timin). Mutasi yang terjadi merupakan mutasi
transversi dimana basa purin menjadi pirimidin. Frekuensi alel AluI(-) pada bull
Holstein tahun 1950-1970 sebesar 0,63 dan frekuensi alel AluI(+) sebesar 0,37.
Frekuensi alel AluI(+) mengalami peningkatan menjadi 0,58 dan penurunan alel
Zulkarnaim et al. (2010) telah melaporkan amplifikasi gen GHR|AluI dengan
metode PCR-RFLP pada sapi Bali, Limousin, Simmental, dan sapi Pesisir
menggunakan runutan primer forward 5’-CGCTTACTTCTGCGAGGTAGACGC-3’
dan primer reverse 5’-GTCTGTGCTCACATAGCCAC-3’ (Andreas, 2010). Hasil
amplifikasi pada ekson 10 menghasilkan fragmen sepanjang 298 pb. Pemotongan
fragmen gen GHR dengan menggunakan enzim restriksi AluI menghasilkan dua alel
(A dan G) serta tiga genotipe, yaitu genotipe AA, GG, dan AG. Pola genotipe AA
dengan tiga pita (167 pb, 81 pb, dan 50 pb), GG memiliki dua pita (131 pb dan 167
pb), dan AG merupakan genotipe gabungan (167 pb, 131 pb, 81 pb, dan 50 pb).
Mutasi gen GHR|AluI mengubah basa Adenin (A) menjadi Guanin (G) pada posisi
81 pb yang menyebabkan perubahan asam amino Serin (AGC) menjadi Glisin
(GGC) pada runutan nukleotida 3338 pb (Gen Bank, Kode Akses. EF207442).
Perhitungan frekuensi genotipe AA, AG, dan GG yang dilakukan Zulkarnaim
et al. (2010) pada sapi Bali berturut-turut (0,988; 0,006; dan 0,006) dengan frekuensi
alel A (0,991) dan G (0,009). Frekuensi genotipe AA, AG, dan GG pada sapi
Limousin (0,238; 0,095; dan 0,667) dengan frekuensi alel A (0,286) dan G (0,714).
Frekuensi genotipe AA, AG, dan GG pada sapi Simmental (0,000; 0,529; dan 0,471)
dengan frekuensi alel A (0,265) dan G (0,735). Frekuensi genotipe AA, AG, dan GG
pada sapi Pesisir (0,604; 0,021; dan 0,375) dengan frekuensi alel A (0,615) dan G
(0,385). Keseimbangan Hardy-Weinberg hanya terdapat pada sapi Simmental,
sedangkan pada sapi Bali, Limousin, dan Pesisir tidak berada dalam keseimbangan
Hardy-Weinberg. Tingkat heterozigositas tertinggi terdapat pada sapi Simmental (Ho
= 0,529) (He = 0,389±0,021) dan terendah pada sapi Bali (Ho = 0,006) (He = 0,018±0,001).
Di Stasio et al. (2005) meneliti keragaman gen GHR pada sapi Piedmontese
menggunakan metode PCR-RFLP. Penelitian dilakukan pada dua grup sampel, yaitu
213 ekor pejantan muda (grup: PTEST) dan 21 ekor sapi RPH (grup: MEAT).
Panjang fragmen hasil amplifikasi GHR pada ekson 10 sepanjang 342 pb. Hasil
pemotongan gen GHR dengan enzim restriksi AluI menghasilkan dua alel, yaitu alel
GHRA (191 pb, 101 pb, dan 50 pb) dan GHRG (191 pb dan 151 pb). Titik mutasi gen
GHR|AluI ditemukan pada posisi 257 pb yang mengubah basa Adenin (A) menjadi
alel GHRA (0,49) dan GHRG (0,51), dengan frekuensi genotipe AA (0,24), AG
(0,50), dan GG (0,26). Pada grup MEAT frekuensi alel GHRA (0,42) dan GHRG
(0,58) dengan frekuensi genotipe AA (0,24), AG (0,37), dan GG (0,39).
Penelitian lain dilakukan oleh Ge et al. (2000) menggunakan metode
PCR-RFLP pada ekson 10. Amplifikasi gen GHR menghasilkan fragmen sepanjang 286
pb. Pemotongan gen GHR dengan enzim restriksi AluI menunjukkan adanya titik
mutasi yang dielektoforesis dengan denaturing gradient gel electrophoresis (DGGE)
pada empat nukleotida tunggal atau Single Nucleotide Polymorphism (SNP) (Gen
Bank, kode akses: AF140284). Hasil pemotongan gen GHR|AluI menghasilkan alel
A dan G, serta tiga genotipe, yaitu genotipe AA, GG, dan AG. Genotipe AA
memiliki tiga pola pita (155 bp, 81 bp, dan 50 bp), GG (131 bp dan 155 bp), dan
genotipe AG (155 bp, 131 bp, 81 bp, dan 50 bp). Titik mutasi gen GHR yang
ditemukan terletak pada posisi 76 (T/C), 200 (G/A), 229 (T/C), dan 257 (A/G). Pada
posisi 200 dan 257 terjadi substitusi asam amino induksi, Ala/Thr dan Ser/Gly, pada
posisi lainnya terjadi silent mutation.
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP)
Polymerase chain reaction (PCR) adalah suatu reaksi in vitro untuk
menggandakan molekul DNA pada target tertentu dengan cara mensintesis molekul
DNA baru yang berkomplemen dengan molekul DNA tersebut dengan enzim
polymerase dan oligonukleotida pendek sebagai primer dalam mesin thermocycler.
Metode ini berjalan secara enzimatik melalui mekanisme perubahan suhu. Proses
yang terjadi dalam mesin PCR meliputi tiga tahap utama, yaitu denaturasi
(pemisahan untai ganda DNA), annealing (penempelan primer), dan ekstensi
(pemanjangan primer). Proses dari mulai denaturasi, penempelan, dan ekstensi
disebut sebagai satu siklus (Muladno, 2002).
Polimerase chain reaction–restriction fragment length polymorphism
(PCR-RFLP) merupakan metode analisis lanjutan dari produk PCR. Metode PCR
memanfaatkan perbedaan pola pemotongan enzim restriksi atau enzim pemotong
yang berbeda pada tiap-tiap mikroorganisme (Orita et al., 1989). Enzim restriksi
akan memotong pada sekuens nukleotida yang spesifik atau recognition sequences
menyatakan bahwa PCR-RFLP merupakan suatu metode sederhana yang biasa
digunakan untuk mengetahui keragaman genotipe. Prinsip kerja RFLP adalah
menghilangkan semua mutasi atau menciptakan sekuen rekognisi baru. Penyisipan
(insersi), penghilangan (delesi), maupun subtitusi nukleotida yang terjadi pada
daerah rekognisi menyebabkan tidak dikenalinya situs pemotongan oleh enzim
restriksi dan menyebabkan perbedaan pola pemotongan DNA (Lewin, 1994).
Metode PCR-RFLP telah digunakan untuk mendapatkan variasi pada setiap
lokasi DNA spesifik, baik pada daerah yang bersifat penyandi atau coding region
pada genom maupun pada daerah yang tidak bersifat berpenyandi atau daerah
non-coding region (Vasconcellos et al., 2003). Analisis RFLP sering digunakan untuk
mendeteksi lokasi genetik dalam kromosom yang menyandikan penyakit yang
diturunkan (Orita et al., 1989) ataupun untuk mendeteksi adanya keragaman pada
gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu
(Sumantri et al., 2007). Nei dan Kumar (2000) menyatakan bahwa atas dasar
terpotong atau tidaknya fragmen DNA dengan enzim pemotong, hasil fragmen
potongan DNA tersebut dapat divisualisasi melalui teknik elektroforesis yang
hasilnya menunjukkan ada tidaknya polimorfisme pada suatu individu dalam
populasi.
Keragaman Genetik
Identifikasi keragaman genetik dalam suatu populasi digunakan untuk
mengetahui dan melestarikan bangsa-bangsa dalam populasi terkait dengan penciri
suatu sifat khusus. Populasi alami biasanya memiliki keragaman genetik yang tinggi.
Informasi keragaman genetik suatu bangsa akan sangat bermanfaat bagi keamanan
dan ketersediaan bahan pangan yang berkesinambungan (Blott et al., 2003).
Keragaman genetik dapat digunakan sebagai parameter dalam mempelajari genetika
populasi dan genetika evolusi (Nei dan Kumar, 2000).
Menurut Nei dan Kumar (2000), tingkat keragaman dalam populasi dapat
digambarkan dari frekuensi alel. Frekuensi alel merupakan rasio relatif suatu alel
terhadap keseluruhan alel yang ditemukan dalam satu populasi. Suatu populasi
dinilai beragam jika memiliki dua atau lebih alel dalam satu lokus dengan frekuensi
yang cukup (biasanya lebih dari 1%). Suatu alel dikatakan polimorfik jika memiliki
Hartl dan Clark (1997) menyatakan bahwa polimorfisme genetik dalam suatu
populasi dapat digunakan dalam menentukan hubungan antar subpopulasi yang
terfragmentasi dalam suatu spesies. Estimasi perhitungan keragaman genetik dalam
populasi secara kuantitatif dapat diperoleh melalui dua ukuran keragaman variasi
populasi, yaitu proporsi lokus polimorfisme dalam populasi dan rata-rata proporsi
individu heterozigot dalam setiap lokus (Nei dan Kumar, 2000). Keragaman genetik
antara subpopulasi dapat diketahui dengan melihat persamaan dan perbedaan
frekuensi alel di antara subpopulasi (Li et al., 2000).
Noor (2008) menjelaskan bahwa dalam Hukum Hardy-Weinberg dinyatakan
frekuensi genotipe suatu populasi yang cukup besar akan selalu dalam keadaan
seimbang bila tidak ada seleksi, migrasi, mutasi, dan genetic drift. Silang dalam dan
silang luar dalam kelompok/populasi yang sama (endogami) juga dapat
mempengaruhi frekuensi genotipe (Vasconcellos et al., 2003). Menurut Falconer dan
Mackay (1996), ukuran populasi juga mempengaruhi perubahan frekuensi genotipe
dari generasi ke generasi. Informasi keragaman genetik suatu populasi menggunakan
beberapa lokus dapat digambarkan melalui nilai heterozigositas.
Derajat heterozigositas, menurut Nei (1987), merupakan rataan persentase
individu heterozigot dalam suatu populasi. Javanmard et al. (2005) menyatakan
bahwa nilai heterozigositas di bawah 0,5 (50%) mengindikasikan rendahnya variasi
suatu gen dalam populasi dan jika nilai Ho lebih rendah dari He maka dapat
mengindikasikan adanya proses seleksi yang intensif (Tambasco et al., 2003). Avise
(1994) juga menyatakan bahwa semakin tinggi derajat heterozigositas suatu populasi
maka daya tahan hidup populasi tersebut akan semakin tinggi. Seiring dengan
menurunnya derajat heterozigositas akibat dari silang dalam dan fragmentasi
populasi, sebagian besar alel resesif yang bersifat lethal semakin meningkat
MATERI DAN METODE Lokasi dan Waktu
Analisis Polymerase Chain Reaction (PCR) serta analisis penciri Polymerase
Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP)
dilaksanakan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan
Genetika Ternak, Fakultas Peternakan, Institut Pertanian Bogor. Analisis DNA
berlangsung pada bulan Juli sampai November 2010.
Materi Sampel Darah
Sampel darah sapi Friesian Holstein berasal dari BIB Lembang, BBIB
Singosari, dan BET Cipelang serta sebagai pembanding digunakan sapi Simmental,
Limousin, Brahman, dan Angus yang berasal dari BET Cipelang. Sampel DNA yang
digunakan merupakan koleksi sampel Laboratorium Genetika Molekuler Ternak,
bagian Pemuliaan dan Genetika Ternak, Fakultas Peternakan, Institut Pertanian
Bogor. Jumlah tiap sampel DNA dan asal pengambilan sampel darah disajikan pada
Tabel 1.
Tabel 1. Jumlah DNA dan Asal Pengambilan Sampel Darah yang Digunakan dalam Penelitian.
No. Bangsa Sapi Jenis
Kelamin
Tipe Sapi Jumlah Sampel (ekor)
Lokasi
1. Friesian Holstein Jantan Perah 17 BIB Lembang
2. Friesian Holstein Jantan Perah 32 BBIB Singosari
3. Friesian Holstein Betina Perah 40 BET Cipelang
Subtotal 89
4. Simmental Betina Pedaging 13 BET Cipelang
5. Limousin Betina Pedaging 14 BET Cipelang
6. Brahman Betina Pedaging 5 BET Cipelang
7. Angus Betina Pedaging 5 BET Cipelang
Ekstraksi DNA
Ekstraksi DNA telah dilakukan mengikuti metode phenol-cloroform
(Sambrook et al., 1989). Bahan-bahan yang digunakan, yaitu 200 µl sampel darah,
1000 µl Destilata Water (DW), 40 µl Sodium Dodecyl Sulphate 10% (SDS), 10 µl
Proteinase K 5 mg/ml, 1x Sodium Tris EDTA (STE), 400 µl larutan fenol, 400 µl
Chloroform Isoamil Alcohol (CIAA), 80 µl NaCl 5 M, 800 µl etanol absolut, etanol
70%, 100 µl TE 80% atau Elution buffer, dan sarung tangan. Alat-alat yang
digunakan, yaitu tabung vaccutainer, rak tabung vaccutainer, rak penyimpan sampel,
rak tabung Eppendorf, tabung Eppendorf 1,5 ml, mikro pipet (200-1000 µl, 20-100
µl, 2-20 µl) beserta tipsnya (warna biru dan kuning), alat mikrosentrifugasi
berpendingin, vortex mixer, autoclave, incubator, freezer, rotary mixer, gunting,
pinset steril, dan alat tulis.
Primer
Primer gen GHR|AluI yang digunakan dalam penelitian ini mengikuti Ge et
al. (2000) yang dimodifikasi oleh Andreas (2010) dengan runutan primer forward
5’-CGCTTACTTCTGCGAGGTAGACGC-3’ dan primer reverse
5’-GTCTGTGCTCACATAGCCAC-3’.
Amplifikasi DNA
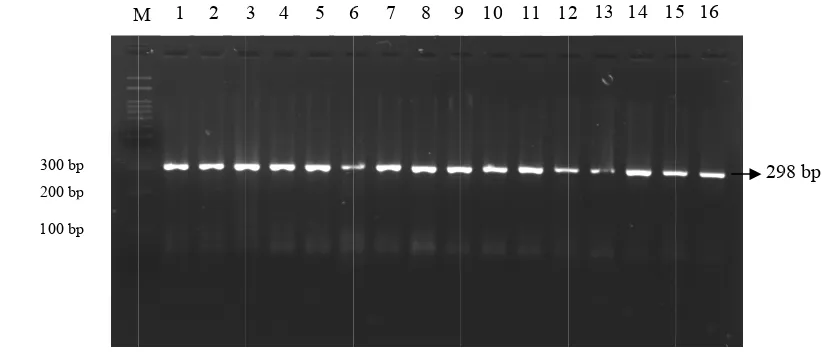
Fragmen gen GHR memiliki panjang produk PCR 298 pb pada ekson 10
bovine Growth Hormon Receptor (Gambar 4). Bahan-bahan yang digunakan
meliputi 1 µl sampel DNA; 9,7 µl destilate water; 1,25 µl buffer; 0,25 µl MgCl2;
0,15 µl dNTPs; 0,1 µl primer; dan 0,05 µl Taq. Alat-alat yang digunakan untuk
amplifikasi DNA antara lain microtube, mikropipet dengan tipsnya, dan mesin
thermal cycler.
Analisis PCR-RFLP
Bahan-bahan yang digunakan dalam analisis PCR-RFLP adalah 5 µl produk
PCR fragmen gen GHR, 1 µl destilate water; 0,7 µl buffer (buffer tango); dan 0,3µl
enzim pemotong AluI. Alat-alat yang digunakan dalam analisis PCR-RFLP antara
Elektroforesis
Bahan-bahan yang digunakan untuk elektroforesis adalah loading dye,
marker 100 pb, agarose, 0,5x TBE (1 M Tris, 0,9 M Asam Borat, 0,01 M EDTA pH
8), produk PCR, dan etidium bromide. Alat-alat yang digunakan untuk elektroforesis
antara lain gelas ukur, gelas kimia, stirrer, magnet stirrer, mikro pipet 10µl dengan
tipsnya, power supply electroforesis 500vA, dan alat foto UVtransiluminator.
Prosedur Penarikan Sampel Data Penelitian
Sampel data penelitian ditarik berdasarkan kriteria bangsa, yaitu bangsa FH
yang diambil dari BIB Lembang, BBIB Singosari, dan BET Cipelang, serta sapi
Simmental, Limousin, Brahman, dan Angus diambil dari BET Cipelang .
Pengambilan dan Penanganan Sampel
Darah diambil melalui vena jugularis menggunakan jarum vennoject dan
tabung vaccutainer tanpa heparin. Ethanol absolute ditambahkan pada sampel darah
dengan perbandingan 1:2 dan disimpan pada suhu ruang.
Ekstraksi DNA
Ektraksi DNA mengikuti metode phenol-chloroform (Sambrook et al., 1989):
Preparasi Sampel. Sampel darah 200 µl dimasukkan ke tabung 1,5 ml. Ethanol
dihilangkan dengan menambahkan air destilasi 1000 µl. Sampel disentrifugasi 8000
rpm selama 5 menit, dan supernatan dibuang.
Degradasi protein. Sebanyak 1x STE sampai volume 400 µl, 40 µl SDS 10%, dan 10 µl proteinase K 5 mg/ml ditambahkan ke dalam sampel. Campuran diinkubasi dan
digoyang pelan pada suhu 55 oC selama 2 jam.
Degradasi Bahan Organik. Sebanyak 400 µl larutan phenol, 400 µl
chloroform:isoamyl alcohol (24:1), dan 40 µl NaCl 5M ditambahkan ke dalam
sampel. Campuran digoyang pelan pada suhu ruang selama satu jam.
dengan menambahkan 800 µl ethanol absolute dan 40 µl NaCl 5 M. Campuran
kemudian diinkubasi pada suhu -20 oC selama satu malam. Pengendapan DNA
dilakukan dengan sentrifugasi 12.000 rpm selama 5 menit. Endapan DNA dicuci
dengan 800 µl alkohol 70%, kemudian diendapkan lagi dan dikeringkan. Sebanyak
100 µl TE 80% (Tris EDTA) ditambahkan pada DNA. Sampel DNA disimpan pada
suhu -20 oC dan siap untuk digunakan. Amplifikasi DNA menggunakan mesin
thermal cycler.
Amplifikasi DNA
Proses amplifikasi DNA secara umum menggunakan metode sebagai berikut.
Satu µl sampel DNA dimasukkan ke dalam tabung PCR, kemudian ditambahkan 0,1
µl primer. Larutan dalam tabung kemudian ditambahkan dengan premix yang terdiri
dari 0,15 µl dNTPs; 0,25 µl MgCl2; 0,05 µl Taq Polymerase (real tag); 1,25 µl
buffer; dan 9,7 µl DW dalam larutan total 12 µl. Tabung kemudian diinkubasi pada
mesin thermal cycler dengan suhu denaturasi awal 94 o C selama 5 menit, denaturasi
pada suhu 94 oC selama 45 detik, penempelan primer pada suhu 62 oC selama 45
detik, ekstensi (perpanjangan DNA baru) pada suhu 72 oC selama 1 menit, dan
ekstensi (perpanjangan) akhir pada suhu 72 oC selama 5 menit.
Analisis PCR-RFLP
Produk PCR yang diperoleh dari hasil amplifikasi fragmen gen GHR|AluI
dipotong menggunakan enzim pemotong AluI (AG|CT). Volume dan bahan pereaksi
yang digunakan untuk setiap enzim pemotong adalah 5 µl sampel produk PCR; 1 µl
DW; 0,7 µl 1x buffer enzim pemotong (buffer tango); dan 0,3 µl enzim pemotong
AluI. Campuran sampel produk PCR fragmen gen GHR|Alu1 dengan bahan pereaksi
diinkubasi di dalam inkubator pada suhu 37 oC selama ± 16 jam.
Elektroforesis DNA Total dan Produk PCR -RFLP
Potongan DNA produk PCR dipisahkan dengan teknik elektroforesis
terhadap DNA total, produk PCR, dan produk pemotong (digested) atau PCR-RFLP
dengan menggunakan gel agarose dengan konsentrasi yang berbeda. Konsentrasi
agarose 1,5% digunakan untuk elektroforesis DNA total (0,45 g/30 ml dan 0,5 x
sesuai dengan konsentrasi yang dibutuhkan, kemudian dipanaskan dalam microwave
sampai mendidih (larutan terlihat bening), larutan didinginkan dengan stirrer di atas
magnet stirrer, dan ditambahkan 5 µl ethidium bromide (10 mg/ml), kemudian
diletakkan di cetakan wel dan dipasang sisir untuk 16 sampel dan dibiarkan sampai
membeku.
Sebelum menjalankan piranti elektroforesis, sampel dimasukkan ke setiap
sumur sampel, larutan atau buffer 0,5x TBE dimasukkan sampai sampel terendam,
dan sampel siap dijalankan. Elektroforesis menggunakan Mupid Electroforesis yang
dijalankan atau dimigrasikan dari kutub negatif (katoda) ke positif (anoda) dengan
arus 100 volt selama 30 menit sampai 45 menit. Hasil elektroforesis diamati dengan
bantuan sinar UVtransilluminator dan hasilnya difoto. Keberadaan pita (band) yang
tergambar dalam hasil elektroforesis memberikan hasil ada tidaknya DNA total,
berhasil tidaknya amplifikasi PCR atau ada tidaknya variasi atau keragaman fragmen
gen GHR|Alu1.
Analisis Data Frekuensi Genotipe dan Alel
Keragaman genotipe pada setiap individu dapat ditentukan melalui pita-pita
DNA yang ditemukan. Frekuensi genotipe merupakan rasio dari jumlah genotipe dari
suatu populasi, sedangkan frekuensi alel merupakan rasio jumlah alel pada suatu
lokus di populasi tertentu. Frekuensi genotipe dan alel gen GHR|AluI dapat dihitung
dengan menggunakan rumus Nei dan Kumar (2000):
χ
ii=
χ
i=
∑
Keterangan: χii =Frekuensi genotipe χi = Frekuensi alel ke-i
nii = Jumlah sampel bergenotipe ii
nij = Jumlah sampel bergenotipe ij
N = Jumlah populasi
Keseimbangan Hardy-Weinberg (HW)
Keseimbangan Hardy-Weinberg diuji dengan perhitungan Chi-Kuadrat (Nei,
1987).
X2
=
∑ EKeterangan: X2 = Nilai uji Chi-kuadrat
O = Jumlah pengamatan genotipe ke-i E = Jumlah harapan genotipe ke-i
Heterozigositas
Derajat heterozigositas alel (Ho) dari suatu populasi dapat digunakan untuk
mengestimasi keragaman genetik dengan dihitung menggunakan rumus Weir (1996)
sebagai berikut:
Ho =
∑
Heterozigositas harapan (He) pada suatu populasi dapat dihitung dengan
menggunakan rumus Nei dan Kumar (2000) sebagai berikut:
He = 1 –
∑
Keterangan : Ho = Nilai heterozigositas pengamatan He = Nilai heterozigositas harapan Xi2 = Frekuensi homozigot
kadar air) (Reardon et al., 2010), dan sifat produksi susu pada sapi Holstein (Aggrey
et al., 1999).
Penentuan genotipe gen GHR pada sapi Fresian Holstein dari BIB Lembang,
BBIB Singosari dan BET Cipelang dilakukan dengan pendekatan PCR-RFLP dengan
menggunakan enzim restriksi AluI. Enzim AluI mengenali situs pemotongan AG|CT.
Berdasarkan sekuen DNA ruas gen GHR yang diamplifikasi terdapat dua situs
pemotong AluI pada posisi 81 pb dan 131 pb, ekson 10 bovine Growth Hormone
Receptor (Gen Bank Kode Akses EF207442) yang menghasilkan fragmen dengan
panjang 50 pb, 81 pb, dan 167 pb yang dikenal dengan alel A (Gambar 4).
Gambar 4. Fragmen Gen GHR|AluI Didasarkan pada Sekuens Gen GHR di
Gen Bank (Kode Akses. EF207442).
Keterangan: Posisi primer (bergaris bawah), situs pemotongan enzim AluI (cetak tebal bergaris bawah berwarna abu-abu) dan titik mutasi (cetak tebal, tulisan miring, bergaris bawah).
Titik mutasi ditemukan pada situs AluI basa A (adenin) menjadi G (guanin)
pada posisi 81 pb (Gambar 4) gen GHR|AluI hasil amplifikasi atau pada posisi basa
nukleotida 3338 pb sesuai sekuen GHR di GenBank (kode Akses EF207442). Titik
mutasi pada posisi 81 pb juga ditemukan pada penelitian yang dilakukan oleh
Zulkarnaim et al. (2010). Mutasi yang menyebabkan perubahan asam amino, yaitu
dari serin menjadi glisin (AGC – serin; GGC - glisin) ini ditemukan pula oleh Ge et al. (2000); Di Stasio et al. (2005); Tatsuda et al. (2008); Han et al.(2009); dan
Reardon et al. (2010). Perubahan tersebut menyebabkan situs pemotong tidak
dikenali oleh enzim AluI yang dikenal dengan alel G (Ge et al., 2000; Di Stasio et
al., 2005). Titik mutasi yang ditemukan pada bull Holstein berbeda yaitu, perubahan
asam amino yang terjadi, yaitu dari A (adenin) menjadi T (timin) (Aggrey et al., Forward
3241 taacttcatc gtggacaacg cttacttctg cgaggtagac gccaaaaagt acattgccct
3301 ggcccctcac gtcgaggctg aatcacacgt agagccaag|c tttaaccagg aagacattta
3361 catcaccaca gaaagcctta ccactacag|c tgggaggtcg gggacagcag aacatgttcc
3421 aagttctgag atacctgtcc cagattatac ctccattcat atagtacagt ctccacaggg 3481 cctcgtactc aatgcgactg ccctgccctt gcctgacaaa gagtttctct catcatgtgg 3541 ctatgtgagc acagaccaac tgaacaaaat catgccatag cttttctttg atttcccatg
Reverse
Alel A : ‘5 – agagccaAG|CTttaaccagg – 3’
Tingkat keragaman genotipe yang muncul menunjukkan adanya pengaruh
gen GHR|AluI terhadap sifat-sifat unggul yang diseleksi untuk mendapatkan bibit
yang berkualitas (Gambar 6). Sapi Friesian Holstein menunjukkan bahwa genotipe
AA merupakan genotipe yang paling banyak di ketiga lokasi, yaitu BIB Lembang
(13/17), BBIB Singosari (24/32), dan BET Cipelang (27/40). Genotipe AA dan AG
memiliki jumlah yang sama pada sapi FH di BIB Lembang (2 ekor) dan BBIB
Singosari (4 ekor), sedangkan di BET Cipelang, sapi FH genotipe AG lebih banyak
dibandingkan genotipe GG (8 dan 5 ekor). Genotipe AA muncul sebagai genotipe
terbanyak terjadi pula pada sapi pedaging Limousin dan Brahman, bahkan pada sapi
Brahman semua sampel bergenotipe AA (100%). Berbeda halnya pada sapi
Simmental dan Angus, genotipe yang paling banyak muncul adalah genotipe AG (6
dan 3) dan genotipe AA merupakan genotipe yang paling sedikit muncul (2 tiap
sampel).
Frekuensi Genotipe dan Alel Gen GHR|AluI
Frekuensi genotipe merupakan rasio dari jumlah genotipe yang muncul pada
suatu populasi. Sementara frekuensi alel merupakan rasio suatu alel terhadap
keseluruhan alel pada suatu lokus dalam populasi. Frekuensi genotipe dan alel
merupakan parameter dasar untuk mempelajari proses terjadinya evolusi karena
perubahan genetik pada sebuah populasi biasanya digambarkan dengan adanya
perubahan pada frekuensi alel (Nei dan Kumar, 2000). Hasil analisis frekuensi
genotipe dan alel gen GHR pada setiap populasi sapi FH dan sapi potong disajikan
pada Tabel 2.
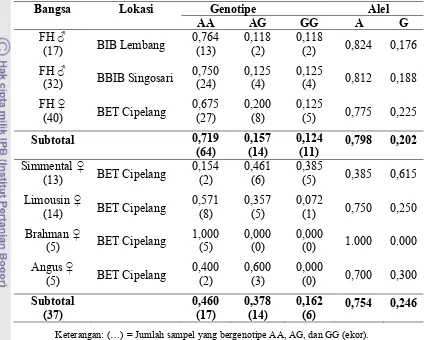
Sapi FH pengamatan di BIB Lembang, BBIB Singosari, dan BET Cipelang
memiliki nilai frekuensi genotipe AA paling tinggi (0,764; 0,750; dan 0,675).
Genotipe AG dan GG memiliki frekuensi yang seimbang pada sapi FH di BIB
Lembang (0,118) dan BBIB Singosari (0,125). Sapi FH yang berasal dari BET
Cipelang memiliki nilai frekuensi genotipe AG lebih tinggi dari GG (0,200 dan
0,125). Frekuensi tertinggi genotipe AA pada penelitian ini berkebalikan dengan
yang telah ditemukan Aggrey et al. (1999) pada bull Holstein, genotipe tertinggi
yang ditemukan adalah genotipe GG namun dengan nama AluI(+/+). Hal ini
disebabkan perbedaan lokasi yang diamati, walaupun menggunakan metode yang
keragaman gen GHR|AluI pada ekson 10 sedangkan pada penelitian yang dilakukan
oleh Agrrey et al. (1999) pada ekson 5 sampai intron 7.
Tabel 2. Frekuensi Genotipe dan Alel
Bangsa Lokasi Genotipe Alel
AA AG GG A G
(32) BBIB Singosari
0,750
Keterangan: (…) = Jumlah sampel yang bergenotipe AA, AG, dan GG (ekor). ♂ = Jantan
♀ = Betina
Frekuensi alel sapi FH dari ketiga lokasi menunjukkan frekuensi alel A
(0,775-0,824) lebih banyak muncul dibandingkan alel G (0,176-0,225). Hal ini
berkebalikan dengan penelitian yang dilakukan oleh Aggrey et al. (1999) pada sapi
pejantan Holstein yang menunjukkan bahwa alel AluI(+) (menyerupai pola pita alel
G) meningkat dan alel AluI(-) (menyerupai pola pita alel A) menurun. Frekuensi alel
AluI(+) pada sapi pejantan Holstein di tahun 1950-1970 meningkat hingga tahun
1980an (0,37 menjadi 0,58).
Sapi pedaging yang berasal dari BET Cipelang memiliki variasi genotipe
tertinggi. Genotipe AA juga merupakan frekuensi tertinggi pada sapi Limousin
AA tertinggi ditemukan juga pada sapi Brahman, bahkan hanya terdapat genotipe
AA (1,000). Hal ini berkebalikan dengan penelitian yang dilakukan pada sapi
Piedmontese (Bos taurus) yang menunjukkan genotipe AA merupakan genotipe
terendah (0,240) (Di Stasio et al., 2005). Hal ini dapat disebabkan oleh perbedaan
jumlah sampel yang digunakan dan lokasi pengamatan.
Sapi Simmental menunjukkan hasil yang sama, yaitu dengan tiga genotipe
(AA, AG, dan GG). Berbeda dengan sapi Angus yang hanya muncul dua genotipe,
yaitu AA dan AG. Genotipe AG merupakan genotipe tertinggi pada sapi Simmental
dan Angus (0,462 dan 0,600), sedangkan genotipe AA merupakan genotipe terendah
(0,154 dan 0,400). Genotipe AG tertinggi menyerupai penelitian pada sapi
Simmental (0,529) (Zulkarnaim et al., 2010) dan sapi Angus (0,504-AB) (Ge et al.,
2003). Dominasi genotipe yang muncul mengindikasikan adanya seleksi pada setiap
populasi untuk mendapatkan sifat-sifat unggul yang diharapkan.
Frekuensi alel A yang tinggi ditemukan pula pada sapi pedaging bangsa
Limousin, Angus, dan Brahman, bahkan pada sapi Brahman hanya terdapat alel A
(0,750; 0,700; dan 1,000). Frekuensi alel A tertinggi pada sapi Angus ditemukan juga
oleh Ge et al. (2000) dengan nilai 0,78. Sapi Simmental sebaliknya memiliki
frekuensi alel terbanyak yaitu alel G (0,615). Hal ini ditemukan juga pada sapi
Piedmontese (Bos taurus) berkisar antara 0,51-0,58 (Di Stasio et al., 2005) dan pada
sapi Simmental dengan frekuensi alel G tertinggi (0,735) yang diidentifikasi oleh
Zulkarnaim et al. (2010).
Sapi FH pengamatan dari ketiga lokasi berifat polimorfik dengan dua alel
yaitu A dan G, serupa dengan sapi pedaging dari BET Cipelang, kecuali pada sapi
Brahman yang hanya memiliki satu alel. Suatu alel dikatakan polimorfik jika
memiliki frekuensi alel sama dengan atau kurang dari 0,99 (Nei, 1987). Sehingga
dapat dikatakan bahwa sapi Brahman bersifat monomorfik karena frekuensi alel
bernilai 1,000.
Keseimbangan Hardy-Weinberg
Hukum Hardy-Weinberg menggambarkan keseimbangan suatu lokus dalam
populasi diploid yang mengalami perkawinan secara acak dan bebas dari faktor yang
pergeseran genetik (Gillespie, 1998). Keseimbangan Hardy-Weinberg yang diuji
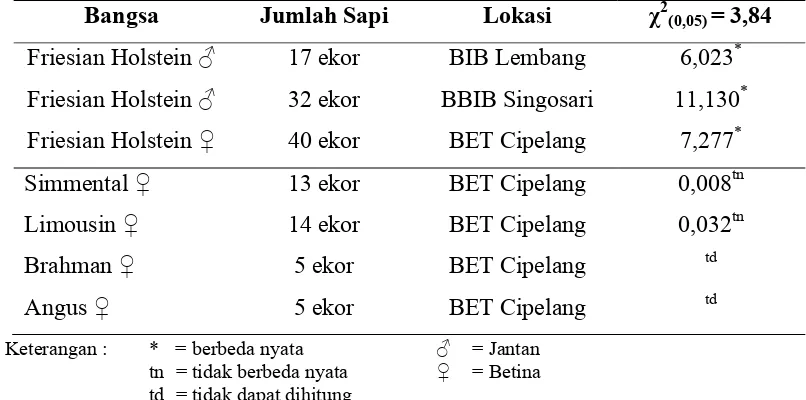
dengan Chi-Kuadrat (χ2) pada setiap populasi disajikan pada Tabel 3.
Tabel 3. Keseimbangan Hardy-Weinberg
Bangsa Jumlah Sapi Lokasi χ2(0,05) = 3,84
Friesian Holstein ♂ 17 ekor BIB Lembang 6,023*
Friesian Holstein ♂ 32 ekor BBIB Singosari 11,130*
Friesian Holstein ♀ 40 ekor BET Cipelang 7,277*
Simmental ♀ 13 ekor BET Cipelang 0,008tn
Limousin ♀ 14 ekor BET Cipelang 0,032tn
Brahman ♀ 5 ekor BET Cipelang td
Angus ♀ 5 ekor BET Cipelang td
Keterangan : * = berbeda nyata ♂ = Jantan tn = tidak berbeda nyata ♀ = Betina
td = tidak dapat dihitung
Hasil analisis Chi-Kuadrat menunjukkan bahwa sapi Friesian Holstein di BIB
Lembang, BBIB Singosari, dan BET Cipelang tidak berada dalam keseimbangan
Hardy-Weinberg (χ2>χ20,05). Suatu populasi dinyatakan dalam keadaan keseimbangan
Hardy-Weinberg, jika frekuensi genotipe (p2, 2pq dan q2) dan frekuensi alel (p dan q)
konstan dari generasi karena akibat penggabungan gamet yang terjadi secara acak ke
dalam populasi yang besar (Vasconcellos et al. 2003). Ketidakseimbangan dapat
disebabkan oleh seleksi, inbreeding, migrasi dan genetic drift (Noor, 2008). Pada
penelitian ini, hal yang paling mempengaruhi suatu populasi tidak berada dalam
keseimbangan Hardy-Weinberg adalah seleksi karena setiap populasi merupakan
hasil dari seleksi untuk mendapatkan bibit unggul.
Perhitungan Chi-Kuadrat pada sapi pedaging berkebalikan dengan sapi FH.
Sapi Simmental dan Limousin berada dalam keseimbangan Hardy-Weinberg.
Penelitian Zulkarnaim et al. (2010) menunjukkan hal yang sama pada sapi
Simmental yang berada pada keseimbangan Hardy-Weinberg, namun berkebalikan
pada sapi Limousin yang tidak berada dalam keseimbangan Hardy-Weinberg. Nilai
Chi-Kuadrat sapi Brahman dan Angus, tidak dapat dihitung karena tidak memenuhi
syarat Chi-Kuadrat, akibat tingkat derajat bebas pada kedua populasi ini bernilai nol
Simmental dan Limousin yang berada pada keseimbangan Hardy-Weinberg bukan
berarti bahwa sapi-sapi pada populasi tersebut tidak mengalami proses seleksi,
namun gen GHR|AluI bukanlah satu-satunya gen yang menjadi indikasi dalam proses
seleksi tersebut.
Heterozigositas
Hasil analisis heterozigositas pada sapi FH dari BIB Lembang, BBIB
Singosari, dan BET Cipelang, sapi Simmental, Limousin, Brahman, dan Angus
disajikan pada Tabel 4.
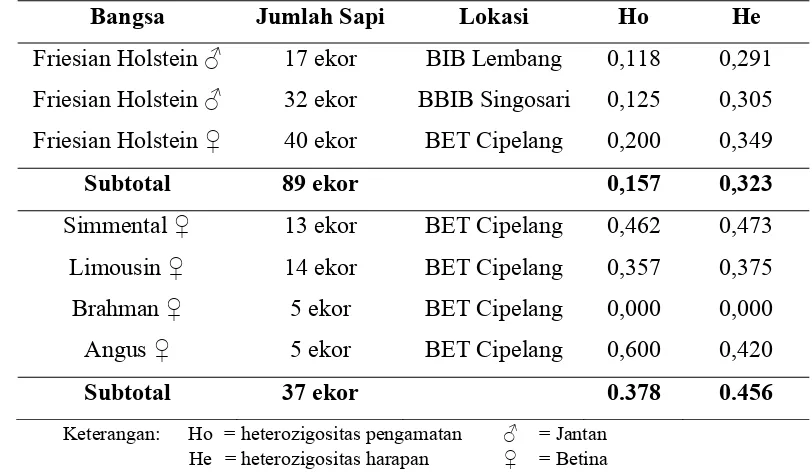
Tabel 4. Nilai Heterozigositas
Bangsa Jumlah Sapi Lokasi Ho He
Friesian Holstein ♂ 17 ekor BIB Lembang 0,118 0,291
Friesian Holstein ♂ 32 ekor BBIB Singosari 0,125 0,305
Friesian Holstein ♀ 40 ekor BET Cipelang 0,200 0,349
Subtotal 89 ekor 0,157 0,323
Simmental ♀ 13 ekor BET Cipelang 0,462 0,473
Limousin ♀ 14 ekor BET Cipelang 0,357 0,375
Brahman ♀ 5 ekor BET Cipelang 0,000 0,000
Angus ♀ 5 ekor BET Cipelang 0,600 0,420
Subtotal 37 ekor 0.378 0.456
Keterangan: Ho = heterozigositas pengamatan ♂ = Jantan He = heterozigositas harapan ♀ = Betina
Pendugaan nilai heterozigositas penting diketahui untuk mendapatkan
gambaran variabilitas genetik (Marson et al., 2005) dan untuk mengetahui tingkat
polimorfisme suatu alel, serta prospek populasi di masa yang akan datang (Falconer
dan Mackay, 1996). Hasil penduga nilai heterozigositas menunjukkan bahwa
heterozigositas pengamatan (Ho) sapi Friesian Holstein di BIB Lembang, BBIB
Singosari, dan BET Cipelang lebih rendah dibandingkan dengan nilai heterozigositas
harapan (He) yaitu berkisar antara 0,118-0,200. Hal ini menunjukkan tingkat
keragaman alel pada populasi tersebut tergolong rendah. Menurut Tambasco et al.
(2003) variasi suatu gen dalam populasi dinyatakan rendah, jika nilai Ho lebih
Javanmard et al. (2005) menambahkan bahwa nilai heterozigositas di bawah 0,5
(50%) mengindikasikan variasi yang rendah suatu gen dalam populasi.
Nilai heterozigositas pengamatan pada sapi Simmental dan Limousin tidak
menunjukkan perbedaan yang besar dengan nilai heterozigositas harapan.
Zulkarnaim et al. (2010) melaporkan hal yang berbeda yang menunjukkan bahwa
nilai heterozigositas pengamatan sapi Simmental lebih tinggi dibandingkan nilai
heterozigositas pengamatan (0,529>0,389±0,021), sedangkan pada sapi Limousin
nilai hetrozigositas pengamatan jauh lebih rendah dibandingkan dengan
heterozigositas harapan (0,095<0,408±0,017). Nilai heterozigositas hasil pengamatan
pada sapi Angus lebih besar dibandingkan nilai heterozigositas harapan dan
merupakan sapi yang memiliki nilai heterozigositas tertinggi. Avise (1994)
menyatakan bahwa semakin tinggi derajat heterozigositas suatu populasi maka daya
tahan hidup populasi tersebut akan semakin tinggi. Sebaliknya, heterozigositas pada
sapi Brahman benilai 0,000 dan merupakan sapi yang memiliki nilai heterozigositas
terendah. Seiring dengan menurunnya derajat heterozigositas akibat dari silang
dalam dan fragmentasi populasi, sebagian besar alel resesif yang bersifat lethal
semakin meningkat frekuensinya (Avise, 1994).
Hartl dan Carlk (1997) dan Vasconcellos et al. (2003) menyatakan bahwa
nilai heterozigositas pengamatan dan nilai heterozigositas harapan juga dapat
digunakan sebagai salah satu cara untuk menduga nilai koefisien biak dalam
(inbreeding) pada suatu kelompok ternak. Nilai heterozigositas, khususnya nilai
heterozigositas harapan secara umum, merupakan indikator yang baik sebagai penciri
genetik yang dapat menjelaskan keragaman genetik pada suatu populasi ternak
KESIMPULAN DAN SARAN Kesimpulan
Gen GHR|AluI pada sapi FH di BIB Lembang, BBIB Singosari, dan BET
Cipelang bersifat polimorfik dengan ditemukannya dua alel, yaitu alel A dan G; serta
tiga genotipe, yaitu AA, AG, dan GG. Frekuensi alel A (0,798) lebih tinggi dari alel
G (0,202). Frekuensi genotipe AA ditemukan paling tinggi pada sapi FH di tiga
lokasi (0,719). Sapi FH pengamatan tidak berada dalam keseimbangan
Hardy-Weinberg dan tingkat heterozigositasnya rendah (0,157).
Sapi pedaging di BET Cipelang memiliki genotipe yang bervariasi. Genotipe
AA, AG, dan GG juga ditemukan pada sapi Simmental dan Limousin. Sapi Angus
hanya memiliki genotipe AA dan AG, dan pada sapi Brahman bersifat monomorfik
dengan genotipe AA (100%). Sapi Simmental dan Limousin berada dalam
keseimbangan Hardy-Weinberg, sedangkan pada sapi Brahman dan Angus nilainya
tidak dapat dihitung. Nilai heterozigositas tertinggi terdapat pada sapi Angus dan
terendah pada sapi Brahman.
Saran
Perlu dilakukan penelitian lebih lanjut untuk mendeteksi keragaman gen
GHR|AluI pada sapi Friensian Holstein. Jumlah sampel yang lebih banyak dengan
populasi dari balai-balai penelitian, industri, dan peternakan rakyat pada berbagai
manajemen yang berbeda-beda akan memberikan gambaran tentang keragaman
genetik yang lebih akurat. Perlu dilakukan penelitian untuk melihat hubungan antara
varian genetik gen GHR|AluI dengan sifat produksi sapi Friensian Holstein dan sifat
UCAPAN TERIMA KASIH
Puji syukur penulis ucapkan kepada Allah SWT yang telah memberikan
rahmat, karunia, dan hidayah-Nya, serta kepada Rasulullah Muhammad SAW atas
tuntunannya sehingga penulis mampu menyelesaikan penelitian dan penulisan
skripsi. Penulis ucapkan terima kasih yang sebesar-besarnya kepada kedua orang tua
tercinta, Bapak Idham Suhaimi, S.ST dan Ibu Eti Setiawati atas kasih sayang dan
do’anya yang tak terhingga. Kakak dan adik, Yesi Riska Perba dan Ines Wishaka
Perba, serta sanak saudara terima kasih atas motivasi dan hiburannya.
Bapak Prof. Dr. Ir. Cece Sumantri, M.Arg.Sc dan Ibu Ir. Anneke Anggraeni,
M.Si, Ph.D terima kasih atas segala perhatian, bimbingan dan masukan selama
penelitian dan penulisan skripsi. Bapak Ir. Andi Murfi, M.Si; Dr. Ir. Ahmad Darobin
Lubis, M.Sc; dan Dr. Rudi Afnan, S.Pt, M.Sc.Agr selaku dosen penguji ujian lisan
terima kasih atas koreksi dan sarannya. Senior LGMT: Kak Erik, Kak Ires, Pak
Ihsan, Pak Andi, Pak Rahmat, Pak Bambang, Pak TR, Kak Surya, Kak Yus, Bu
Suryana, Mbak Mira, Kak Citra, David, Mbak Dina, Bapak Dr. Zakaria, S.Pt, M.Si,
dan Prof. Dr. Ir. Muladno, MSA atas bimbingan dan sarannya. Teman-teman LGMT;
Icha, Fanny, Gabby, Dinny, Priskila, Ulin, Irine, Revy, Lenny, Gina, Desi, Vania,
Cintya, Dian Dinar, dan Ferdy atas kebersamaan dan dukungan semangatnya.
Teman-teman Wisma Mega 1: Uut, Kak Ticut, Dihard, Resti, Henong, Dede
Urang, Umi, Nisful, Keating, Kak Ana, dan Kak Eri, Vivi, Dedek, dan Didi, serta
teman-teman IPTP 44, Tantia, Riri, Tari, Naila, dan Dani terima kasih atas motivasi
dan kebersamaannya. Keluarga besar Ikatan Keluarga Mahasiswa Bumi Sriwijaya
(IKAMUSI) terima kasih atas keceriaan dan hebohnya, serta teman-teman A01-A02,
Asrama Putri A2 lorong 1 kamar 138, teman-teman SD Negeri 4 Kepahiang, SD
Negeri 1 Sembawa, SMP Negeri 3 Banyuasin III, dan keluarga besar SMA Plus
Negeri 2 Banyuasin III terima kasih atas motivasi dan do’anya.
Bogor, 3 Mei 2011
DAFTAR PUSTAKA
Aggrey, S. E., J. Yao, M. P. Sabour, C. Y. Lin, D. Zadworny, J. F. Hayes, & U. Kuhnlein. 1999. Markers within the regulatory region of the growth hormone receptor gene and their association with milk-related traits in Holstein. J. Hered.90: 148-151.
Akers, E. D., J. C. Forrest, D. E. Gerard, & E. W. Mills. 2006. Principles of meat science. Mammary growth and lactation in dairy cows. 4th Ed. J. Dairy Sci. 89: 1222-1234.
Andreas, E. 2010. Telaah kualitas daging serta identifikasi keragaman gen GH dan GHR pada kerbau. Tesis. Sekolah Pascasarjana, Institut Pertanian Bogor, Bogor.
Avise, J.C. 1994. Molecular Markers, Natural History and Evolution. Chapman and Hall, Inc., New York.
Ayuk, J. & M. C. Sheppard. 2006. Growth hormone and its disorders. Postgrad. Med. J.82: 24-30.
Beauchemin, V. R., M. G. Thomas, D. E. Franke, & G. A. Silver. 2006. Evaluation of DNA polymorphisms involving growth hormone relative to growth and carcass characteristics in Brahman steers. Genet. Mol. Res.5: 438-447.
Blakely, J. & D. H Bade. 1998. Ilmu Peternakan. 4th Ed. Terjemahan: Bambang Srigandono. Gadjah Mada University Press, Jogjakarta.
Bloot, S., J. J. Kim, S. Moisio, A. S. Kuntzel, A. Cornet, P. Berzi, N. Cambisano, C. Ford, B. Grisart, D. Johnson, L. Karim, P. Simon, R. Snell, R. Spelman, J. Wong, J. Vilkki, M. Georges, F. Farnir, & W. Coppieters. 2003. Molecular dissection of a quantitative traits locus: a phenylalanine-to-tyrosine substitution in the transmembrane domain of the bovine growth hormone receptor is associated with a major effect on milk yield and composition. Genet. 163: 253-266.
Bradley, D. G, D. E. MacHugh, P. Cunningham, & R. T. Loftus. 1996. Mitochondrial diversity and the origins of African and European cattle. Proc. Natl. Acad. Sci. USA 93: 5131-5135.
Burton, J. L., B. W. Mc. Bride, E. Block, & D. R. Glimm.1994. A review of bovine growth hormone. Can. J. Anim. Sci.74: 167-201.
Curi, R. A., H. N. de Oliveira, A. C. Silveira, & C. R. Lopes. 2006. Growth and
carcass traits associated with GH1|AluI and POU1F1|HinfI gene
polymorphism in Zebu and crossbred beef cattle. Genet. Mol. Bio.29: 56-61.
Djojosoebagio, S. 1996. Fisiologi Kelenjar Endokrin. Penerbit Universitas Indonesia, Jakarta.
Dwiyanto, K., A. Anggraeni, Sugiarti, Nurhasanah, H. Setyanto, & L. Praharani. 2000. Pengkajian sistem budidaya sapi perah untuk meningkatkan produktivitas. Prosiding Hasil Penelitian Bagian Proyek Rekayasa Teknologi Peternakan/ARMP-II. Pusat Penelitian dan Pengembangan Peternakan. Badan Penelitian dan Pengembangan Pertanian. Departemen Pertanian, Bogor.
Falconer, D.S. & T. F. C. Mackay. 1996. Introduction to Quantitative Genetics. 4th Ed. Longman, New York.
Frago, L. M. & J. A. Chowen. 2005. Basic physiology of the growth hormone/insulin-like growth factor axis. In: Varela-Nieto, I. & J. A. Chowen (Eds). The Growth Hormone/Insulin-like Growth Factor Axis During Development. USA: Springer: 1-25.
Ge, W., M. E. Davis, H. C. Hines, K. M. Irvin, & R. C. M. Simmen. 2003. Association of single nucleotide polymorphism in the growth hormone and growth hormone receptor genes with blood serum insulin-like growth factor I concentration and growth traits in Angus cattle. J. Anim. Sci.81: 641-648.
Ge, W., M. E. Davis, H. C. Hines, & K. M. Irvin. 2000. Rapid communication: Single nucleotide polymorphisms ditected in exon 10 of the bovine growth hormone receptor gene. J. Anim. Sci.78: 2229-2230.
Gillespie, J. H. 1998. Population Genetics, A Concies Guide. The Johns Hopkins University Press, London.
Godowski, P. J., D. W. Leung, L. R. Meacham, J. P Galgani, R. Hellmiss, R. Keret, P. S. Rotwein, J. S. Parks, Z. Laron, & W. I. Wood.1989. Characterization of the human growth hormone receptor gene and demonstration of a partial gene deletion in two patiens with Laron-type dwarfism. Proc. Natl. Acad. Sci. USA86: 8083-8087.
Hale, C. S., W. O. Herring, H. Shibuya, M. C. Lucy, D. B. Lubahn, D. H. Keisler, & G. S. Johnson. 2000. Decreased growth in Angus streers with a short TG-microsatellite allele in the P1 promoter of growth hormone receptor gene. J. Amin. Sci.78: 2099-2140.
Han, S. H., I. C. Cho, J. H. Kim, M. S. Ko, H. Y. Jeong, H. S. Oh, & S. S. Lee. 2009. A GHR polymorphism and its association with carcass traits in Hanwoo cattle. Gene. Genom31: 35-41.
Hartl, D. L. & A. G Clark. 1997. Principle of Population Genetic. Sinauer Associates, Sunderland, MA.