Lampiran 1. Gambar Sampel Aksesi Andaliman
Gambar Andaliman Dairi
Lampiran 2. Pembuatan Larutan Stok dan Buffer
A. Pembuatan Larutan Stok
• CTAB 5 % (100 ml): Timbang NaCl sebanyak 2.0 gram dan CTAB sebanyak 5.0 gram. Masukkan bahan kimia ke dalam erlenmeyer dan ditambahkan 100 ml
aquades. Aduk campuran larutan dengan menggunakan stirrer kemudian
diletakkan diatas hote plate.
• Tris HCl 1 M pH 8.0 (100 ml): Timbang Tris sebanyak 12.114 gram. Masukkan Tris ke dalam erlenmeyer dan ditambahkan 80 ml aquades. Aduk
campuran larutan dengan menggunakan stirrer kemudian diletakkan diatas hote
plate. Selanjutnya, ditambahkan 4.2 ml HCl pekat sedikit demi sedikit sampai pH
mencapai 8. Masukkan ke dalam gelas ukur, kemudian volume ditepatkan
dengan aquades hingga volume larutan menjadi 100 ml.
• Tris HCl 1 M pH 7.4 (50 m): Timbang Tris sebanyak 6.057 gram. Masukkan Tris ke dalam erlenmeyer dan ditambahkan 30 ml aquades. Aduk campuran
larutan dengan menggunakan stirrer kemudian diletakkan diatas hote plate.
Selanjutnya, ditambahkan NaOH 2.5 M sedikit demi sedikit sampai pH mencapai
7.4. Masukkan ke dalam gelas ukur, kemudian volume ditepatkan dengan
aquades hingga volume larutan menjadi 50 ml.
• EDTA O.5 M pH 8.0 (100 ml): Timbang EDTA sebanyak 18.612 gram dan NaOH 2.0 gram. Masukkan bahan kimia ke dalam erlenmeyer dan ditambahkan
80 ml aquades. Aduk campuran larutan dengan menggunakan stirrer kemudian
diletakkan diatas hote plate. Selanjutnya, ditambahkan HCl pekat sedikit demi
sedikit sampai pH mencapai 8. Masukkan ke dalam gelas ukur, kemudian
• NaCl 5 M (l00 ml): Timbang NaCl sebanyak 29.22 gram. Masukkan ke dalam erlenmeyer dan ditambahkan 80 ml aquades. Aduk campuran larutan dengan
menggunakan stirrer kemudian diletakkan diatas hote plate. Masukkan ke dalam
gelas ukur, kemudian volume ditepatkan dengan aquades hingga volume larutan
menjadi 100 ml.
**Semua bahan di atas disterilkan dengan menggunakan autoklaf kecuali CTAB 5%.
B. Pembuatan Larutan Buffer
• Buffer Ekstraksi/CTAB (100 ml): Campurkan40 ml CTAB 5%, 25.1 ml NaCl 5 M, 4 ml EDTA 0.5 M pH 8.0, 10 ml Tris HCl 1 M pH 8.0 dan 20.8 ml aquades.
• Buffer TAE 50 X (100 ml): Campurkan 24.2 ml Tris HCl 1 M pH 7.4, 5.7 ml Asam Asetat Glasial, 10 ml EDTA 0.5 M PH 8.0, dan aquades hingga volume
larutan menjadi 100 ml.
• Buffer TAE 1X (500 ml): Campurkan 10 ml Buffer TAE 50 X dan 490 ml aquades.
• Buffer TE (50 ml): Campurkan 0.5 ml Tris HCl 1 M PH 8.0, 0.1 ml EDTA 0.5 M PH 8.0 dan 49.4 ml aquades.
• Kloroform Isoamilalkohol 24 : 1 (50 ml): Campurkan 48 ml Kloroform dan 2 ml Isoamilalkohol.
Lampiran 3. Alur Penelitian
Sampel DNA Andaliman
Isolasi DNA
Uji Kualitas
Uji Kuantitas
Proses PCR-RAPD
Elektroforesis
Lampiran 4. Proses Isolasi DNA
Sampel daun andaliman ditimbang 0.2 gram dan digerus sambil ditambahkan nitrogen cair dan PVPP
Sampel dimasukkan ke dalam tabung yang bberisi 1 ml buffer
ekstraksi dan 10 µl β-mercaptoetanol
Tabung di vortex dan diinkubasi dalam waterbath selama 30
menit pada suhu 650C
Supernatan ditambahkan 1 ml KIAA (24:1) dan disentrifus dengan kecepatan 13.000 rpm selama 10 menit
Supernatan ditambahkan 1 ml KIAA (24:1) dan disentrifus dengan kecepatan 13.000 rpm selama 10 menit
Supernatan ditambahkan 1 ml isopropanol dan diinkubasi pada
suhu 40C selama semalama
Tabung disentrifus pada pada kecepatan 13.000 rpm selama 10 menit dan dikeringkan
Pelet dilarutkan dengan buffer TE 100µl
Campuran ditambahkan dengan etanol absolute dingin dan
diinkubasi dalam freezer (-200C) selama 30 menit
Campuran disentrifus dengan kecepatan 13.000 rpm selama 10 menit
Pelet dicuci dengan etanol 70% dan dikeringkan
Pelet dilarutkan dengan 100 µl buffer TE
Lampiran 4. Proses PCR-RAPD
Komposisi Master Mix volume 25 µl : Go Taq PCR 12.5 µl
Nuclease free water 9.5 µl Primer 1 µl DNA sampel 2 µl
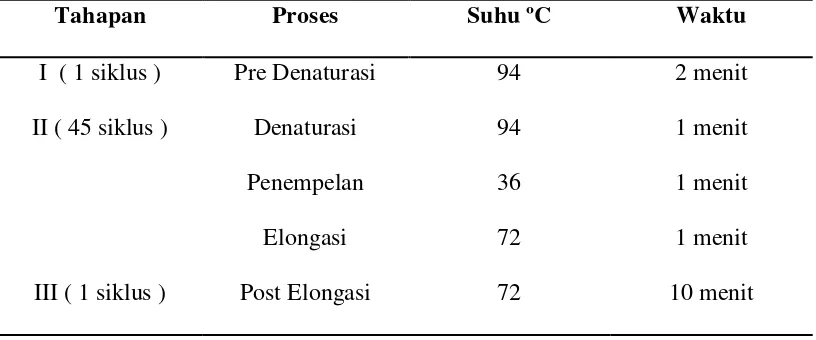
Running PCR sebanyak 45 siklus :
Denaturasi awal 940C 2 menit
Denaturai 940C 1 menit
Annealing 360C 1 menit
Extension 720C 2 menit
Post extension 720C 10 menit
Kondisi akhir PCR 40C tak terbatas
Lampiran 5. Proses elektroforesis hasil PCR-RAPD
Agar rose 2.6 gram ditambahkan dengan 130 ml buffer TAE 1 x
Campuran dipanaskan dengan hot plate
Campuran ditambahkan 1.5 µl EtBr
Larutan gel dituang kedalam cetakan yang telah dipasang sisir
Gel yang telah mengeras dipindahkan ke dalam chamber
berisi buffer TAE 1 x
Sampel hasil PCR, marker dan loading dye dimasukkan ke dalam
sumur gel dengan perbandingan (8:5:2)
Alat elektroforesis dihubungkan dengan power supply 80 volt
Lampiran 6. Foto Uji Kuantitas
Gambar 10. Alat spektrofotometer
Lampiran 8. Hasil Fotosintesis
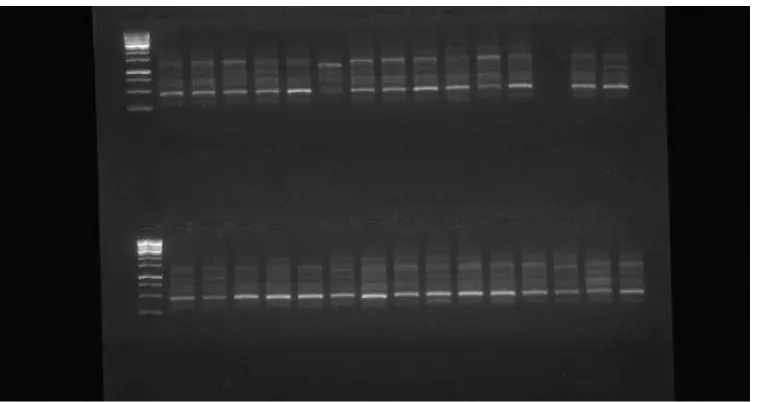
Gambar 12. Hasil elektoroforesis dengan Primer OPD-13
Gambar 13. Hasil elektroforesis dengan Primer OPI-20
Gambar 14. Hasil elektoforesis dengan Primer OPN-10
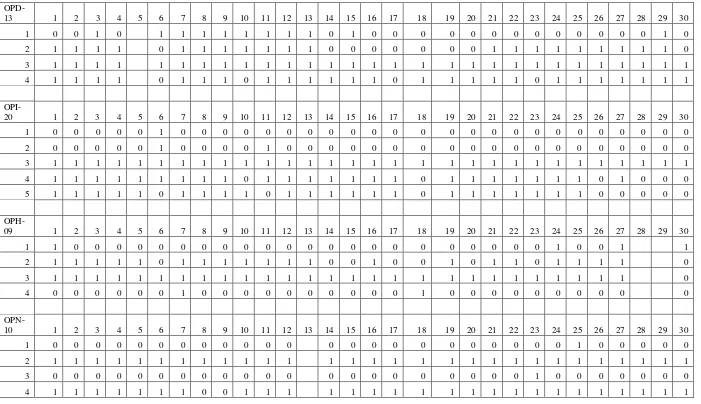
Lampiran 9. Hasil Skoring Pita 30 Individu Pada Kelima Primer
5 0 1 0 1 0 1 1 0 1 1 0 1 0 0 1 1 0 1 0 0 0 0 0 0 0 0 0 0 0 6 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 OPM-
01 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 0 0 1 1 1 1 1 1 1 1 1 1 3 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 4 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 5 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 6 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0
DAFTAR PUSTAKA
Azizah, A. 2009. Perbandingan Pola Pita Amplifikasi Dna Daun, Bunga Kelapa Sawit Normal dan Abnormal. Institut Pertanian Bogor . Bogor
Andayanie, L. 2000. Kajian Daya Insektisida Alami Nabati Kulit Buah Manggis (Garcinia mangostana L), buah Andaliman (Zanthoxylum acanthopodium DC.), getah Gambir (Uncaria gambir Roxb.)dan daun the (Camellia sintetis L.) terhadap perkembangan hama gudang Sitophilus zeamais Motsch.
Skripsi. Bogor: Fakultas Teknologi pertanian IPB. Hal. 36.
Bahagiawati. 2011. Peran Markah Molekuler Dalam Pemuliaan Tanaman. Badan Litbang Pertanian. Edisi 16-22 Maret 2011 No.3397 Tahun XLI.
Bardakci,F. 2001. Random Ampilfied Polymorphic DNA (RAPD) markers. Turk.J.
Biol. 25: 185-37.
Elfrod, S. dan Stansfield, W. 2007. Genetika. Edisi Keempat. Penerjemah :Damaring, T. W. Penerbit Erlangga. Jakarta.
Ferreira, M.E dan D. Gratapaglia.1998. Introducao ao uso de marcadores em analise genetic. Embrapa-Cenargen.Brasilia.
Hasairin, A. 1994. Etnobotani Tanaman Rempah dalam Makanan Adat
Masyarakat Batak Angkola Dan Mandailing. Thesis. Bogor: Program
Pascasarjana IPB
Hsuang, Keng, 1978. Orders and Families of Malayan Seed Plants. Dalam Wijaya , C.H. 1999. Andaliman, Rempah tradisional Sumatera Utara dengan aktivitas antioksidan dan antimikroba. Bul.Teknol. dan Industri Pangan. 10(2): 58-65.
Hutami,S., I.Mariska, dan Y.Supriati. 2005 . Peningkatan Keragaman Genetik Tanaman melalui Keragaman Somaklonal. J. Agro. Biogen.2(2):81-88. Jones, C.J., K.J. Edwards, S. Castagiole, M.O.Winfield, F. Sala, C. van del Wiel,
G. Bredemeijer, B. Vosman, M. Matthes, A. Dally, R. Brettsshneider, P. Bettini, M, Buiatti, E. Maestri, A. Malcevschi, N.Marmiroli,R. Aert, G. Volkaert, J. Rueda, R. Linacero, A. Vasques and A. Karp. 1997. A
Reproducibility testing of RAPD, AFLP and SSR markers in plants by a network of European laboratories. Molecular Breeding 3 (5): 382-390
Lim, L.S, R. Wickneswari, S. L. Lee and A. Latif. 2002. Genetic variation of
Dryobalanops aromatica Gaertn. F. (Dipterocapaceae) in Peninsular
Malaysia using microsatellite DNA markers. Forest Genetics 9 (2) :
Karsinah, Sudarsono, Setyobudi, L., dan Aswidionnoor, H., 2002. Keanekaragaman Genetik Plasma Nutfah jeruk berdasarkan AnalisisPenanda RAPD. Jurnal Bioteknologi Pertanian 7 (1) : 8-16
Maftuchah dan A. Zainuddin. 2013. Studi Pendahuluan variasi genetik jarak pagar (Jatrpha curcas L.) lokal berdasarkan Random Amplified Polymorphic DNA . Pusat pengembangan Bioteknologi universitas Muhammadiyah Malang (123-131).
Mahardika, I.G.N.K. 2003. Polymerase Chain Reaction. Laboratorium Virologi. Fakultas Kedokteran Hewan.Universitas Udayana. Denpasar.
Mangoendidjojo, W., 2003. Polymerase Chain Reaction. Laboratorium Virologi. Fakultas Kedokteran Hewan. Universitas Udayana. Denpasar.
Mulia,L.2000. Kajian Aktivitas Antimikroba Andaliman (Zanthoxylum acanthopodium ) dan Antarasa (Litsea cubeba). Skkripsi . Institut Pertanian
Bogor .Bogor, hlm 7-21.
Murray, RK, Graner DK, Mayes PA Rodwell VW. 2000. Biokimia Harper.
Andry H, penerjemah; Anna PB, Tiara MN, editor. Jakarta: Penerbit Buku Kedokteran EGC. Terjemahan dari: Harper’s Biochemistry.
Napitupulu,B., Sorth,S., dan Mery, S., 2004. Potensi andaliman sebagai Food Additive tradisional etnis batak Sumatera Utara. BPTP Sumatera Utara. Medan, Hlm. 53-56
Pandin , D.S., 2010. Penanda DNA untuk Pemuliaan Tanaman kelapa (Cocos nucifera L.) Perspektif 9 (1) ;21-35.
Parhusip, A.J., Posman S., Adelina T. 1999. Studi Tentang Aktifitas Antimikroba Alami pada Andaliman. Seminar Nasional Teknologi Pangan.
Prana,T.K dan S.N Hartati. 2003. Identifikasi sidik jari DNA Talas (Colocasia esculenta L.Schott) Indonesia dengan teknik RAPD. Jurnal Natur Indonesia
5(2): 107-112.
Rahayu, E.S., dan S., Handayani, 2010. Keragaman genetik pandan asal Jawa Barat berdasarkan penanda inter simple sequence repeat. Marka Sains 14 ; 158-162.
Rholf, F. J. 2000.NT SYS-pc: Numerical Taxonomy and Multivariate Analysis System Version 2.1 User Guide. Department of Ecology and Evolution State University of New York.
Setiyo, I.E., 2001. Pemetaan dan Keragaman Genetik RAPD pada Kelepa Sawit sungai pancur (RISPA). Tesis. Program Pascasarjana IPB. Bogor.
Simajuntak, SP. 2006. Perkecambahan Biji Andaliman (Zanthoxylum
acanthopodium DC) Deskripsi dan Perkecambahan. Hayati Vol.10 No.1
Siregar, B.L., 2003. Andaliman (Zanthoxylum Acanthapodium DC.) di Sumatera
Utara; Deskripsi dan Perkecambahan. J. Hayati hlm. 38-40.
Suryanto D. 2003. Melihat Keanekaragaman organisme melalui beberapa teknik genetika molekuler . USU Digital Library [terhubung berkala].
Wijaya, C. H. 1999. Andaliman, Rempah Tradisional Sumatera Utara Dengan Aktivitas Antioksidan dan Antimikroba. Bul. Teknol. Dan Industri Pangan. 10(2).
Wijaya, C.H. 2000. Isolasi dan Identifikasi senyawa trigeminal aktif buah andaliman (Zanthoxylum acanthopodium DC). Hayati 7:9-95.
METODE PENELITIAN
Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan di Laboratorium Terpadu Fakultas Kedokteran Universitas sumatera Utara, Medan yang akan dimulai pada bulan April 2015 sampai dengan bulan September 2015.
Bahan dan Alat
Bahan tanaman digunakan dalam penelitian ini adalah materi genetik DNA Andaliman yang sudah di isolasi peneliti yang sebelumnya Sinaga (2014) pada tiga populasi alami tanaman aren hasil koleksi dari berbagai lokasi di Sumatera Utara
Bahan kimia digunakan adalah adalah cetyl trimethylammonium bromide
(CTAB) 5%, NaCl, Tris, HCl, NaOH, isopropanol, ethylenediamine tetraacetic
(EDTA), Asam Asetat Glasial, agarose, Ethidium Bromide (EtBr), kloroform,
isoamilalkohol, β-Mercaptoethanol, polyvinilpyrolidone (PVPP), etanol 70%,
etanol absolut, DNA Marker I00 bp Ladder, Go Taq ® Green Master Mix, nitrogen cair, loading dye, aquades, aquabidestila dan Primer OPD-13, OPI-20, OPH-09. OPM-01 dan OPN-10.
Alat yang digunakan dalam penelitian ini adalah gunting, timbangan digital, hotplate, mortar, centrifuge, freezer, vortex, freezer, tabung eppendorf 2
ml dan 1,5 ml, mikropippete 1-50 µl, 100-500 µl dan 200-1000 µl, sarung tangan karet, tip pipet kristal, kamera,oven, pH meter, pengaduk magnetik, alat-alat gelas (beaker gelas, erlenmeyer, dll), UV Transluminator (UV Tec Cambridge 20 UV),
elektroforesis (Power PAC 3000, Biorad), PCR (Therma Cycler), Gel-Doc (UV
Metode Penelitian
Metode penelitian yang digunakan adalah Metode RAPD (Random
Amplified Polymorphic DNA) berdasarkan PCR (Polymerase Chain Reaction)
Pelaksanaan Penelitian
Pengambilan Sampel Daun
Daun andaliman yang digunakan adalah daun dari populasi alam di daerah kabuupaten dairi dan sekitarnya. Daun dipilih yang masih muda/lembut berwana hijau kemerahan, kemudian dicuci bersih , dilap pakai tisu dan kemudian dibawa ke Laboratorium Terpadu Fakultas Kedokteran.
Isolasi DNA
Tahapan isolasi yang dilakukan adalah daun andaliman dibuang tulang daunnya lalu dicuci dan dikeringkan dengan tisu dan ditimbang masing-masing 0,1-0,3 gram. Daun dipotong halus dengan gunting secara melintang. Daun andaliman digerus menggunakan mortar sambil ditambahkan nitrogen cair dan PVPP. Setelah halus, sampel dimasukkan ke dalam tabung sentrifus yang berisi 1 ml buffer ekstraksi dan β-mercaptoetanol 10 µl kemudian tabung dikocok menggunakan vortex lalu tabung diinkubasi dalam waterbath selama 30 menit
pada suhu 650C. Setiap 10 menit sekali tabung dibolak balik dengan perlahan-lahan. Setelah itu, tabung diinkubasi pada suhu ruang selama 4-5 menit lalu 1 ml kloroform:isoamilalkohol (24:1) ditambahkan ke dalam tabung.
Tabung disentrifus dengan kecepatan 13.000 rpm pada suhu ruang selama 10 menit. Supernatan yang diperoleh dipindahkan pada tabung sentrifus lain, lalu 1 ml kloroform:isoamilalkohol (24:1) ditambahkan ke dalam tabung lalu tabung dikocok menggunakan vortex dan tabung disentrifus lagi dengan kecepatan
dalam lemari es (40C) selama satu malam kemudian tabung disentrifus kembali
dengan kecepatan 13.000 rpm pada suhu 40C selama 10 menit. Supernatan yang diperoleh dibuang kemudian pelet dikeringanginkan. Pelet yang sudah kering dilarutkan dengan buffer TE sebanyak 100 µl kemudian tabung dispin manual hingga homogen.
Kemudian etanol absolut dingin ditambahkan lalu dibolak-balik hingga homogen. Setelah itu, supernatan diinkubasi dalam freezer (-200C) selama 2 jam
kemudian supernatant disentrifus lagi dengan kecepatan 13.000 rpm pada suhu 40C selama 10 menit. Supernatan dibuang sedangkan pelet dicuci menggunakan etanol 70% dan pellet dikeringanginkan. Pelet DNA yang sudah kering dilarutkan dengan 100 µl buffer TE. Simpan DNA dalam freezer (-200C).
Uji Kuantitas DNA
Pengujian dilakukan dengan menggunakan alat spektrofotometri. Larutan stok DNA diambil sebanyak 2 µl, kemudian alat dijalankan. Absorbansi (A) diukur pada panjang gelombang 260 nm dan 280 nm. Tingkat kemurnian DNA ditentukan dengan nilai perbandingn A260/A280. Menurut Wilson dan Walker
(2010), sampel DNA murni akan menghasilkan rasio A260/A280 berkisar 1.8-2.0.
Nilai kemurnian yang lebih dari 2.0 menunjukkan bahwa sampel mengandung kontaminan RNA, sedangkan nilai kemurnian yang kurang dari 18 menunjukkan bahwa sampel mengandung kontaminan protein (Yuwono, 2006).
Amflifikasi dan Genotyping
Amflifikasi mengikuti prosedur baku analisis RAPD, sesuai prosedur Wiliiam et al., (1990). Amflifikasi dilakukan dengan menggunakan 5 primer
Sebelum running PCR dilakukan pengenceran DNA dengan mengambil 5 ɰl stok DNA dan ditambahkan 45 µ ml ddH2O sehinnga diperoleh 50 µl aliquot
DNA . kemudian dilakukan pengenceran primer yaitu tube primer disentrifius 5 menit setelah itu ditambahkan dd H2O sesuai ukuran molar. Dibuat aliquot primer yaitu dengan mengambil 10-15 ɰl stok primer.
Persiapan awal PCR adalah mencairkan komponen untuk running PCR yaitu paket PCR produksi Promega dalam kotak berisi pecahan es. Untuk mempermudah pembuatan larutan master dimisalkan 5 sampel yang akan digunakan maka larutan master terdiri atas :dd H²O 9.5 ɰ x 5= 47.5 ɰl, Go tag 12,5 x 5 = 62,5 ɰl, aliquot primer 1 ɰ x 5 = 5 ɰl. Dari tube diambil 23 ɰl ke tube yang lain sehingga diperoleh 20 tube untuk PCR dan ditambahkan masing-masing DNA sebanyak 2 ɰl. Kemudian tabung dispin manual. Tabung berisi stok DNA dan campuran master dimasukkan dalam block sampel dimesin PCR dengan suhu annealing 36ºC. Reaksi amplifikasi Gene Amp PCR Applied
Biosystem di desain waktu,suhu dan jumlah siklus termal 45 kali (3 jam 51 menit)
berdasarkan yang digunakan peneliti Setiyo (2001). Proses amplifikasi PCR dapat dilihat pada tabel 1.
Setelah reaksi PCR selesai DNA hasil amplifikasi disimpan dalam suhu ±20º C bila sedang tidak digunakan.
Elektroforesis
Sebelum dilakukan elektroforesis disiapkan gel agarose konsentrasi 2 % (b/v). Agarose ditimbang 2,6 g kemudian dilarutkan dengan menambahkan 130 ml buffer TAE 1x. Larutan tersebut dimasukan ke dalam elenmeyer, kemudian dipanaskan dan diaduk dengan pengaduk magnetik hingga larutan menjadi bening. Setelah larutan dipanaskan kemudian didingikan ditambah larutan etidium bromide 1 µl kemudian dipanaskan kembali lalu didinginkan dengan cara yang sama. Setelah larutan agak dingin ( suhu ± 60º C) larutan dimasukkan dalam cetakan agar yang telah dipasang sisir pembuat lubang ( well-forming combs) dan dibiarkan memadat selama ± 45 menit atau sampai gel mengeras. Well-forming
combs dilepas secara perlahan dan gel agarose siap digunakan untuk
elektroforesis.
Untuk elektroforesis tray yang berisi gel agarose diletakkan dalam tank elektroforesis dan larutan buffer TAE 1x dituang ke dalam tank tersebut ± 500 ml (hingga terendam) hingga 1 mm diatas permukaan gel atau sampai batas yang telah ditemukan. Contoh DNA yang telah disiapkan dimasukkan ke dalam sumur pada gel.
Setelah semua sampel dimasukkan ke dalam sumur (well), tank
telah dielektroforesis dilakukaan dengan UV transluminator dan jika pita/band molekul DNA kelihatan terang maka didokumentasikan.
Analisis Data
Penentuan Skoring Marka RAPD
Untuk menentukan keragaman genetik, produk PCR – RAPD diskoring berdasarkan muncul tidaknya pita DNA. Pita yang muncul pada gel diasumsikan sebagai alel RAPD. Keragaman alel RAPD ditentukan dari perbedaan migrasi alel pada gel masing-masing individu sampel. Berdasarkan ada atau tidaknya pita, profil pita diterjemahkan ke dalam data biner. Pita yang muncul diberi kode -1(ada) dan -0 (tidak ada) ( Ferreira dan Grattapaglia, 1994).
Penentuan Ukuran Pasangan Basa
Ukuran fragmen basa (pasangan basa = bp) produk PCR ditentukan dengan menggunakan software UVITEC Cambridge FireReader. Fragmen DNA yang digunakan yaitu 100 bp DNA ladder . Dengan menggunakan software UVITEC Cambridge FireReader maka ukuran pita DNA hasil amplifikasi dapat terukur seperti pada gambar. Ukuran pita DNA (base pairs) ini akan berpacuan dari ladder yang kita gunakan. Program ini akan mengukur pita yang muncul berdasarkan ukuran ladder yang digunakan. Pengukuran pola pita yang terbentuk ini dengan pendar cahaya DNA yang terbentuk saat proses elektroforesis dengan sinar UV.
Matriks jarak atau ketidaksamaan genetik untuk semua kombinasi pasangan individu dapat dilakukan dengan dua tipe analisis deskriptif dari keragaman : (1)
Principal Coordinates Analysis (PCoA), suatu jenis analisis faktorial pada tabel
Tree (NJtree) berdasarkan Saitou dan Nei (1978) untuk memperoleh gambaran
HASIL DAN PEMBAHASAN
Data Geografis dan Morfologis 30 Aksesi Andaliman
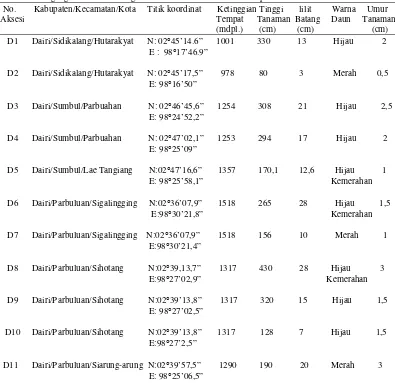
Tiga puluh sampel aksesi andaliman yang diambil dari 3 kabupaten (Dairi, Tanah Karo, dan Simalungun) menunjukkan adanya perbedaan warna daun bagian bawah. Setiap sampel menunjukkan tingkat kemerahan setiap daun berbeda, sehingga dikelompokkam pada 3 tingkat warna, yaitu berwarna hijau, hijau kemerahan dan merah. Data dapat dilihat pada tabel 2.
Baru E : 98°43’44”
Umur aksesi andaliman yang digunakan sebagai sampel umumnya tanaman yang berumur 1- 4 tahun. Gambar 14,15,16 dan 17 (Lampiran 2), menunjukkan perbedaan tinggi dan kanopi tanaman andaliman yang digunakan sebagai sampel, namun tidak dibahas dalam penelitian ini.
Uji Kuantitas DNA
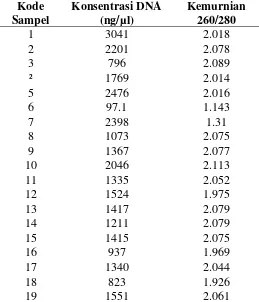
Uji kuantitatif DNA dilakukan secara spektrofotometri pada panjang gelombang 260 nm dan 280 nm sehingga diperoleh nilai kemurnian dan konsentrasi DNA hasil isolasi. Panjang gelombang 260 nm merupakan serapan maksimum untuk asam nukleat, sedangkan panjang gelombang 280 nm merupakan serapan maksimum untuk protein. Hasil pengukuran dapat dilihat pada Tabel 3.
Tabel 3.Hasil uji kuantitatif 30 aksesi DNA tanaman andaliman
20 1801 2.073
Kemurnian DNA yang diperoleh pada penelitian ini berkisar 1.143 – 2.113. Dari 30 sampel DNA yang diisolasi hanya 4 sampel yang nilai kemurnian nya berkisar 1.8 – 2.0. Yaitu aksesi nomor 12, 16, 18, dan 28. Menunjukkan bahwa sampel DNA telah murni.
Sampel dengan nilai kemurnian dibawah 1.8 sebanyak 4 sampel. Yaitu aksesi nomor 6, 7, 26 dan 27. Hal ini menunjukkan bahwa stok DNA masih banyak mengandung polysakarida. Sedangkan sampel dengan kemurnian diatas 2.0 sebanyak 22 sampel. Yaitu aksesi nomor 1, 2, 3, 4, 5, 8, 9, 10, 11, 13, 14, 15, 17, 19, 20, 21, 22, 23, 24, 25, 29 dan 30. Hal ini menunjukkan bahwa sampel masih belum murni dan mengandung banyak protein.
Prinsip dasar pada spektrofotometri adalah sampel harus jernih dan larut sempurna. Tidak ada partikel koloid apalagi suspensi. DNA yang mengandung basa-basa purin dan pirimidin dapat menyerap cahaya UV. Pita ganda DNA dapat menyerap cahaya UV pada 260 nm, sedang kontaminan protein atau phenol dapat menyerap cahaya pada 280 nm. Dengan adanya perbedaan penyerapan cahaya UV ini, sehingga kemurnian DNA dapat diukur dengan menghitung nilai absorbansi 260 nm dibagi dengan nilai absorbansi 280 (Å260/Å280)
Menurut Haris et al. (2003), konsentrasi DNA akan berdampak pada
kualitas fragmen hasil amplifikasi. Konsentrasi DNA yang terlalu rendah akan menghasilkan fragmen yang sangat tipis pada gel atau bahkan tidak terlihat secara visual, sebaliknya konsentrasi DNA yang terlalu tinggi akan menyebabkan fragmen terlihat tebal sehingga sulit dibedakan antara satu fragmen dengan fragmen lainnya.
Salah satu keuntungan pemakaian analisis keragaman genetik tanaman dengan menggunakan teknik molekuler yang memanfaatkan teknologi amplifikasi PCR adalah kuantitas DNA yang diperlukan hanya sedikit. Di samping itu, dalam pelaksanaan teknik RAPD tingkat kemurnian DNA yang dibutuhkan tidak perlu terlalu tinggi, atau dengan kata lain teknik amplifikasi PCR relatif toleran terhadap tingkat kemurnian DNA. Meskipun demikian, dalam suatu teknik isolasi DNA masih diperlukan suatu tahapan untuk meminimalkan senyawa-senyawa kontaminan yang dapat mengganggu reaksi PCR seperti polisakarida dan metabolit sekunder. Hal ini disebabkan keberadaan polisakarida dan metabolit sekunder dalam sel tanaman sering menyulitkan dalam isolasi asam nukleat (Maftuchah dan Zainuddin, 2013).
Hasil PCR dengan marka RAPD
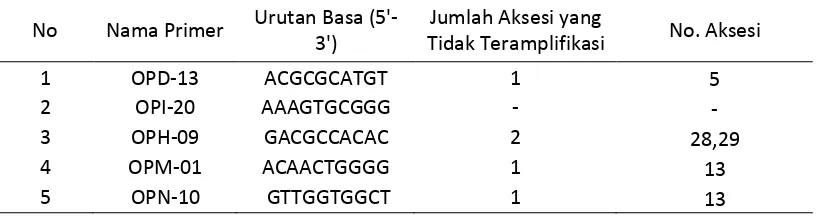
Satu dari lima primer yang digunakan mengamplifikasi DNA pada 30 Aksesi andaliman yaitu Primer OPI-20 Sedangkan 4 Primer lagi yaitu : OPD-13, OPH-09, OPM-01, OPN-10 tidak mengamplifikasi DNA pada 30 aksesi. Jumlah DNA yang paling banyak teramplifikasi terdapat pada OPH-09 yaitu aksesi nomor 28,29, dan 30 . Sedangkan yang paling sedikit terdapat pada Primer OPD-13, OPM-01, OPN-10 yaitu 1 pada aksesi no 13. Sementara OPH-09 tidak mengamplifikasi DNA 2 aksesi pada aksesi nomor 28, dan 29.
Tabel 4. Hasil Amplifikasi Lima Primer yang Digunakan
No Nama Primer Urutan Basa
(5'-Pita yang muncul memiliki ukuran basa dan intensitas pita yang bervariasi. Perbedaan intensitas pita DNA dipengaruhi oleh sebaran situs penempelan primer pada genom, kemurnian dan konsentrasi genom dalam reaksi. Banyaknya pita yang dihasilkan oleh setiap primer tergantung pada sebaran situs yang homolog pada genom (Williams dkk, 1990). Fragmen yang tidak muncul disebabkan tidak terjadinya amplifikasi, terjadi karena munkin primer yang digunakan tidak sesuai dengan DNA cetakan. Beberapa bukti percobaan menunjukkan bahwa perbedaan satu pasang basa saja sudah cukup menyebabkan ketidaksesuaian cetakan primer yang kemudian mencegah amplifikasi (William dkk., 1990).
atau yang terlalu tinggi menyebabkan tidak terjadinya amplifikasi. Rasio yang rendah antara primer dan DNA cetakan dapat menyebabkan produk RAPD yang dihasilkan tidak konsisten. Magnesium merupakan komponen yang penting dalam reaksi PCR dan mempengaruhi kualitas profil RAPD yang dihasilkan (Pharmawati, 2009). Magnesium mempengaruhi penempelan primer serta aktifitas enzim (Padmalatha dan Prasad, 2006). Konsentrasi MgCl2 yang tinggi juga mempengaruhi jumlah band yang dihasilkan dan mengakibatkan penurunan intensitas band tertentu.
Secara umum, hasil penelitian ini menunjukkan bahwa pola pita yang dihasilkan oleh lima primer yang digunakan memperlihatkan pola pita yang berbeda. Ukuran pita-pita yang dihasilkan bervariasi antara 320 bp – 2.540 bp. Total pola pita dari kelima primer yang tampak sebanyak 25 dengan rata-rata 5 pita per primer dengan dengan pita polimorfik sebanyak 21 pita dan pita yang monomorfik sebanyak 4 pita. Persentase pita yang polimorfik bervariasi sebesar 75% sampai 100% dengan rata-rata 84.20% untuk seluruh primer.
250 bp
Jumlah pola pita tertinggi terdapat pada primer OPM 01 dan OPN 10 yang berjumlah 6 pola pita sedangkan jumlah pola pita terendah terdapat pada primer OPD 13 dan OPH 09 yang berjumlah 4 pola pita. Ukuran pita tertinggi terdapat pada primer OPH 09 sebesar 2540 bp sedangkan ukuran pita terendah terdapat pada primer OPI 20 sebesar 320 bp.
Primer OPD-13 menunjukkan pola pita yang berjumlah 4 pita dengan ukuran pita berkisar 468.238 – 2462.792 bp. Persentase pita polimorfik sebesar 75% dan persentase monomorfis sebesar 25 %.
Gambar 2.Elektroforegram amplifikasi 30 DNA Andaliman dengan primer OPD-13, ket ; M = marker ladder 100 bp , Kab Dairi : (1-18), Kab Karo :
(19-21), Dan Kab Simalungun (22-30)
Primer OPI-20 menunjukkan pola pita yang berjumlah 5 pita dengan ukuran pita berkisar 320.415 – 1717.12 bp . Persentase pita polimorfis sebesar 80% . Dan persentase monomorfis sebesar 20%.
Gambar 2.Elektroforegram amplifikasi 30 DNA Andaliman dengan primer OPI-20, ket ; M = marker ladder 100 bp , Kab Dairi : (1-18), Kab Karo :
(19-21), Dan Kab Simalungun (22-30)
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30
Primer OPH-09 menunjukkan pola pita yang berjumlah 4 pita dengan ukuran pita berkisar 500 bp – 2540.66 bp. Persentase pita polimorfis sebesar 100 %. Dan persentasse monomorfis sebesar 0 %.
Gambar 4.Elektroforegram amplifikasi 30 DNA Andaliman dengan primer OPH-09, ket ; M = marker ladder 100 bp , Kab Dairi : (1-18), Kab Karo :
(19-21), Dan Kab Simalungun (22-30)
Primer OPN-10 menunjukkan pola pita yang berjumlah 6 pita dengan ukuran pita berkisar 386.465 bp – 1297.55 bp. Persentase pita polimorfis sebesar 83 %. Dan persentase monomorfis sebesar 17%.
Gambar 5. Elektroforegram amplifikasi 30 DNA Andaliman dengan primer OPN-10, ket ; M = marker ladder 100 bp , Kab Dairi : (1-18), Kab Karo :
(19-21), Dan Kab Simalungun (22-30)
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30
Gambar 6. Elektroforegram amplifikasi 30 DNA Andaliman dengan primer OPM-01, ket ; M = marker ladder 100 bp , Kab Dairi : (1-18), Kab Karo :
(19-21), Dan Kab Simalungun (22-30)
Keberhasilan suatu primer dalam mengamplifikasi DNA cetakan ditentukan oleh ada tidaknya homologi sekuen nukleotida primer dengan sekuen nukleotida DNA cetakan. Selain itu juga dipengaruhi oleh kualitas dan kuantitas DNA, konsentrasi MgCl2, enzim Taq DNA polimerase, dan suhu pelekatan
primer (Wibowo, 2010).
Menurut Yuwono (2006) primer yang tidak spesifik dapat menyebabkan teramplifikasinya daerah lain dalam genom yang tidak dijadikan sasaran atau sebaliknya tidak ada daerah genom yang teramplifikasi. Keberhasilan teknik ini ditentukan oleh ada tidaknya situs penempelan primer, kemurnian DNA dan keutuhan DNA cetakan (Bardakci, 2001). Konsentrasi DNA genom merupakan faktor terpenting dalam reaksi amplifikasi. Konsentrasi DNA yang terlalu tinggi dapat meningkatkan kontaminan yang menggangu reaksi amplifikasi
(Chen, 2000).
menyangkut suhu denaturasi dan annealing DNA dalam mesin PCR. Suhu
denaturasi yang rendah dapat menyebabkan belum terbukanya DNA utas ganda sehingga tidak dimungkinkan terjadinya polimerisasi DNA baru. Proses penempelan primer pada utas DNA yang sudah terbuka memerlukan suhu optimum, sebab suhu yang terlalu tinggi dapat menyebabkan amplifikasi tidak terjadi atau sebaliknya suhu yang terlalu rendah menyebabkan primer menempel pada sisi lain genom yang bukan sisi homolognya; akibatnya dapat teramplifikasi banyak daerah tidak spesifik dalam genom tersebut. Suhu penempelan (annealing)
ini ditentukan berdasarkan primer yang digunakan yang dipengaruhi oleh panjang dan komposisi primer (Suryanto, 2003).
Konsentrasi primer berpengaruh terhadap intensitas produk PCR-RAPD. Menurut Padmalatha dan Prasad (2006) konsentrasi primer yang terlalu rendah atau yang terlalu tinggi menyebabkan tidak terjadinya amplifikasi. Rasio yang rendah antara primer dan DNA cetakan dapat menyebabkan produk RAPD yang dihasilkan tidak konsisten. Magnesium merupakan komponen yang penting dalam reaksi PCR dan mempengaruhi kualitas profil RAPD yang dihasilkan (Pharmawati, 2009). Magnesium mempengaruhi penempelan primer serta aktifitas enzim (Padmalatha dan Prasad, 2006). Konsentrasi MgCl2 yang tinggi juga mempengaruhi jumlah band yang dihasilkan dan mengakibatkan penurunan intensitas band tertentu.
dihasilkan oleh setiap primer tergantung pada sebaran situs yang homolog pada genom (Williams et al., 1990). Adanya perbedaan pola pita yaitu berdasarkan
jumlah dan ukuran pita menggambarkan adanya genom tanaman yang sangat kompleks (Grattapaglia et al., 1992).
Menurut Demeke dan Adams (1994), amplifikasi DNA dengan primer acak pada analisis RAPD biasanya menghasilkan 5-20 fragmen untuk setiap primer. Jumlah fragmen hasil amplifikasi dengan RAPD memang lebih rendah dibandingkan dengan hasil amplifikasi menggunakan AFLP (Haris et al., 2003).
Kelemahan RAPD adalah pemunculan pita DNA kadang–kadang tidak konsisten. Hal ini lebih sering terjadi jika suhu annealing yang digunakan terlalu
Analisis Hubungan Genetik Andaliman
Berdasarkan elektroforesis hasil amplifikasi dengan menggunakan program software Darwin 5.05 (Perrier dan Jacquemoud-Collet, 2009) dari 30 aksesi yang dianalisis hanya 26 aksesi yang diproses oleh software, karena ada beberapa aksesi yang tidak memenuhi persentasi yang distandarkan.
Data biner hasil skoring amplifikasi 5 primer yang diolah dengan software DARwin dihasilkan radial filogenetik yang menunjukkan kekerabatan aksesi andaliman dimana 26 aksesi andaliman yang dikelompokkan menjadi 3 klustering. Kluster I terbagi lagi menjadi 2 subkluster yaitu : subkluster IA dan subkluster IB. Subkluster IA terdiri dari aksesi yang berasal dari Dairi (D18), Karo (K2 dan K3) , dan Simalungun (S2, S3, S5 dan S8) yang mengelompok secara acak (D, K dan S). Subkluter IB terdiri dari aksesi yang berasal dari Simalungun (S1 dan S4).
Kluster II terbagi lagi menjadi 2 subkluster yaitu subkluster IIA dan IIB. Subkluster IIA terdiri atas aksesi yang berasal dari Dairi (D3, D7, D8, D9, D10, D11, dan D12), dan subkluster IIB terdiri dari aksesi yang berasal dari Dairi (D2 dan D4) .
Kluster III terbagi lagi menjadi 3 subkluster yaitu subkluster IIIA dan IIIB. Subkluster IIIA terdiri atas aksesi yang berasal dari Dairi (D1, D14 dan D15), Simalungun (S9). Dan subkluster IIIB terdiri dari aksesi yang berasal dari Dairi (D13) dan Simalungun (S6 dan S7). Hasil clustering menunjukkan bahwa keragaman genetik dari andaliman pada 3 kabupaten tersebut adalah tinggi.
Hasil klustering 3 aksesi andaliman, dapat dilihat bahwa pada kelompok kedua, yang terdiri dari subkluster IIA dan IIB merupakan aksesi yang berasal dari 1 kabupaten yaitu kabupaten Dairi . Pada kelompok kedua ini tidak terdapat aksesi yang berasal dari kabupaten lain , meskipun aksesi dari kabupaten dairi lainnya menyebar pada kelompok yang lainnya.
kemerahan dan merah. Setiap jenis andaliman yang berbeda berwarna daun sama menyebar pada ketiga kelompok tersebut. Hal ini menunjukkan bahwa perbedaan secara morfologi tidak menentukan bahwa perbedaan tersebut dipengaruhi oleh faktor genetik.
Menurut Sitanggang dan Habeahan (1999) ada tiga jenis andaliman yang terdapat di kawasan Danau Toba yaitu : Sihorbo (buah besar, kurang aromatis dan produksi rendah ), Simanuk ( buah kecil, aroma dan rasa lebih tajam dari Sihorbu) dan jenis Sitanga (aroma sangat tajam sehingga lebih mirip bau kepinding, produksi tinggi namun kurang disenangi masyarakat). Ketiga jenis andaliman ini merupakan pengelompokkan masyarakat secara morfologi dan pengamatan visual yang tidak terukur. Dari hasil filogenetik andaliman dengan marka RAPD ini juga dapat dilihat bahwa andaliman dapat dikelompokkan menjadi 3 kelompok besar , namun tidak dapat dihubungkan dengan pengelompokkan secara morfologi secara spesifik untuk setiap sampel aksesi andaliman tersebut.
Menurut Zulhalmi (2013) informasi keragaman genetik tanaman pada tingkat individu , spesies maupun populasi perlu diketahui sebagai dasar pertimbangan dalam menyusun strategi konservasi, pemuliaan , pengelolaan, dan pemanfaatan sumber daya genetik tanaman secara berkelanjutan. Dari filogenetik dari 25 aksesi andaliman dapat dilihat bahwa andaliman memiliki tingkat keanekaragaman yang tinggi , dimana menurut Hutami (2006) Keragaman genetik ini dapat dimanfaatkan untuk merakit varietas unggul.
Gambar 8. Profil radial neighbor-joining 25 aksesi andaliman dari 3 kabupaten (Dairi, Tanah karo, dan Simalungun) yang dianalisis berdasarkan
Matrix Dissimilarity Simple Matching.
IA
IB
I
IIIA
III
IIIB
IIB
II
Hasil skoring pada setiap fragmen pita DNA yang terbentuk setelah amplifikasi dapat diketahui kekerabatan setiap aksesi andaliman dari 3 kabupaten tersebut. Nilai bootstrap pada 1000 mereplikasi untuk mengetahui tingkat kekerabatan pengelompokkan ditunjukkan pada (Gambar 9).
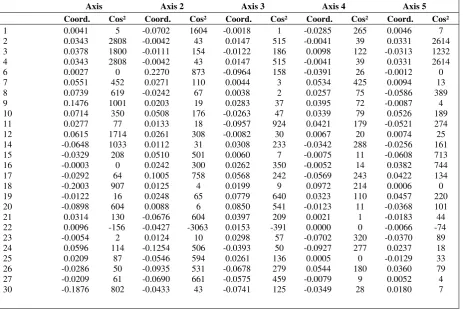
Gambar 9. Faktorial analisis (Principal Coordinate Analysis ) aksis I (horizontal)Dan aksis 2 (vertikal) dengan 5 primer marka RAPD.
Gambar 9, menunjukkan perbedaan faktorial analisis (PCoA) diantara 3 kelompok tercermin dari perbedaan pada hasil aksis 1 dan 2 (Tabel 7) yang mampu menjelaskan total 46.82% . dua puluh enam aksesi tanaman andaliman tersebar pada kuadran yang berbeda .
Faktorial analisis (Principal Coordinates Analysis) aksis 1 (horizontal)
adalah 25.52% dan aksis 2 (vertikal) adalah 21,3% dengan 5 primer marka RAPD. Dari hasil tersebut dapat diketahui bahwa 25 aksesi andaliman tersebut menyebar pada beberapa daerah pada keempat zona tersebut . Hal ini menunjukkan bahwa aksesi andaliman tersebut memiliki keragaman genetik yang tinggi , setiap aksesi tidak mengelompok pada satu sisi.
Keragaman yang tinggi di dalam populasi memberikan dasar yang luas untuk program pengembangan. Dasar untuk seleksi dalam proses ini sama seperti konservasi ex-situ tetapi lebih difokuskan pada tingkat tertinggi dari heterozigositas. Untuk menghasilkan program seleksi yang efektif, seleksi dengan individu yang lebih banyak dilakukan di dalam populasi sehingga variasi genetik yang tinggi dapat dijaga (Lim et al., 2002).
KESIMPULAN DAN SARAN
Kesimpulan
Dari 5 primer yang digunakan dalam mengamplifikasi DNA aksesi andaliman persentase polimorfik primer tersebut adalah 83,33%. Ukuran basa fragmen kelima primer tersebut adalah 320 – 2540 bp. Untuk 26 aksesi andaliman dari tiga kabupaten tersebut menunjukkan keragaman molekuler yang tinggi, dengan nilai faktorial analisis (PCoA) yaitu 46,82%. Dari bootstrap 1000 yang digunakan dalam penentuan filogenetik andaliman tersebut dapat diketahui bahwa aksesi andaliman tersebut membentuk 3 pengelompokkan secara genetik dan memilki keragaman genetik yang tinggi.
Saran
TINJAUAN PUSTAKA
Botani Tanaman
Hsuang Keng (1978) dalam Wijaya (1999) menyatakan bahwa sistematika tanaman andaliman adalah sebagai berikut: Kingdom : Plantae , Divisio: Spermatophyta , Subdivisio : Angiospermae , Klass : Dicotyledonae ,Sub klass : Rosidae, Ordo : Rutales , Family : Rutaceae , Genus : Zanthoxylum, Spesies :
Zanthoxylum acanthopodium DC.
Sistem perakaran (radix) tanaman antarasa adalah sistem akar tunggang, karena akar lembaga tumbuh terus menjadi akar pokok yang bercang-cabang menjadi akar-akar yang lebih kecil lagi. Akar pokok yang berasal dari akar lembaga disebut Radix primana (Mulia, 2000).
Andaliman (zanthoxylum acanthopodium DC) tumbuh sebagai pohon
berbatang kuas, bukan merambat. Batang-batangnya berdahan banyak, daun-daunya kecil , mirip seperti bunga mawar . Di sekujur batang , ranting, dari bawah ke bawah ujung dipenuhi duri-duri yang tajam, seperti duri mawar. Namun duri andaliman lebih besar dan kokoh . tinggi pohon rata-rata 2-4 meter , jarak lebih dari 5 meter. Usia produktif kurang dari 7 tahun. Buah andaliman muncul dari antara duri-duri itu lazimnya diapit dari duri-duri , buah tumbuh dari duri (Simanjuntak, 2006).
Andaliman (Zanthoxylum acanthopodium DC) merupakan tanaman yang
tanaman yang mempunyai tingkat keanekaragaman yang cukup tinggi, terbukti dengan jumlah spesiesnya yang mencapai 250 spesies sebagian besar terbesar di benua Amerika, sedangkan di Asia banyak ditemukan di daratan China dan india . daun andaliman tersebar, bertangkai dan merupakan daun majemuk menyirip beranak daun gasal , dengan panjang berkisar 5-20 cm dan lebar 3-15 cm, terdapat kelenjar minyak (Sabri, et al, 2008).
Gambar 1.Bagian Tanaman Andaliman (A) Pohon Andaliman, (B) Daun Andaliman, (C) Buah Andaliaman
Bunga andaliman adalah bunga majemuk terbatas yang terletak dibagian ketiak daun, ukuran bunga kecil dengan dasar bunga rata atau berbentuk kerucut. Kelopak bunga 5-7, panjang 1-2 cm, berwarna kuning pucat dan berkelamin dua . buah berbentuk kotak sejati atau kapsul , diameter 2-5 mm, pada saat muda berwarna hijau sedangkan pada saat tua berwarna hitam mengkilap, mempunyai kulit yang keras dan setiap buahnya mempunyai biji saru (Siregar, 2003) .
jika kering akan berwarna hitam. Buah andaliman yang baru dipetik sebaiknya dibungkus dengan daun pisang sebab kalau dibiarkan terbuka, akan cepat rusak dan buahnya langsung berubah hitam, dan pecah-pecah, dan bijinya akan keluar dari kulit. Oleh karena itu, untuk mendapatkan satu kilogram andaliman sangat sulit. Memanen andaliman buah perdana biasanya lebih mudah karena tangkainya lebih panjang-panjang sehingga lebih mudah memetik tetapi karena durinya masih runcing pemetikan buah sebaiknya dilakukan lebih hati-hati
(Winarno, et al, 2008 ).
Kulit luar biji (semen) antarasa yang tipis merupakan pelindung utama pada bagian biji yang ada di dalam. Warna lapisan kulit luarnya coklat kehitam-hitaman, sedangkan lapisan kulit dalamnya biasanya tipis.(Mulia, 2000)
Syarat Tumbuh
Andaliman banyak tumbuh di tanah kering di dataran tinggi dan rendah. Tumbuhan yang hidup subur di atas 1.200 m dpl itu mempunyai sifat antibakteri
Salmonella typhy, Shigella disentriae, dan Escherichia coli. Andaliman adalah
sumbernya senyawa polifenolat, monoterpen dan seskuiterpen, serta kuinon. Selain itu dalam andaliman juga terdapat kandungan minyak atsiri seperti geraniol, linalool, cineol, dan citronellal yang menimbulkan kombinasi bau mint dan lemon (Simangunsong, 2008).
Di Indonesia, tumbuhan ini tumbuh liar di pegunungan dengan ketinggian
1400 m dpl pada temperatur 15-180 C. Asal tumbuhan ini dari daerah Himalaya
Subtropis. Di dunia, tumbuhan ini tersebar antara lain di India Utara, Nepal, Pakistan
Timur, Myanmar, Thailand, dan Cina. Di Cina, tumbuhan ini tumbuh pada ketinggian
Keragaman Genetik
Keragaman genetik dalam suatu populasi tanaman sangat penting, agar seleksi dengan maksud untuk mendapatkan karakter-karakter unggul dapat dilakukan. Makin tinggi keragaman genetik maka peluang untuk mendapatkan genotipe unggul semakin besar (Greech and Reich, 1971), dan menunjukkan besarnya pengaruh genetik terhadap sifat yang diekspresikan (Knight, 1979). Jika keragaman genetik suatu tanaman sangat sempit sehingga seleksi sulit dilakukan maka, salah satu cara untuk meningkatkan keragaman genetik adalah melalui mutasi. Mutasi adalah terjadinya perubahan materi genetik pada tingkat genom, kromosom, DNA atau gen sehingga mengakibatkan terjadinya keragaman genetik (Soeranto, 2003). Dalam bidang pemuliaan tanaman, teknik mutasi dapat meningkatkan keragaman genetik sehingga memungkinkan pemulia melakukan seleksi genotipe tanaman sesuai dengan tujuan pemuliaan yang dikehendaki. (Pandin, 2010).
Keragaman genetik yang tinggi merupakan salah satu faktor penting untuk merakit varietas unggul baru. Peningkatan keragaman genetik dapat dilakukan dengan memanfaatkan plasma nutfah yang tersedia di alam dan dapat pula dengan melakukan persilangan. Sifat-sifat tertentu sering tidak ditemukan pada sumber gen yang ada sehingga teknologi lainnya perlu diterapkan (Hutami et al, 2005).
dalam kegiatan seleksi . Program pemuliaan jangka panjang yang memanfaatkan plasma nutfah untuk memperbaiki sifat-sifat agronomi dari aksesi/jenis terpilih harus didasarkan pada perkiraan determinasi genetik yang lebih akurat, sehingga penentuan individu tanaman sebagai bahan dalam perbaikan genetik dapat dilakukan dengan tepat (Rahayu dan Handayani, 2010).
Keragaman yang tinggi didalam populasi memberikan dasar yang luas untuk program pengembangan. Dasar untuk seleksi dalam proses ini sama seperti konservasi ex-situ tetapi lebih difokuskan pada tingkat tertinggi dari heterozigositas. Untuk menghasilkan program seleksi yang efektif, seleksi dengan individu yang jumlahnya lebih banyak dilakukan di dalam populasi sehingga variasi genetik yang tinggi dapat dijaga (Lim et al., 2002).
Dalam pemuliaan tanaman, keragaman genetik dalam populasi tanaman mempunyai arti yang sangat penting (Mangoendidjojo, 2003) untuk pengembangan sumber genetik yang diperlukan dalam pemuliaan tanaman (Karsinah et al., 2002). Tingkat keragaman individu dalam populasi
menggambarkan status keberadaan spesies tersebut di alam. Populasi dengan keragaman genetik yang tinggi mempunyai peluang hidup yang lebih baik karena mempunyai kemampuan yang lebih baik untuk beradaptasi dengan lingkungannya.
yang tinggi pada populasinya akan memiliki lebih banyak variasi alel yang dapat diseleksi (Elfrod dan Stansfield, 2007).
PCR (Polymerase Chain Reaction)
Reaksi Berantai Polimerase (Polymerase Chain Reaction / PCR) adalah
metode amplifikasi suatu sekuen DNA tertentu. PCR merupakan cara yang sensitif, selektif, dan sangat cepat untuk memperbanyak sekuen DNA yang diinginkan (Murray et al., 2009).
Empat komponen utama pada proses PCR adalah (1) DNA cetakan, yaitu fragmen DNA yang akan dilipatgandakan, (2) Oligonukleotida primer, yaitu suatu sekuen oligonukleotida pendek (15 – 25 basa nukleotida) yang digunakan untuk mengawali sintesis rantai DNA. Primer yang digunakan dalam PCR ada dua yaitu oligonukleotida yang mempunyai sekuen yang identik dengan salah satu rantai DNA cetakan pada ujung 5’ – fosfat dan oligonukleotida yang identik dengan sekuen pada ujung 3’ – OH rantai DNA cetakan yang lain, (3) Deoksiribonukleotida trifosfat (dNTP), yang terdiri atas dATP, dCTP, dGTP, dTTP, dan (4) Enzim DNA polimerase yaitu enzim yang berfungsi sebagai katalis dalam reaksi sintesis rantai DNA. Komponen lainnya yang juga berperan penting adalah senyawa buffer (Yuwono, 2006).
Primer biasanya terdiri dari 10-20 nukleotida dan dirancang berdasarkan
hibridisasi primer harus dikontrol dengan hati-hati agar dapat diperoleh pita-pita DNA yang utuh dan baik (Suryanto, 2003)..
Keunggulan PCR yaitu (1) Polimerase – DNA dapat diarahkan untuk sintesis wilayah DNA tertentu. Teknik PCR sebenarnya mengeksploitasi berbagai sifat alami replikasi DNA. Dalam proses tersebut, polimerase – DNA menggunakan DNA berserat tunggal sebagai cetakan untuk mensintesis serat baru yang komplementer. Cetakan berserat tunggal dapat diperoleh dengan mudah dilaboratorium melalui pemanasan DNA berserat ganda pendek untuk memulai
(prime) proses sintesis. Posisi awal dan akhir sintesis DNA pada PCR dapat
ditentukan dengan menyediakan suatu oligonukleotida sebagai primer yang menempel secara komplementer pada cetakan sesuai dengan keinginan peneliti dan (2) PCR menghasilkan amplifikasi wilayah DNA tertentu. Serat DNA dapat berfungsi sebagai cetakan untuk mensintesis bila primer oligonukleotida disediakan untuk masing – masing serat. Sepasang primer dapat dipilih yang membatasi “flanking” wilayah dari DNA yang ingin diperbanyak sehingga serat
DNA yang baru disintesis dimulai dari posisi primer, membentang sampai melewati posisi primer dari serat lainnya (Mahardika, 2003).
Random Amplified Polymorphic DNA (RAPD)
relatif tidak terbatas jumlahnya,bahan-bahan yang digunakan relatif lebih murah, preparasi lebih mudah,dan memberikan hasil lebih cepat dibandingkan dengan analisis molekular lainnya (Weising et al., 1995). Metode RAPD mampu mendetekasi sekuen nukleotida dengan hanya menggunakan satu primer. Primer tersebut akan berikatan dengan utas tunggal genom yang satu dan pada utas DNA pasangannya dengan arah berlawanan. Selama situs penempelan primer masih berada pada jarak yang dapat diamplifikasi pada umumnya tidak lebih dari 5000 pasangan basa (pb), maka akan diperoleh produk DNA amplifikasi (Weising et al., 1995). Polimorfisme RAPD merupakan hasil dari perbedaan panjang DNA hasil amplifikasi (Powell et al., 1996)
Teknik RAPD (Random Amplified Polymorphic DNA) merupakan salah
satu dari beberapa teknik pembuatan penanda berbasis DNA dengan melibatkan penggunaan mesin PCR (Polymerase Chain Reaction). Teknik PCR-RAPD dapat
digunakan untuk mengidentifikasi perbedaan genotip normal dan abnormal, berdasarkan perbedaan pada pita DNA yang dapat teramplifikasi dengan random primer. Pita DNA yang berbeda akan dianalisis lebih lanjut untuk mengetahui perbedaan urutan basa DNA antara genotip normal dan abnormal (Azizah,2009).
Penggunaan penanda RAPD relatif sederhana dan mudah dalam hal preparasi. Teknik RAPD memberikan hasil yang lebih cepat dibandingkan dengan teknik molekuler lainnya. Teknik ini juga mampu menghasilkan jumlah karakter yang relatif tidak terbatas, sehingga sangat membantu untuk keperluan analisis keanekaragaman organisme yang tidak diketahui latar belakang genomnya. Pada tanaman tahunan RAPD dapat digunakan untuk meningkatkan efisiensi seleksi awal. Teknik RAPD sering digunakan untuk membedakan organisme tingkat tinggi (eucaryote). Namun demikian beberapa peneliti menggunakan teknik ini
untuk membedakan organisme tingkat rendah (procaryote) atau melihat
perbedaan organisme tingkat rendah melalui piranti organel sel seperti mitokondria (Suryanto, 2003).
Dalam program pemuliaan tanaman, diperlukan identifikasi baik karakter morfologi maupun molekuler untuk menguji keragaman genotip klon-klon yang akan dipilih untuk tetua persilangan. Pemakaian teknik RAPD memiliki resolusi yang sebanding dengan RFLP dalam hal analisis kekerabatan antar genotif dan mampu menghasilkan jumlah karakter yang tidak terbatas sehingga sangat membantu dalam analisis keragaman genetik tanaman yang tidak diketahui latar belakang genomnya. Analisis RAPD hanya memerlukan sejumlah kecil DNA sehingga sangat sesuai untuk species tanaman berkayu. RAPD memerlukan biaya lebih rendah dibandingkan biaya untuk uji kekerabatan berdasarkan analisis DNA yang lain. Metode RAPD menggunakan primer dengan ukuran sepuluh basa sering digunakan untuk studi kekerabatan, identifikasi varietas, pemetaan genetik, analisis struktur DNA organisme dan finger printing suatu individu organisme.
digunakan sebagai penanda molekuler untuk berbagai karakter agronomis penting. Pemakaian marka molekuler RAPD banyak digunakan untuk menyusun kekerabatan beberapa individu dalam spesies maupun kekerabatan antar spesies. Penggunaan kekerabatan ini dapat dijadikan rujukan dalam pemuliaan persilangan untuk mendapatkan keragaman yang tinggi dari hasil suatu persilangan penanda RAPD yang efektif dalam mengevaluasi silsilah bahan, sementara SSR sangat penting untuk mengenali perbedaan antara karakteristik kuantitatif
(Maftuchah, 2001).
PENDAHULUAN
Latar Belakang
Andaliman merupakan rempah liar yang tumbuh di kawasan Danau Toba Provinsi Sumatera Utara. Andaliman banyak digunakan masyarakat suku Batak Toba sebagai bumbu masakan yang khas Sumatera Utara , seperti naniarsik, naniura, natinombur. Umumnya masakan-masakan khas Sumatera Utara yang menggunakan andaliman memilki daya simpan yang cukup lama. Daya awet andaliman diduga karena adanya antimikroba yang terkandung di dalamnya (Parhusip et al., 2009).
Saat ini andaliman diperhitungkan menjadi sumber senyawa aromatik dan minyak esensial. Buahnya mengandung senyawa aromatik dengan rasa pedas dan getir yang khas. Jika dimakan meninggalkan efek menggetarkan alat pengecap dan menyebabkan lidah terasa kebal. Beberapa penelitian membuktikan bahwa kandungan terpenoidnya mempunyai aktivitas antioksidan dan antimikrob (Tarigan 1999, Wijaya 1999), juga mempunyai efek imunostimulan (Wijaya 1999). Hal ini memberi peluang bagi andaliman sebagai bahan baku senyawa antioksidan atau antimikrob bagi industri pangan dan industri farmasi
(Siregar, 2003).
insektisida untuk menghambat pertumbuhan Sitophilus zeamais (Andayanie,
2000). Senyawa metabolit yang terdapat pada tanaman andaliman hanya bisa dimanfaatkan jika dilakukan pengekstraksian bagian dari tanaman tersebut. Pemanfaatan metabolit secara langsung dari tanamannya dibutuhkan banyak biomassa atau bagian tanaman sehingga mengganggu kelangsungan hidup dan keberadaan tanaman ini. Hingga kini usaha budidaya tanaman ini sulit dilakukan. Pada umumnya penyebaran tanaman ini dilakukan oleh burung, hal ini terbukti tidak ada ditemukannya anakan andaliman di sekitar tanaman induknya
(Siregar, 2003).
Populasi andaliman masih sangat terbatas, kira-kira 1000-2000 pohon, dengan produksi 7-10 kg per pohon/tahun pada tanaman dewasa. Bibit yang diperoleh petani berasal dari hutan, karena benih andaliman tidak mau berkecambah walaupun kondisi tempat tumbuhnya sudah optimal . dibudidayakan dengan sistem pekarangan. Rata-rata petani yang menanam andaliman 1-5 batang (Napitupulu, et al, 2004).
Keanekaragaman genetik dapat terjadi karena adanya perubahan nukleotida penyusun DNA. Perubahan ini mungkin dapat mempengaruhi fenotipe suatu organisme yang dapat dilihat secara langsung atau mempengaruhi reaksi individu terhadap lingkungan tertentu. Secara umum keanekaragaman genetik dari suatu populasi dapat terjadi karena adanya mutasi, rekombinasi, atau migrasi gen dari satu tempat ke tempat lain (Suryanto, 2003).
Penanda molekuler banyak digunakan dalam analisis keragaman genetik tumbuhan, salah satunya adalah Random Amplified Polymorphic DNA (RAPD).
kelebihan dalam pelaksanaan dan analisisnya. Dibandingkan dengan penanda DNA yang lain, seperti Restriction Fragment Length Polymorphisms (RFLP) dan
Simple Sequence Repeats (SSR). Teknik RAPD lebih murah, mudah dilakukan,
cepat memberikan hasil, menghasilkan polimorfisme pita DNA dalam jumlah banyak, tidak memerlukan pengetahuan tentang latar belakang genom yang dianalisis dan mudah memperoleh primer acak yang diperlukan untuk menganalisis genom semua jenis organisme. Walaupun metode ini kurang sempurna dan memiliki kelemahan dalam konsistensi produk amplifikasi (Jones et
al., 1997), tetapi kelemahan ini dapat diatasi dengan mengoptimalkan ekstraksi,
kondisi PCR dan pemilihan primer yang tepat.
Teknik RAPD (Random Amplified Polymorphic DNA) merupakan salah
satu dari beberapa teknik pembuatan penanda berbasis DNA dengan melibatkan penggunaan mesin PCR (Polymerase Chain Reaction). Teknik PCR-RAPD dapat
digunakan untuk mengidentifikasi perbedaan genotip normal dan abnormal, berdasarkan perbedaan pada pita DNA yang dapat teramplifikasi dengan random primer. Pita DNA yang berbeda akan dianalisis lebih lanjut untuk mengetahui perbedaan urutan basa DNA antara genotip normal dan abnormal (Azizah, 2009).
Andaliman merupakan rempah yang sangat bermanfaat bagi kesehatan manusia maupun sebagai bahan pengawet makanan dan minuman. Namun pada saat ini budidaya andaliman sangat sedikit dilakukan, disamping tempat tumbuh yang terbatas. Untuk mengembangkan tanaman ini maka diperlukan suatu informasi penting tentang tanaman tersebut . oleh karena itu penulis tertarik untuk mengetahui keanekaragaman genetik dari tanaman andaliman Sumatera Utara dengan menggunakan teknik Random Amplified Polymorphic DNA (RAPD), sebagai langkah awal dari pemuliaan tanaman.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengetahui pola pita DNA pada andaliman Sumatera Utara berdasarkan marka Random Amplifield Polymorphic DNA (RAPD) dengan primer OPD-13, OPI-20, OPH-09, OPM-01, OPN-10.
Manfaat Penelitian
Manfaat yang diperoleh dengan mengidentifikasi keragaman genetik andaliman tersebut antara lain :
1. Tersedianya informasi mengenai keragaman genetik andaliman di Sumatera Utara
ABSTRAK
INDRIANI MAYA SARI SEMBIRNG : Aplikasi Penanda 5 Primer RAPD
(Random Amplifield Polymorphic DNA) Untuk Analisis Andaliman (Zanthoxylum
acanthopodium DC.) Sumatera Utara, dibimbing oleh Lolie Agustina P.Putri dan
Hot Setiado.
Penelitian ini bertujuan untuk mengetahui pola pita DNA pada andaliman Sumatera Utara berdasarkan marka Random Amplified Polymorphic DNA dengan primer OPD-13, OPI-20, OPH-09, OPN-10, OPM-01. Penelitian ini telah dilakukan di Laboratorium Terpadu Fakultas Kedokteran USU pada April – September 2015. Populasi yang diamati meliputi 30 aksesi tanaman andaliman yang berasal dari Sumatera Utara terdiri dari 3 lokasi yaitu Kabupaten Dairi, Kabupaten Karo, Kabupaten Simalungun. Perhitungan dan analisis deskripstif dari penelititan ini menggunakan software DARwin 5.05. Hasil penelitian menunjukkan bahwa dari 5 primer yang digunakan dalam penelitian ini telah diperoleh total 25 pola pita DNA. Ukuran pita DNA yang dihasilkan bervariasi berkisar antara 320 bp sampai 2540 bp. Persentasi pita yang polimorfik bervariasi berkisar antara 75 % sampai 100 %. Dari 30 aksesi yang dianalis, 26 aksesi yang bias diproses oleh software, karena ada beberapa aksesi yang tidak teramplifikasi sehingga tidak memenuhi persentasi yang distandarkan. Analisis kluster dari 26 aksesi andaliman asal Sumatera Utara menunjukkan tingkat keragaman genetik yang tinggi dan memiliki hubungan kekerabatan yang dekat dengan membentuk 3 kluster utama dan 6 subkluster. Sebagian mengelompok berdasarkan populasinya dan sebagian lainnya mengelompok secara acak. Aksesi yang berasal dari Dairi menyebar pada 3 kluster, aksesi yang berasal dari Tanah Karo mengelompok sendiri sedangkan aksesi yang berasal dari Simalungun menyebar pada 2 kluster.
ABSTRACT
INDRIANI MAYA SARI SEMBIRING: 5 Primary Applications RAPD Markers
(Random Amplifield Polymorphic DNA) For Andaliman Analysis (Zanthoxylum
acanthopodium DC.) North Sumatra, supervised by Lolie Agustina P.Putri and
Hot Setiado.
The purpose of the research was to find out genetic diversity of Andaliman from North Sumatera based on Random Amplified Polymorphic DNA markers with primer OPD-13, OPI-20, OPH-09, OPN-10, OPM-01. This research was conducted at Integrated Laboratory of Faculty of Medicine , University of Suamtera Utara April-September 2015. The population observed 30 andaliman plant accessions were from North Sumatra consists of three locations: Dairi, Karo, Simalungun. DARWIN 5:05 software was used to calculate and describe the results. The results showed that the amplification of 25 accessions of andaliman using 5 random primer obtanined a total of 25 DNA band numbers. The size of DNA bands were varied ranging from 320 bp to 2540 bp. The percentage of polymorphic bands were also varied between, from 75% to 100%. From 30 of 26 accessions were processed by software due to miss-umplified as well as standard of percentage was excluded. Cluster analysis from 26 accession of andaliman from North Sumatera showed high genetic diversity and had near genetic relationship among them to form three main clusters and 6 subkluster. Some of them were grouped based on their population and some others were randomly. Accession from Dairi spread into 3 clusters, accession from Tanah Karo cluster itself while the accessions originating from Simalungun extended in 2 clusters.
acanthopodium DC.) SUMATERA UTARA
S K R I P S I
OLEH :
INDRIANI MAYA SARI SEMBIRING 110301240 / PEMULIAAN TANAMAN
PROGRAM STUDI AGROTEKNOLOGI
FAKULTAS PERTANIAN
UNIVERSITAS SUMATERA UTARA
APLIKASI PENANDA RAPD (Random amplifield polymorphicDNA) UNTUK ANALISIS KERAGAMAN GENETIK ANDALIMAN
(Zanthoxylum acanthopodium DC.) SUMATERA UTARA
S K R I P S I
OLEH :
INDRIANI MAYA SARI SEMBIRING 110301240 / PEMULIAAN TANAMAN
Skripsi Sebagai Salah Satu Syarat Untuk Mendapatkan Gelar Sarjana Pertanian Di Fakultas Pertanian
Universitas Sumatera Utara, Medan
PROGRAM STUDI AGROTEKNOLOGI
FAKULTAS PERTANIAN
UNIVERSITAS SUMATERA UTARA
Judul Penelitian : Analisis Penanda RAPD (Random Amplifield Polymorphic DNA )
Untuk Analisis Keragaman Genetik Andaliman (Zanthoxylum
acanthopodium DC.) Sumatera Utara
Nama : Indriani Maya Sari Sembiring NIM : 110301240
Program Studi : Agroekoteknologi Minat Studi : Pemuliaan Tanaman
Disetujui oleh : Komisi Pembimbing
(Dr. Ir. Lollie Agustina P.Putri, MSi) (Ir. Hot Setiado, MS
Ketua Anggota
ABSTRAK
INDRIANI MAYA SARI SEMBIRNG : Aplikasi Penanda 5 Primer RAPD
(Random Amplifield Polymorphic DNA) Untuk Analisis Andaliman (Zanthoxylum
acanthopodium DC.) Sumatera Utara, dibimbing oleh Lolie Agustina P.Putri dan
Hot Setiado.
Penelitian ini bertujuan untuk mengetahui pola pita DNA pada andaliman Sumatera Utara berdasarkan marka Random Amplified Polymorphic DNA dengan primer OPD-13, OPI-20, OPH-09, OPN-10, OPM-01. Penelitian ini telah dilakukan di Laboratorium Terpadu Fakultas Kedokteran USU pada April – September 2015. Populasi yang diamati meliputi 30 aksesi tanaman andaliman yang berasal dari Sumatera Utara terdiri dari 3 lokasi yaitu Kabupaten Dairi, Kabupaten Karo, Kabupaten Simalungun. Perhitungan dan analisis deskripstif dari penelititan ini menggunakan software DARwin 5.05. Hasil penelitian menunjukkan bahwa dari 5 primer yang digunakan dalam penelitian ini telah diperoleh total 25 pola pita DNA. Ukuran pita DNA yang dihasilkan bervariasi berkisar antara 320 bp sampai 2540 bp. Persentasi pita yang polimorfik bervariasi berkisar antara 75 % sampai 100 %. Dari 30 aksesi yang dianalis, 26 aksesi yang bias diproses oleh software, karena ada beberapa aksesi yang tidak teramplifikasi sehingga tidak memenuhi persentasi yang distandarkan. Analisis kluster dari 26 aksesi andaliman asal Sumatera Utara menunjukkan tingkat keragaman genetik yang tinggi dan memiliki hubungan kekerabatan yang dekat dengan membentuk 3 kluster utama dan 6 subkluster. Sebagian mengelompok berdasarkan populasinya dan sebagian lainnya mengelompok secara acak. Aksesi yang berasal dari Dairi menyebar pada 3 kluster, aksesi yang berasal dari Tanah Karo mengelompok sendiri sedangkan aksesi yang berasal dari Simalungun menyebar pada 2 kluster.
ABSTRACT
INDRIANI MAYA SARI SEMBIRING: 5 Primary Applications RAPD Markers
(Random Amplifield Polymorphic DNA) For Andaliman Analysis (Zanthoxylum
acanthopodium DC.) North Sumatra, supervised by Lolie Agustina P.Putri and
Hot Setiado.
The purpose of the research was to find out genetic diversity of Andaliman from North Sumatera based on Random Amplified Polymorphic DNA markers with primer OPD-13, OPI-20, OPH-09, OPN-10, OPM-01. This research was conducted at Integrated Laboratory of Faculty of Medicine , University of Suamtera Utara April-September 2015. The population observed 30 andaliman plant accessions were from North Sumatra consists of three locations: Dairi, Karo, Simalungun. DARWIN 5:05 software was used to calculate and describe the results. The results showed that the amplification of 25 accessions of andaliman using 5 random primer obtanined a total of 25 DNA band numbers. The size of DNA bands were varied ranging from 320 bp to 2540 bp. The percentage of polymorphic bands were also varied between, from 75% to 100%. From 30 of 26 accessions were processed by software due to miss-umplified as well as standard of percentage was excluded. Cluster analysis from 26 accession of andaliman from North Sumatera showed high genetic diversity and had near genetic relationship among them to form three main clusters and 6 subkluster. Some of them were grouped based on their population and some others were randomly. Accession from Dairi spread into 3 clusters, accession from Tanah Karo cluster itself while the accessions originating from Simalungun extended in 2 clusters.
KATA PENGANTAR
Puji dan syukur penulis ucapkan kepada Tuhan Yang Maha Esa karena atas berkat dan rahmat-Nya penulis dapat menyelesaikan skripsi ini dengan baik dan tepat pada waktunya.
Adapun judul dari skripsi ini adalah “Aplikasi Penanda 5 Primer RAPD
(Random Amplifield Polymorphic DNA) Untuk Analisis Andaliman
(Zanthoxylum acanthopodium DC.) Sumatera Utara” yang merupakan salah
satu syarat untuk mendapatkan gelar sarjana di Fakultas Pertanian Universitas Sumatera Utara, Medan
Pada kesempatan ini penulis mengucapkan terima kasih kepada Dr.Ir.LollieAgustinaP.Putri,M.Si selaku ketua pembimbing dan Ir. Hot Setiado, MS. selaku anggota pembimbing yang telah membimbing dan
memberikan berbagai masukan berharga kepada penulis dari mulai menetapkan judul, melakukan penelitian sampai dengan skripsi ini selesai, Penulis mengucapkan terimakasih yang sebesar-besarnya kepada orang tua penulis yang telah membesarkan dan mendidik penulis.
teman-teman Pemuliaan Tanaman stambuk 2011, dan IMKA Mbuah Page FP USU.
Penulis menyadari bahwa skripsiini masih jauh dari kesempurnaan, oleh karena itu penulis mengharapkan kritik dan saran yang bersifat membangun demi perbaikan skripsi di masa yang akan datang.
Medan, November 2015
DAFTAR ISI
PCR (Polymerase Chan Reaction) ... 10
RAPD ( Random Amplified Polymorphic DNA) ... 11
BAHAN DAN METODE Tempat dan Waktu Penelitian ... 15
Bahan dan Alat ... 15 Analisis Profil Pita Hasil Isolasi DNA Tanaman Andaliman ... 23
Analisis Profil Pita Hasil Amplifikasi PCR Tanaman Andaliman ... 27
Analisis Kluster Tanaman Andaliman ... 35
KESIMPULAN DAN SARAN Kesimpulan ... 41
Saran ... 41
DAFTAR PUSTAKA
DAFTAR TABEL
No. Hal
1. Siklus, amplifikasi, DNA 30 aksesi tananaman andaliman ... 19
2. Data geografis dan morfologis 30 aksesi andaliman ... 23
3. Hasil uji kuantitatif 30 aksesi andaliman ... 25
4. Hasil amplifikasi lima primer yang digunakan ... 28
5. Hasil scoring pita 30 aksesi andaliman ... 53
6. Hasil koordinat faktorial matriks jarak ketidaksamaan genetic ... 56
DAFTAR GAMBAR
No. Hal
1. Bagian tanaman andaliman ... 6
2. Elektroforegram amplifikasi DNA andaliman dengan primer OPD-13 ... 29
3. Elektroforegram amplifikasi DNA andaliman dengan primer OPI-20 ... 30
4. Elektroforegram amplifikasi DNA andaliman dengan primer OPH-09 ... 30
5. Elektroforegram amplifikasi DNA andaliman dengan primer OPM-10 ... 31
6. Elektroforegram amplifikasi DNA andaliman dengan primer OPN-10 ... 31
7. Dendogram 25 aksesi tanaman andaliman asal Sumatera Utara yang dianalisis berdasarkan matrix disimilarity simple matching ... 34
8. Profil radial Neighbour-Joining Tree (NJTree) dari 25 aksesi andaliman di Sumatera Utara yang dianalisis berdasarkan matrix disimilirity simple matching ... 35
9. Analisis factor Principal Coordinates Analysis (PCoA) aksis 1 (horizontal) dan aksis 2 (vertikal) yang dianalisis berdasarkan matrix dissimilarity simple matching ... 36
10. Alat Spektrofotometer ... 49
11. Hasil uji kuantitatif ... 49
12. Hasil elektoforesis dengan Primer OPD-13 ... 51
13. Hasil elektoforesis dengan Primer OPI-20 ... 51
14. Hasil elektoforesis dengan Primer OPH-09 ... 51
15. Hasil elektoforesis dengan Primer OPN-10 ... 52
DAFTAR LAMPIRAN
No. Hal
1. Gambar sampel aksesi andaliman ... 41
2. Pembuatan larutan stok dan buffer ... 43
3. Alur penelitian ... 45
4. Proses isolasi DNA ... 46
5. Proses PCR-RAPD ... 48
6. Foto uji kuantitas ... 49
7. Hasil uji kuantitatif 30 aksesi tanaman andaliman... 50
8. Hasil fotosintesis ... 51
9. Matriks jarak ketidaksamaan genetik pada 25 aksesi andaliman di Sumatera Utara ... 55