ISOLASI DAN AMPLIFIKASI FRAGMEN GEN COX3 PADA IKAN Ompok hypophthalmus (Bleeker 1846) DARI SUNGAI TAPUNG PROVINSI RIAU
Khairunnisa Ul Fitri1, Roza Elvyra 2, Dewi Indriyani Roslim 3 1
Mahasiswa Program S1 Biologi 2
Dosen Bidang Zoologi Jurusan Biologi 3
Dosen Bidang Genetika JurusanBiologi Fakultas Matematika dan Ilmu Pengetahuan Alam
Kampus BinaWidya Pekanbaru, 28293, Indonesia ulfitrikhairunnisa@yahoo.com
ABSTRACT
Selais danau fish (Ompok hypophthalmus) is one of the fish which inhabit the floodplain river. Genetic information on Ompok hypophthalmus is still limited. This research aims to obtain total DNA of O. hypophthalmus and DNA fragment amplified from COX3 gene. Samples of ten O. hypophthalmus, retrieved from a floodplain river, Tapung River, were isolated using the isolation Kit. Purification of DNA has been done using Dneasy Blood and Tissue Kit from Qiagen. Total DNA was amplified using COX3 F1 and COX3 R1 primers which were designed using sequence of COX3 gene from Ictalurus punctatus (Genbank 2002). The result indicated that eight samples of total DNA were successfully isolated from ten of fish samples taken. Amplification of total DNA using primer COX3 F1 and COX3 R1 produces DNA fragments which sized 671 bp.
Keywords : COX3 gene, DNA isolation, Ompok hypophthalmus, floodplain river, Tapung River
ABSTRAK
Ikan selais danau (Ompok hypophthalmus) merupakan salah satu jenis ikan sungai paparan banjir. Informasi genetik pada ikan Ompok hypophthalmus masih terbatas, untuk itu perlu dikaji informasi ilmiah berbasis genetik berdasarkan gen COX3 pada genom mitokondria. Penelitian ini bertujuan untuk memperoleh DNA total ikan selais danau dan fragmen DNA hasil amplifikasi dari gen COX3. Sepuluh sampel Ompok hypophthalmus yang diperoleh dari sungai paparan banjir yaitu Sungai Tapung, kemudian diisolasi menggunakan Kit isolasi dan purifikasi DNA Dneasy Blood and Tissue Kit dari Qiagen. Molekul DNA total yang diperoleh diamplifikasi menggunakan primer COX3 F1 dan COX3 R1 yang didesain menggunakan sekuen gen COX3 Ictalurus punctatus sebagai acuannya (Genbank 2002). Hasil penelitian menunjukkan delapan sampel DNA total berhasil diisolasi dari sepuluh sampel yang diambil. Hasil amplifikasi menggunakan primer COX3 F1 dan COX3 R1 menghasilkan fragmen DNA berukuran 671 bp.
Kata kunci : gen COX3, isolasi DNA, Ompok hypophthalmus, PCR, sungai paparan banjir
PENDAHULUAN
Provinsi Riau adalah salah satu provinsi yang memiliki ekosistem sungai paparan banjir (floodplain river) (Elvyra 2009). Sungai paparan banjir merupakan suatu ekosistem yang unik, ekosistem ini subur dan sangat dipengaruhi oleh fluktuasi curah hujan. Salah satu sungai paparan banjir yang terdapat di Provinsi Riau adalah Sungai Tapung. Sungai Tapung memiliki kedalaman 8-12 meter dengan panjang ±90 km. Sungai Tapung terbagi atas dua muara, yaitu Tapung Kanan dan Tapung Kiri (Diskanlut Provinsi 2007b). Menurut Azwir (2006) Sungai Tapung Kiri memiliki substrat berpasir, aliran dari sungai ini bermuara di Sungai Siak.
Sungai paparan banjir adalah salah satu ekosistem yang produktif (Mustakim 2008). Pada saat musim penghujan banyak kelompok ikan yang memijah dan membesarkan anak-anak ikan. Berbagai jenis ikan yang bernilai ekonomis dan bersifat endemik hidup dan berkembang biak di sungai paparan banjir. Salah satu jenis ikan yang hidup di sungai paparan banjir adalah ikan Ompok hypophthalmus dicirikan sebagai kelompok ikan bersungut (catfish) dan merupakan salah satu jenis ikan sungai paparan banjir. Ompok hypophthalmus di Provinsi Riau dikenal dengan sebutan ikan selais danau. Ikan selais danau merupakan jenis ikan yang sangat di digemari oleh masyarakat, khususnya di Provinsi Riau. Beberapa tahun belakangan produksi dari penangkapan ikan menurun. Penurunan ini antara lain disebabkan gagalnya regenerasi ikan di
ekosistem sungai akibat rusaknya habitat dari ikan tersebut. Kerusakan habitat disebabkan disekitar daerah aliran sungai telah beralih fungsi menjadi lahan perkebunan sawit dan penurunan kualitas air diakibatkan adanya sedimentasi yang cukup tinggi (Diskanlut Provinsi Riau 2007a).
Berdasarkan keberadaan ikan ini sebagai ikan sungai paparan banjir, sumber ekonomi dan sumber keanekaragaman hayati di Indonesia khususnya wilayah Provinsi Riau, maka perlu dilakukan penelitian berbasis genetik menggunakan teknik molekuler untuk mendapatkan penanda genetik dari ikan Ompok hypophthalmus dan untuk menunjang usaha pelestarian ikan Ompok hypophthalmus yang ada di sungai paparan banjir di Provinsi Riau.
Sebelum mendapatkan informasi ilmiah mengenai penanda genetik ikan, tahap awal yang dilakukan terlebih dahulu adalah dengan mengisolasi DNA total ikan dan amplifikasi wilayah genom mitokondria berdasarkan gen COX3. Penelitian ini bertujuan untuk memperoleh DNA total ikan selais danau dan fragmen DNA hasil amplifikasi dari gen COX3.
METODE PENELITIAN
Penelitian dilakukan pada bulan Oktober 2014 - April 2015. Penelitian ini dilaksanakan di Laboratorium Zoologi dan Genetika Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Riau, Pekanbaru. Sampel Ompok hypophthalmus diperoleh dari
Sungai Tapung desa Flamboyan Kabupaten Kampar, Provinsi Riau.
Alat yang digunakan adalah rak tabung mikro, pipet mikro berukuran 10 µl, 100 µl, dan 1000 µl, tip mikro, tabung 50 ml, tabung mikro berukuran 1,5 ml dan 0,2 ml, gunting, pinset, spatula, waterbath, timbangan analitik, aluminium foil, hot plate, kamera (Olympus SP-500 UZ), UV transluminator, mesin sentrifus, perangkat elektroforesis horizontal, dan mesin PCR. Bahan yang digunakan pada penelitian ini adalah Kit isolasi dan purifikasi DNA Dneasy Blood and Tissue Kit dari Qiagen yang terdiri dari: buffer ATL, AL, AW1, AW2, AE, dan Proteinase K. Selain bahan-bahan diatas dibutuhkan juga etanol absolut dan buffer TE [1 mM EDTA pH 8,0; 10 mM Tris-HCl pH 8,0; 40 mg/ml RNAse]. Amplifikasi fragmen DNA dengan PCR memerlukan TopTaq Master Mix PCR dari Qiagen yang terdiri dari: 2x TopTaq Master Mix, 10x CoralLoad concentrate, dan dH2O. Selain itu dibutuhkan juga
sepasang primer gen COX3 yaitu primer COX3-F1 5’ CCA CCA AGC ACA TGC ATA C -3’ dan primer COX3 - R1 5’CGA ATC CCA AGT GGT GTT CT -3’ yang didesain menggunakan program primer3 output dengan sekuen Ictalurus punctatus sebagai acuan gen COX3 (Genbank 2002), loading dye, dan DNA ladder. Bahan untuk pembuatan gel agarosa: 1% agarosa untuk elektroforesis DNA total dan 1,2% agarosa untuk elektroforesis hasil PCR, Buffer TBE (Tris base - Boric acid – EDTA pH 8), etidium bromida, dan akuades.
a. Pengambilan Sampel
Sepuluh sampel ikan selais danau dari Sungai Tapung yang diperoleh dari nelayan, diidentifikasi menggunakan kunci identifikasi menurut Kottelat et al. (1993). Setelah itu cuplikan otot bagian dekat ujung ekor ikan dipotong dengan ukuran 1x1 mm kemudian dimasukkan ke dalam tabung mikro yang berukuran 1,5 ml yang telah berisi etanol absolut. Tabung diberi label pada bagian luarnya, selanjutnya disimpan di dalam kulkas. b. Penanganan Sampel dari
Lapangan
Sampel otot ikan yang telah disimpan di dalam etanol absolut dikeluarkan secukupnya, dan diletakkan diatas tisu sambil dikeringanginkan agar etanolnya menguap. Sampel kemudian ditimbang seberat 20 mg lalu dimasukkan ke dalam tabung mikro 1,5 ml yang baru. Setelah itu sampel dicuci dengan larutan 1x TE sebanyak 100 µl. kemudian divorteks selama 15 detik, setelah itu disentrifus selama 2 menit pada kecepatan 3000 rpm. Larutan 1x TE dibuang dengan menggunakan pipet mikro, lakukan proses ini sebanyak 3 kali. Sampel kembali dikeringanginkan diatas tisu hingga etanol yang tersisa menguap. Proses selanjutnya adalah isolasi dan purifikasi DNA.
c. Isolasi dan Purifikasi
Proses isolasi dan purifikasi DNA total otot ikan dilakukan menggunakan Kit isolasi dan purifikasi DNA Dneasy Blood and Tissue Kit dari Qiagen. Otot ikan dicacah halus kemudian dimasukkan ke dalam tabung mikro 1,5
ml lalu ditambahkan larutan Buffer ATL sebanyak 180 µl. Sampel divorteks selama 15 detik kemudian diberi penambahan proteinase K sebanyak 20 µl lalu divorteks kembali selama 15 detik. Setelah itu, sampel diinkubasi dalam waterbath dengan suhu 56ºC selama 1-3 jam sambil sesekali divorteks selama 15 detik. Selanjutnya, buffer AL ditambahkan kedalam sampel sebanyak 200 µl kemudian divorteks selama 15 detik. Langkah selanjutnya, etanol absolut ditambahkan sebanyak 200 µl lalu divorteks selama 15 detik dan disentrifus selama 2 menit dengan kecepatan 4000 rpm. Supernatan dipindahkan ke dalam collection tube yang telah terpasang dengan spin column, kemudian sampel disentrifus selama 2 menit dengan kecepatan 400 rpm. Supernatan yang tertinggal di colletion tube dibuang, kemudian spin column dipindahkan ke dalam collection tube yang baru, kemudian ditambahkan buffer AW1 sebanyak 500 µl, setelah itu sampel disentrifus kembali selama 2 menit dengan kecepatan 4000 rpm. Buang larutan yang ada di collection tube kemudian pindahkan spin colomn ke dalam collection tube yang baru, lalu ditambahkan 500 µl buffer AW2 ke dalam sampel dan disentrifus selama 10 menit dengan kecepatan 4000 rpm. Supernatan yang tertinggal di collection tube dibuang kemudian pindahkan spin column ke dalam tabung mikro 1,5 ml yang baru, ditambahkan 200 µl buffer AE untuk mengelusi DNA yang melekat pada membran spin colomn. Selanjutnya, sampel disentrifus selama 2 menit dengan kecepatan 4000 rpm. Supernatan yang diperoleh disimpan sebagai stok DNA 1. Proses elusi diulang sebanyak 2 kali. Pindahkan spin
colomn ke tabung mikro 1,5 ml yang baru, lalu ditambahkan buffer AE sebanyak 200 µl. Sampel kembali disentrifus selama 2 menit dengan kecepatan 400 rpm (proses sentrifus diulangi sebanyak 2 kali), supernatan yang diperoleh disimpan sebagai stok DNA 2. Supernatan disimpan pada suhu 4ºC dan selanjutnya digunakan sebagai cetakan dalam proses amplifikasi wilayah gen COX3 menggunakan PCR. d. Elektroforesis Hasil Purifikasi
DNA Total
Hasil purifikasi DNA total diperiksa kualitas dan keutuhannya menggunakan mesin elektroforesis. Hasil purifikasi DNA total dielektroforesis pada 1% gel agarosa dalam larutan 1x TBE (Tris base – Boric acid – EDTA). Molekul DNA total diwarnai dengan etidium bromida (0,5 µg/ml) kemudian divisiualisasi diatas UV transluminator (WiseUv WUV-M20, Daihan Scientific) dengan pada gelombang 302 nm dan difoto dengan kamera berfilter.
e. Amplifikasi Wilayah Gen COX3 dari Mitokondria
Proses amplifikasi gen COX3 dilakukan menggunakan TopTaq Master Mix Kit PCR dari Qiagen. Adapun volume dari tiap komponen mix PCR yang dimasukkan ke dalam tabung mikro ukuran 0,2 ml antara lain 44 µl 10x CoralLoad, 132 µl dH2O, 220 µl 2x
TopTaq Master Mix. Selain itu dibutuhkan juga sepasang primer COX3-F1 5’ CCA CCA AGC ACA TGC ATA C -3’ dan primer COX3 - R1 5’CGA ATC CCA AGT GGT GTT CT -3’ yang sudah diencerkan dengan volume 8,8 µl
primer COX3 FI dan 8,8 µl primer COX3 R1 3’ yang telah didesain menggunakan program primer3 output (www.genome.wi.mit.edu) dengan sekuen Ictalurus punctatus (Genbank 2002) sebagai acuan gen COX3. Total volume PCR yang digunakan adalah 50 µl. Kondisi PCR yang digunakan mengikuti Elvyra dan Duryadi (2007) dengan sedikit modifikasi suhu pada tahapan annealing. Adapun tahapan proses PCR meliputi: pra-PCR pada suhu 94ºC selama 5 menit, diikuti dengan 35 siklus yang terdiri dari 3 tahapan yaitu: denaturasi pada suhu 94 ºC selama 1 menit, annealing dengan suhu optimasi 51,6 ºC selama 1 menit dan elongasi pada suhu 72 ºC selama 1 menit. Langkah terakhir adalah post-PCR 72 ºC selama 5 menit.
f. Elektroforesis Hasil PCR
Fragmen DNA hasil PCR dielektroforesis pada 1,2% gel agarosa dalam larutan 1x TBE (Tris base – Boric acid – EDTA) dengan menggunakan alat elektroforesis. Setelah itu gel agarosa diwarnai dengan etidium bromida (0,5 µg/ml) lalu divisiualisasi diatas UV transluminator (WiseUv WUV-M20, Daihan Scientific) dengan panjang gelombang 302 nm dan difoto dengan kamera (Olympus SP-500 UZ) berfilter. HASIL DAN PEMBAHASAN
a. Molekul DNA Total
Proses ekstraksi DNA merupakan proses untuk memisahkan DNA dari komponen sel lainnya. Hasil ekstraksi DNA O. hypophthalmus dicek kualitas DNAnya menggunakan mesin
elektroforesis. Elektroforesis merupakan suatu proses memigrasikan partikel bermuatan di bawah pengaruh medan listrik dalam kondisi yang konstan. Pada saat arus listrik diberikan, molekul akan bermigrasi melalui media (biasanya berupa gel), semakin kecil ukuran molekul maka kecepatan bermigrasinya semakin cepat begitu juga sebaliknya (Green & Sambrook 2012).
Gel yang biasanya digunakan sebagai media elektroforesis adalah agarosa. Agarosa berasal dari ekstrak rumput laut yang sudah dimurnikan dan merupakan polisakarida yang tidak bermuatan (netral). Elektroforesis gel agarosa dapat memisahkan sampel DNA yang berukuran 200-50.000 bp, sedangkan untuk memisahkan DNA dengan ukuran lebih pendek yang berkisar 5-500 bp dapat digunakan gel poliakrilamid (Green & Sambrook 2012).
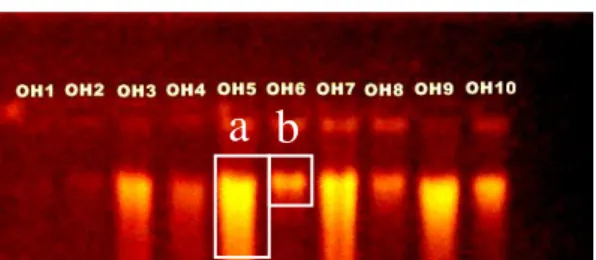
Hasil elektroferesis berupa fragmen DNA dapat divisualisasikan dengan beberapa cara, yaitu dengan penambahan etidium bromida (EtBr) ke dalam gel sewaktu pembuatannya atau dengan cara gel direndam di dalam larutan etidium bromida sebelum divisualisasikan dengan UV transluminator. Molekul etidium bromida berpendar ketika diiluminasi dengan cahaya ultraviolet (UV). Etidium bromida merupakan pewarna mutagenik yang akan mengikat dengan cara menyisip diantara ikatan basa pada untai ganda DNA sehingga menyebabkan transmisi sinar UV menjadi dapat dilihat (Fairbanks & Andersen 1999). Profil hasil eletroforesis DNA total sampel O. hypophthalmus tersaji pada Gambar 1.
Gambar 1 Profil pita DNA total ikan Ompok hypophthalmus menggunaka 1% gel agarosa. Keterangan: OH (Ompok hypophthalmus) Berdasarkan hasil elektroforesis dari sepuluh sampel O. hypophthalmus yang diisolasi, hanya delapan sampel yang berhasil diisolasi yaitu ditandai munculnya pita DNA total pada saat divisualisasi menggunakan UV transluminator. Dari delapan sampel yang berhasil diisolasi menunjukkan hasil yang bervariasi (Gambar 1). Molekul DNA total dikatakan baik apabila profil pada pita DNA menunjuk pita yang tebal, tegas dan tidak terdapat smear (b). Pita DNA yang smear (a) disebabkan karena DNA terdegradasi pada saat proses isolasi. Sampel yang pita DNA nya muncul dan bagus dipilih untuk kemudian diamplifikasi fragmen DNA mitokondrianya pada bagian gen COX3 menggunakan mesin PCR.
b. Amplifikasi Fragmen DNA Mitokondria dari Gen COX3 Amplifikasi merupakan proses perbanyak fragmen DNA secara in vitro menggunakan teknik PCR (Polymerase Chain Reaction) pada daerah yang spesifik yang dibatasi oleh sepasang primer. Salah satu faktor keberhasil dari
proses amplifikasi PCR yaitu dari pemilihan primer. Primer berperan sebagai penanda fragmen sampel DNA yang akan diamplifikasi. Primer merupakan potongan untaian DNA pendek utas tunggal (oligonukleotida) dengan panjang berkisa 10 - 40 basa (Zein & Prawiradilaga 2013). Pada kondisi tertentu primer akan mengenali dan berikatan dengan untaian DNA komplemennya yang terletak diawal dan diakhir dari fragmen DNA target.
Molekul DNA total hasil ekstraksi digunakan sebagai cetakan untuk proses amplifikasi. Pada penelitian ini digunakan sepasang primer yaitu primer COX3-F1 5’ CCA CCA AGC ACA TGC ATA C -3’ dan primer COX3-R1 5’CGA ATC CCA AGT GGT GTT CT -3’ yang telah didesain menggunakan
program primer3 output
(www.genome.wi.mit.edu) dengan sekuen Ictalurus punctatus (Genbank 2002) sebagai acuan gen COX3. Adapun tahapan PCR untuk mengamplifikasi gen COX3 antara lain : pra-PCR 94ºC selama 5 menit, denaturasi 94ºC selama 1 menit, untuk annealing, tahapan annealing merupakan tahapan yang penting pada proses PCR karena jika suhu annealing terlalu rendah maka akan terjadi penempelan primer pada template (cetakan) DNA tidak spesifik sedangkan jika suhu annealing terlalu tinggi maka penempelan primer pada template DNA akan lepas kembali sehingga produk PCR tidak terbentuk (Wahyudi 2001). Proses annealing menggunakan optimasi suhu 51,6ºC selama 1 menit, elongasi 72ºC selama 1 menit, amplifikasi dilakukan sebanyak 35 siklus kemudian diakhir dengan post-PCR 72ºC selama 5 menit. Jumlah siklus yang dilakukan pada proses PCR bervariasi tergantung
a b
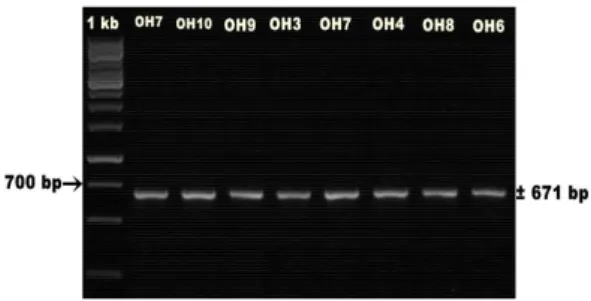
pada produk akhir PCR yang diharapkan. Siklus 23-35 biasanya sudah cukup untuk menghasilkan fragmen DNA, untuk kondisi PCR seperti jumlah siklus pada penelitian ini mengikuti Elvyra dan Duryadi (2007) dengan jumlah siklus sebanyak 35 siklus. Produk PCR selanjutnya dideteksi dengan mesin elektroforesis menggunakan gel agarosa 1,2%, kemudian divisualisasi dengan bantuan UV transluminator (WiseUv WUV-M20, Daihan Scientific) dengan panjang gelombang 302 nm dan difoto dengan kamera (Olympus SP-500 UZ) berfilter (Gambar 2).
Gambar 2 Profil fragmen gen COX3 Ompok hypophthalmus menggunakan 1,2% gel agarosa. Keterangan: OH (Ompok hypophthalmus) Dari hasil amplifikasi gen COX3 pada ikan O. hypophthalmus menggunakan primer COX3 F1 dan COX3 R1 menghasilkan fragmen gen COX3 yang berukuran ±671 bp dengan kualitas fragmen DNA yang baik.
KESIMPULAN
Molekul DNA total telah berhasil diperoleh pada penelitian ini. Hasil amplifikasi menggunakan primer COX3 F1 dan primer COX3 R1 menghasilkan
fragmen gen COX3 yang berukuran ±671 bp dengan kualitas fragmen DNA yang baik.
UCAPAN TERIMA KASIH
Penulis mengucapkan terima kasih kepada DIKTI DP2M yang telah membiayai penelitian ini melalui Hibah Kompetensi atas nama Dr. Roza Elvyra, M.Si tahun 2015.
DAFTAR PUSTAKA
Azwir. 2006. Analisa pencemaran air sungai Tapung oleh limbah industri kepala sawit PT. Peputra Materindo di kabupaten Kampar [tesis]. Semarang: Program Pasca Sarjana Ilmu Lingkungan, Universitas Diponegoro.
Dinas Perikanan dan Kelautan Provinsi Riau. 2007a. Statistik perikanan tangkap Provinsi Riau. Pekanbaru: Diskanlut Provinsi Riau.
Dinas Perikanan dan Kelautan Provinsi Riau. 2007b. Sungai Tapung. Pekanbaru: Diskanlut Provinsi Riau.
Elvyra R, Duryadi D. 2007. Kajian penanda genetik gen sitokrom b DNA Sungai Kampar Riau. J Natur Ind 10:6-12.
Elvyra R. 2009. Kajian Keragaman genetik dan biologi reproduksi ikan lais di Sungai Kampar Riau [disertasi]. Bogor: Sekolah Pascasarjana, Institut Pertania Bogor.
Fairbank DJ, Andersen WR. 1999. Genetics: The Continuity of Life. New York: Brooksdole Publishing Company.
Genbank. 2002. Ictalurus punctatus mitochondrion, complete genome. http://www.ncbi.nml.nih.gov/nucc ore/NC_003489.1. [Diakses pada 29 Maret 2015]
Green MR, Sambrook J. 2012. Molecualr Cloning: A Labolatory Manual. 4th ed. New York: Cold Spring Harbor Labolatory Press Kottelat M, Whitten AJ, Kartikasari SN,
Wirdjoatnodjo S. 1993. Freswater fishes of western Indonesia and Sulawesi. Jakarta: Periplus edition (HK) in collaboration with the environment Rep. of Indonesia.
Mustakim M. 2008. Kajian Kebiasaan Makanan dan Kaitannya Dengan Aspek Reproduksi Ikan Betok (Anabas testudineus Bloch) Pada Habitat Yang Berbeda Di Lingkungan Danau Melintang Kutai Kartanegara Kalimantan Timur [tesis].Bogor : Sekolah Pasca Sarjana. Institut Pertanian Bogor.
Wahyudi TH. 2001. Pengaruh suhu annealing dan jumlah siklus yang berbeda pada program PCR terhadap keberhasilan isolasi dan amplifikasi mtDNA ikan Patin (Pangasius hypophthalmus) [skripsi]. Bogor: Fakultas Perikanan dan Ilmu kelautan, Intitut Pertanian Bogor.
Zein MSA, Prawiradilaga DM. 2013.DNA Barcode Fauna Indonesia. Jakarta: Kencana.