LAMPIRAN
Lampiran 1. Profil elektroforegram perbandingan primer SSR-3574
Keterangan: M = DNA ladder 1kb (Marker), A = isolasi DNA berasal dari bagian akar, D = isolasi DNA berasal dari bagian daun, B = isolasi DNA berasal dari bagian
buah
Lampiran 2. Profil elektroforesis perbandingan primer SSR-3574
Keterangan: M = DNA ladder 1kb (Marker), D = DNA stok berasal dari bagian daun, B = DNA stok berasal dari bagian buah P = DNA stok berasal dari bagian pollen,
O = DNA stok berasal dari bagian bunga betina
33D 33A 34D 34A 35D 35A 36D 36A 15D 15B 100bp 16D 16B 17D 17B 18D 18B 6D 6B 7D
Lampiran 3. Profil elektroforegram pengulangan proses PCR dengan Primer Beta pada sampel bagian akar
Keterangan: M = DNA ladder 1kb (Marker), A = isolasi DNA berasal dari bagian akar, B = isolasi DNA berasal dari bagian buah
Lampiran 4. Profil elektroforegram yang dianalisis menggunakan UVITEC FIREREADER
Lampiran 5. Profil elektroforegram yang dianalisis menggunakan UVITEC FIREREADER
Lampiran 6. Pembuatan larutan stok dan buffer
A. Pembuatan Larutan Stok
• CTAB 5 % (100 ml): Timbang NaCl sebanyak 2.0 gram dan CTAB
sebanyak 5.0 gram. Masukkan bahan kimia ke dalam erlenmeyer dan
ditambahkan 100 ml aquades. Aduk campuran larutan dengan menggunakan
stirrer kemudian diletakkan diatas hotplate.
• Tris HCl 1 M pH 8.0 (100 ml): Timbang Tris sebanyak 12.114 gram.
Masukkan Tris ke dalam erlenmeyer dan ditambahkan 80 ml aquades. Aduk
campuran larutan dengan menggunakan stirrer kemudian diletakkan diatas
hotplate. Selanjutnya, ditambahkan 4.2 ml HCl pekat sedikit demi sedikit
sampai pH mencapai 8. Masukkan ke dalam gelas ukur, kemudian volume
ditepatkan dengan aquades hingga volume larutan menjadi 100 ml.
• Tris HCl 1 M pH 7.4 (50 m): Timbang Tris sebanyak 6.057 gram.
campuran larutan dengan menggunakan stirrer kemudian diletakkan diatas
hotplate. Selanjutnya, ditambahkan NaOH 2.5 M sedikit demi sedikit sampai
pH mencapai 7.4. Masukkan ke dalam gelas ukur, kemudian volume
ditepatkan dengan aquades hingga volume larutan menjadi 50 ml.
• EDTA 0.5 M pH 8.0 (100 ml) : Timbang EDTA sebanyak 18.612 gram dan
NaOH 2.0 gram. Masukkan bahan kimia ke dalam erlenmeyer dan
ditambahkan 80 ml aquades. Aduk campuran larutan dengan menggunakan
stirer kemudian diletakkan diatas hotplate. Selanjutnya, ditambahkan HCl
pekat sedikit demi sedikit sampai pH mencapai 8. Masukkan ke dalam gelas
ukur, kemudian volume ditepatkan dengan aquades hingga volume larutan
menjadi 100 ml.
• NaCl 5 M(l00 ml): Timbang NaCl sebanyak 29.22 gram. Masukkan ke
dalam erlenmeyer dan ditambahkan 80 ml aquades. Aduk campuran larutan
dengan menggunakan stirrer kemudian diletakkan diatas hotplate. Masukkan
ke dalam gelas ukur, kemudian volume ditepatkan dengan aquades hingga
volume larutan menjadi 100 ml.
**Semua bahan di atas disterilkan dengan menggunakan autoklaf kecuali CTAB
5%.
B. Pembuatan Larutan Buffer
• Buffer Ekstraksi/CTAB (100 ml): Campurkan 40 ml CTAB 5%, 25.1 ml
NaCl 5 M, 4 ml EDTA 0.5 M pH 8.0, 10 ml Tris HCl 1 M pH 8.0 dan 20.8 ml
• Buffer TAE 50 X (100 ml): Campurkan 24.2 ml Tris HCl 1 M pH 7.4, 5.7
ml Asam Asetat Glasial, 10 ml EDTA 0.5 M PH 8.0, dan aquades hingga
volume larutan menjadi 100 ml.
• Buffer TAE 1X (500 ml): Campurkan 10 ml Buffer TAE 50 X dan 490 ml
aquades.
• Buffer TE (50 ml): Campurkan 0.5 ml Tris HCl 1 M pH 8.0, 0.1 m lEDTA
0.5 M pH 8.0 dan 49.4 ml aquades.
• Kloroform Isoamilalkohol 24 : 1 (50 ml): Campurkan 48 ml Kloroform dan
2 ml Isoamilalkohol.
Lampiran 7. Hasil uji kuantitas dengan nanospektrometer
Sampel A260/A280 Konsentrasi (ng/µl)
6D 2.38 765.60
6B 2.01 244.90
7D 2.45 616.60
7B 2.09 255.00
9D 1.83 172.50
9B 2.15 500.60
15D 2.06 407.00
15B 2.04 422.50
16D 2.04 817.60
16B 2.06 400.10
17D 2.57 739.40
17B 2.08 712.50
18D 1.63 257.20
18B 2.01 66.60
19D 2.36 692.10
19B 1.78 60.40
21D 1.78 270.80
21B 3.17 336.40
23D 1.90 226.90
23B 2.11 597.80
25D 2.04 471.70
25B 2.08 606.30
28D 1.88 570.30
28B 2.11 468.30
30D 1.94 929.90
30B 2.15 786.80
31D 2.00 338.70
31P 1.95 53.50
32D 2.03 951.20
32O 1.74 706.09
33D 1.99 724.30
33A 1.96 112.50
34D 2.05 863.40
34A 1.89 120.50
Lampiran 8. Data binari skoring
6B 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0
6D 0 0 0 0 0 0 0 1 1 0 1 0 0 1 0 0
7B 0 0 0 1 0 0 0 0 1 0 0 0 1 1 1 0
7D 0 1 0 1 0 0 0 0 1 0 1 0 1 1 0 0
9B 0 0 0 0 0 0 0 1 1 0 0 0 0 1 0 0
9D 0 0 0 1 0 0 0 1 1 0 1 0 0 1 0 0
15B 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0
15D 0 0 0 0 0 0 0 1 1 0 1 0 0 1 0 0
16B 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0
16D 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0
17B 0 0 0 0 0 0 0 1 1 1 0 0 0 1 0 0
17D 0 0 0 1 0 0 0 1 1 1 1 0 0 1 1 0
18B 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0
18D 0 0 0 0 0 0 0 1 1 0 1 0 0 1 0 0
19B 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0
19D 0 0 0 1 1 0 1 1 1 0 1 0 0 1 0 1
21B 0 0 0 1 0 0 0 0 1 0 1 0 0 1 0 1
21D 0 0 0 1 0 0 0 1 1 0 1 0 0 1 0 1
23B 0 0 0 1 0 1 1 1 1 0 1 0 0 1 0 1
23D 0 0 1 1 0 1 1 1 1 0 1 0 0 1 0 1
25B 0 0 0 1 0 1 1 1 1 0 1 0 0 1 0 0
25D 0 0 0 1 0 1 1 1 1 0 0 0 0 1 0 0
28D 0 0 0 1 0 1 1 1 1 0 0 1 0 1 0 1
30B 0 0 0 1 0 0 1 1 1 0 1 0 0 1 0 1
30D 0 0 0 1 0 1 0 1 1 0 1 0 0 1 0 1
31D 0 0 0 1 0 0 0 1 1 1 1 0 0 1 0 0
31P 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0
32D 1 1 0 1 0 0 1 1 1 1 0 1 1 0 0 0
32O 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0
33A 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0
33D 0 0 0 0 0 0 0 1 1 0 1 0 0 1 0 0
34A # # # # # # # # # # # # # # # #
34D 0 0 0 1 0 0 0 1 1 0 1 0 0 1 0 0
35A # # # # # # # # # # # # # # # #
35D 0 0 0 1 0 0 0 1 1 0 1 0 0 1 0 0
36A # # # # # # # # # # # # # # # #
Lampiran 9. Data PCoA (Principal Coordinates Analysis)
Sampel
Axis 1 Axis 2 Axis 3 Axis 4 Axis 5
Coord. Cos² Coord. Cos² Coord. Cos² Coord. Cos² Coord. Cos²
6B -0.2911 769 0.1363 169 -0.0297 8 0 0 0 0
6D 0.026 24 0.1264 566 -0.1287 586 0 0 0 0
7B 0.3263 461 -0.1167 59 -0.1604 111 0 0 0 0
7D 0.3996 819 -0.1038 55 -0.1632 137 0 0 0 0
9B -0.0918 229 0.143 557 -0.0929 235 0 0 0 0
9D 0.1321 859 0.0885 386 -0.0456 102 0 0 0 0
15B -0.2911 769 0.1363 169 -0.0297 8 0 0 0 0
15D 0.026 24 0.1264 566 -0.1287 586 0 0 0 0
16B -0.2911 769 0.1363 169 -0.0297 8 0 0 0 0
16D -0.2911 769 0.1363 169 -0.0297 8 0 0 0 0
17B -0.058 45 0.1058 151 -0.0761 78 0 0 0 0
17D 0.1852 462 0.0318 14 -0.0675 61 0 0 0 0
18B -0.2911 769 0.1363 169 -0.0297 8 0 0 0 0
18D 0.026 24 0.1264 566 -0.1287 586 0 0 0 0
19B -0.5406 951 0.0551 10 0.0952 29 0 0 0 0
19D 0.2288 566 0.0046 0 0.1305 184 0 0 0 0
21B 0.4169 -0.0567 20 -0.1091 75 0 0 0 0
21D 0.179 746 0.0608 86 0.0069 1 0 0 0 0
23B 0.2367 643 0.0054 0 0.1661 317 0 0 0 0
23D 0.2473 533 -0.0181 3 0.1926 323 0 0 0 0
25D 0.1327 233 0.0327 14 0.2021 541 0 0 0 0
28B 0.0478 118 0.1026 544 -0.002 0 0 0 0 0
28D 0.1876 275 -0.0292 7 0.313 765 0 0 0 0
30B 0.2144 736 0.0298 14 0.1109 197 0 0 0 0
30D 0.2103 660 0.0351 18 0.0718 77 0 0 0 0
31D 0.1587 538 0.0611 80 -0.0288 18 0 0 0 0
31P -0.2911 769 0.1363 169 -0.0297 8 0 0 0 0
32D 0.0827 27 -0.1254 63 0.2718 294 0 0 0 0
32O -0.5406 951 0.0551 10 0.0952 29 0 0 0 0
33A -0.5406 951 0.0551 10 0.0952 29 0 0 0 0
33D 0.026 24 0.1264 566 -0.1287 586 0 0 0 0
34A -0.1795 40 -0.6661 546 -0.0537 4 0.528 343 0.2335 67
34D 0.1321 859 0.0885 386 -0.0456 102 0 0 0 0
35A -0.1795 40 -0.6661 546 -0.0537 4 -0.4662 267 0.3405 143
35D 0.1321 859 0.0885 386 -0.0456 102 0 0 0 0
36A -0.1795 40 -0.6661 546 -0.0537 4 -0.0618 5 -0.574 405
36D 0.0973 103 0.0509 28 -0.1837 366 0 0 0 0
Keterangan : Axis Eigenvalue Inertia%
1 0.06456 33.67
2 0.04319 22.52
3 0.0147 7.67
4 0.01316 6.86
Lampiran 11. Data ukuran pasangan basa amplikon DNA
Sampel Produk PCR
6B 6D 7B 7D 9B 9D 15B 15D 16B 16D 17B 17D 18B 18D
843 1934 2248 1934 1934 1934 843 1934 843 843 1934 2248 843 1934
738 1060 1934 1756 843 1060 738 1060 738 738 991 1934 738 1060
843 1756 1060 738 843 843 843 991 843
738 1060 843 738 738 738 843 738
340 843 428 428 738
Lampiran 12. Data ukuran pasangan basa amplikon DNA
Sampel Produk PCR
19B 19D 21B 21D 23B 23D 25B 25D 28B 28D 30B 30D 31D 31P
738 3029 3029 3029 3029 3029 1934 1934 1934 3029 3029 3029 1934 843
2248 1934 1934 1934 1934 1060 843 843 1934 2248 2248 1060 738
1934 1060 1060 1060 1060 843 738 738 1146 1934 1934 991
1060 843 843 843 843 738 568 428 843 1060 1060 843
843 428 738 738 738 568 501 738 843 843 738
738 428 568 568 501 428 568 738 738 428
568 501 501 428 501 568 501
462 428 428 428 428 428
Lampiran 13. Data ukuran pasangan basa amplikon DNA
Sampel Produk PCR
32D 32O 33A 33D 34A 34D 35A 35D 36A 36D
1756 738 738 1934 1934 1934 2248
1146 1060 1060 1060 1934
991 843 843 843 1060
843 738 738 738 843
738 738 428 428 738
568 340
428
340
Lampiran 14. Foto kegiatan penelitian
Isolasi DNA
Uji Kuantitas
Elektroforesis
DAFTAR PUSTAKA
Allorerung, D., M. Syakir, Z. Poeloengan, Syafaruddin, dan W. Rumini. 2010. Budidaya Kelapa Sawit. Pusat Penelitian dan Pengembangan Perkebunan. Bogor.
Bahagiawati. 2011. Bioteknologi jalan pintas angkat produksi petani. Agro Inovasi. Edisi 16-22 Maret 2011 No. 3397 Tahun XLI.
Ferreira, M. E. and D. Grattapaglia. 1998. Introducao ao uso de marcadores moleculares em analise genetica. Embrapa-Cenargen. Brasilia.
Graham A. dan C. R. Newton. 1997. PCR (Polymerase Chain Reaction). Ed Ke-2. New York: Springer Verlag.
Hadi, M. M. 2004. Teknik Berkebun Kelapa Sawit. Adicita. Yogyakarta.
Handoyo, D. dan Rudiretna, A. 2000. Prinsip Umum dan Pelaksanaan Polymerase Chain Reaction (PCR) [General Principles and Implementation of Polymerase Chain Reaction]. Unitas, 9 (1): P. 17-29.
Hartley, C. 1988. The Oil Palm. London: Longman.
Kementrian Perdagangan. 2013. Market Brief Kelapa Sawit dan Olahannya. ITPC – Hamburg.
Kiswanto, J. H. Purwanta., dan B. Wijayanto. 2008. Teknologi Budidaya Kelapa Sawit. Balai Besar Pengkajian Dan Pengembangan Teknologi Pertanian, Badan Penelitian dan Pengembangan Pertanian. Lampung.
Lincoln, R. E. dan John W. P. 1950. Inheritance of beta-carotene in tomatoes. Genetics. 35(206):206-212.
Malaysian Palm Oil Board. 2003. Natural Palm Oil from Tropical Tradition.
Mangoensoekarjo, S. dan H. Semangun. 2008. Manajemen Agrobisnis Kelapa Sawit. Gadjah Mada University Press. Yogyakarta.
Moehs, C. P., Tian L., Osteryoung K. W., Dellapenna D. 2001. Analysis carotenoid biosynthetic gene expression during marigold petal development. Plant Mol. Biology. 45: 281-293.
Olson, J. A. 1999. Carotenoids. dalam: Shils ME, Olson JA, Shike M, Ross AC, eds. Modern Nutrition in Health and Disease, 9th edition. Baltimore, MD: Williams & Wilkins. Hal. 525 – 541.
Padmalatha, K., & Prasad, M.N.V. (2006). Optimization of DNA isolation and PCR protocol for RAPD analysis of selected medicinal and aromatic plants of conservation concern from Peninsular India. African Journal of Biotechnology, 5(3), 230-234.
Perrier, X. and Jacquemoud-Colled J.P. 2009. DARwin Software. http://darwin.cirad.fr/darwin
Puchooa, D. (2004). A simple, rapid and efficient method for the extraction of genomic DNA from lychee (Litchi chinensis Sonn.). African Journal of Biotechnology, 3(4), 253-255.
Putri, L. A. P. 2010. Pendugaan Parameter Genetik dan Karakterisasi Molekuler Keragaman Genetik dengan Marka Mikrosatelit (SSR) pada Kelapa Sawit. Disertasi. Program Pascasarjana IPB. Bogor.
Putri, L. A. P., Muhdi, Meiriani, dan Diana S. 2007. Desain primer spesifik untuk gen yang berperan pada biosintesis beta karoten kelapa sawit. Agrista. Vol. 11 No. 2., 73-80.
Rasid, O. A., Kadir A. P. G., Singh R., dan Ravigadevi S. 2011. Lycopene cyclase genes for modification of carotenoid content. MPOB Information Series No. 480.
Rasid, O. A., Singh R., Harikhrishna K., Ho C. L., Cheah S. C. 2003. Isolation of partial DNA sequence coding for lycopene β-cyclase from oil palm (E. guineensis). Proceedings of the PIPOC 2003 International Palm Oil Congress (Agriculture). Kuala Lumpur, Malaysia. Pp: 781-788
Rasid, O. A., Wan N. S., Nor H. A., Masura S. S., Zulqarnain M., Ho C. L., Sambanthamurthi R., dan Suhaimi N. 2008. RT-PCR amplification and cloning of partial DNA sequence coding for oil palm (Elais oleifera) phytoene synthase gene. AsPac J. Mol. Biol. Biotechnol., Vol 16(1): 17-24
Reddy, B. V. S., Ramesh S., Longvah T. 2005. Prospects of breeding for micronutrients and beta-carotene-dense sorghums. Journal of SAT Agricultural Research, 14 pages. DOI:10.3914/ICRISAT.0126.
Sambanthamurthi, R., Sundram, K. & Tan, Y. 2000. Chemistry and biochemistry of palm oil. Prog. Lipid Res. 39, 507–558.
Santra M., Santra D. K., Rao V. S., Taware S. P., Tamhankar S. A. 2005. Inheritance of β-carotene concentration in durum wheat (Triticum turgidum L. ssp. durum). Euphytica. 144: 215–221.
Sastrosayono, S. 2003. Budidaya Kelapa Sawit. Agromedia Pustaka. Jakarta.
Singh, R., Low, E. T., Ooi, L. C., Ong-Abdullah, M., Nookiah, R., Ting, N. C., et al. (2014). The oil palm VIRESCENS gene controls fruit colour and encodes a R2R3-MYB. Nat. Commun. 5, 4106. doi: 10.1038/ncomms5106
Soehardjo. 1999. Vademescum Kelapa Sawit. PT. Perkebunan Nusantara IV (Persero). Pematang Siantar.
Suryanto, D. 2003. Melihat keanekaragaman organisme melalui beberapa teknik genetika molekuler.
Wilkins, T.A. and Smart, L.B. 1996. Isolation of RNA from plant tissue in Krieg, P.A. (ed). A Laboratory guide to RNA isolation, analysis and synthesis. New York: Wiley – Liss.
Ye J., Coulouris G., Zaretskaya I., Cutcutache I., Rozen S., dan Thomas L. M. 2012. Primer-BLAST: A tool to design target specific primers fro polymerase chain reaction. BMC Bioinformatics. 13:134.
Yuwono, T. 2006. Teori dan Aplikasi Polymerase Chain Reaction (PCR). Penerbit ANDI. Yogyakarta.
BAHAN DAN METODE
Waktu dan Tempat Penelitian
Penelitian ini dilakukan di Laboratorium Kultur Jaringan Tanaman
Fakultas Pertanian Universitas Sumatera Utara Medan dan
Laboratorium Bio Molekuler PT. Socfin Indonesia SSPL Bangun Bandar
Desa Martebing Kecamatan Dolok Masihul Kabupaten Serdang Bedagai
Sumatera Utara. Penelitian ini dilaksanakan pada bulan Februari 2016 sampai
bulan Agustus 2016.
Bahan dan Alat Penelitian
Bahan yang digunakan dalam penelitian ini adalah 4 sampel akar muda, 13
sampel daging buah muda, 19 sampel daun muda (daun tombak), 1 sampel
pollen, dan 1 sampel bunga betina yang berasal dari tanaman kelapa sawit yang
dikeluarkan oleh PT. Socfin Indonesia, air, aquades, ddH2O,
Polyvinylpolypyrolidone (PVPP), nitrogen cair, buffer ekstraksi CTAB,
β-merkaptoethanol, tube 2 ml, tube 1,5 ml, tube 0,2 ml, Khloroform IsoAmil
Alkohol, isopropanol, buffer TE, ethanol absolute, ethanol 70%, larutan TBE 1x
dan 0,5x, larutan TAE 1x, gel agarose, es, nuclease free water, dan loading buffer.
Digunakan dua primer yaitu primer spesifik Beta (terdiri dari primer Beta Forward
(5’-CCA CTG GCT TCT CTA GGC-3’) dan primer Beta Reverse (5’-CCT CTC
TAT CGG CCA GAC-3’)) dan primer SSR-3574 sebagai primer pembanding.
Alat yang digunakan dalam penelitian ini adalah mortar dan alu, alumunium foil,
pestle, mikropipet, rak tube, waterbath, stirer, centrifuge, kulkas, freezer,
komputer, nanospec, timbangan analitik, erlenmeyer, microwave, cetakan agarose,
Metode Penelitian
Metode penelitian yang digunakan adalah metode marka molekuler
dengan menggunakan primer spesifik berdasarkan PCR
PELAKSANAAN PENELITIAN
Pengambilan Sampel
Semua alat disiapkan seperti pisau dan plastik sampel, lalu diambil sampel
akar muda, buah muda, daun muda (daun tombak), pollen, dan bunga betina yang
berasal dari tanaman kelapa sawit yang dikeluarkan oleh PT. Socfin Indonesia di
lapangan dari masing-masing populasi dengan cara memotong bagian
menggunakan pisau yang telah disemprot alkohol setiap kali sampel diambil.
Sampel dibersihkan dengan air bersih kemudian dibuang bagian yang tidak
diperlukan dan kemudian dipotong kecil-kecil. Dimasukkan potongan sampel ke
alumunium foil atau amplop coklat lalu simpan ke dalam kulkas.
Isolasi DNA
Sampel yang sudah dicuci dan bersih ditambahkan ke dalam mortar dingin
sebanyak 0,1 g. Kemudian ditambahkan ± 0,1 g Polyvinylpolypyrolidone (PVPP)
dan nitrogen cair, gerus hingga halus. Serbuk halus yang dihasilkan kemudian
dimasukkan ke dalam tube 1,5 ml yang berisi 1 ml buffer Castillo yang telah
dipanaskan dan diberi 2 μl β-merkaptoethanol 1%. Campuran tersebut kemudian
dikocok menggunakan stirer, lalu dipanaskan selama 30 menit pada suhu 65oC di
waterbath. Larutan dibiarkan dingin pada suhu kamar lalu, ditambahkan
chloroform isoamil alkohol 1 Volume. Larutan tersebut disentrifius selama 10
menit dengan kecepatan 11.000 rpm. Supernatan diambil dan dipindahkan ke
dalam tube 2 ml yang baru. Kemudian ditambahkan 1 volume Chloroform:
Isoamil alkohol, Tube tersebut dikocok bolak balik, kemudian tube tersebut
disentrifius selama 10 menit dengan 11 kecepatan 13.000 rpm, cairan bagian atas
ditambahkan isopropanol dingin sebanyak 1 volume. Dikocok perlahan hingga
homogen kemudian simpan dalam kulkas 1 – 4 jam, kemudian disentrifius selama
10 menit dengan kecepatan 11.000 rpm. Cairan dan pellet DNA dibuang dan
dikeringkan dengan cara membalikkan tabung. Pellet DNA dilarutkan dalam
0,1 ml buffer TE, kemudian ditambahkan ethanol absolute sebanyak 2,5 volume.
Larutan tersebut dikocok hingga homogen dan disimpan dalam freezer selama
30 menit atau semalam. Kemudian larutan tersebut disentrifius selama 10 menit
dengan kecepatan 12.000 rpm. Cairan tersebut kemudian dibuang dan pellet DNA
diambil kemudian dicuci dengan ethanol 70% lalu dikeringkan. Pellet DNA yang
sudah kering dilarutkan dalam 50 μl buffer TE atau nuclease free water,
kemudian simpan dalam freezer. Kualitas DNA kemudian diuji dengan
elektroforesis gel agarose 2% dan konsentrasinya diukur dengan nanospec pada
panjang gelombang 260 dan 280 nm.
Uji Kuantitas DNA
Sebanyak 2 μl stok DNA diukur menggunakan Nanospektrometer sinar
UV. Absorban diukur pada panjang gelombang 260 nm dan 280 nm. Kemurnian
DNA ditentukan dari nilai perbandingan A260/A280.
Konsentrasi DNA = faktor konversi x faktor pengenceran x A260
Tingkat kemurnian DNA ditetapkan dengan membandingkan nilai absorbansi
pada A260 nm terhadap A280 nm. Bila rasio perbandingan menunjukan nilai
1,8 ‒ 2,0 maka tingkat konsentrasi DNA dinyatakan memenuhi syarat untuk
Uji Kualitas DNA
Uji kualitas DNA dilakukan dengan elektroforesis metode standar dengan
cara memasukkan 2 μl stok DNA ditambah 1 μl loading dye ke dalam sumur gel
agarose 0.8%.
Sebelum dilakukan elektroforesis disiapkan gel agarose konsentrasi 0,8%
(b/v). Agarose ditimbang 1,04 g kemudian dilarutkan kedalam 130 ml
buffer TAE 1x atau buffer TBE 1x. Larutan tersebut dimasukkan kedalam
Erlenmeyer kemudian dipanaskan dan diaduk dengan pengaduk magnetik hingga
larutan menjadi bening kemudian didinginkan dengan cara dialirkan tabung
tersebut pada air yang mengalir. Setelah larutan agak dingin (suhu ± 60oC),
ditambahkan 1 μl etidium bromida, diaduk sambil dipanaskan kembali ± 2 menit
dan didinginkan sampai ± 60oC kemudian larutan dimasukkan dalam cetakan agar
yang telah dipasang sisir pembuat lubang dan dibiarkan memadat selama ± 40
menit. Gel yang telah memadat dimasukkan kedalam elektroforesis dan diberi
larutan TAE 1x atau TBE 0,5x sebanyak ± 670 ml (hingga terendam). Contoh
DNA yang telah disiapkan dimasukkan ke dalam sumur gel. Setelah semua lubang
sumur gel berisi selanjutnya dielektroforesis. Running elektroforesis dilakukan
pada kondisi 75 volt selama 60 menit. Visualisasi DNA yang telah
dielektroforesis dilakukan dengan UV Transluminator dan didokumentasikan.
Kualitas DNA dinyatakan baik bila hasil elektroforesis menunjukkan pola
pita yang terang dan fokus. Artinya DNA yang dihasilkan cukup solid, utuh dan
Amplifikasi dan Genotiping
Amplifikasi dilakukan dengan menggunakan primer spesifik Beta Forward
dan Beta Reverse yang telah teruji dari penelitian Putri et al. (2007).
Sebelum running PCR dilakukan pengenceran DNA dengan mengambil
9 µl stok DNA dan ditambah 15 µl ddH2O sehingga diperoleh 24 µl aliquot DNA.
Kemudian dilakukan pengenceran primer yaitu tube primer disentrifius selama 5
menit setelah itu ditambahkan ddH2O sesuai ukuran molar. Dibuat aliquot primer
yaitu dengan mengambil 10 – 15 µl stok primer.
Persiapan awal PCR adalah mencairkan komponen untuk running PCR
yaitu paket PCR produksi Promega dalam kotak berisi pecahan es. Untuk
mempermudah pembuatan larutan master dimisalkan 5 sampel yang akan
digunakan maka larutan master terdiri atas: ddH2O 3,5 µl x 5 = 17,5 µl, Go Tag
7,5 µl x 5 = 37,5 µl, aliquot primer 1 µl x 5 = 5 µl. Dari tube diambil 15 µl ke tube
yang lain sehingga diperoleh 5 tube untuk PCR dan ditambahkan masing-masing
DNA sebanyak 2 µl. Kemudian tabung dispin manual. Tabung berisi stok DNA
dan campuran master dimasukkan dalam blok sampel di mesin PCR dengan
annealing 54o C. Reaksi amplifikasi thermocycler didesain waktu, suhu dan
jumlah siklus termal 30 ‒ 35 kali (± 2 jam 51 menit) yang telah digunakan pada
tanaman kelapa sawit, tertera pada Tabel 1 (Putri et al., 2007).
Setelah reaksi PCR selesai DNA hasil amplifikasi disimpan dalam suhu
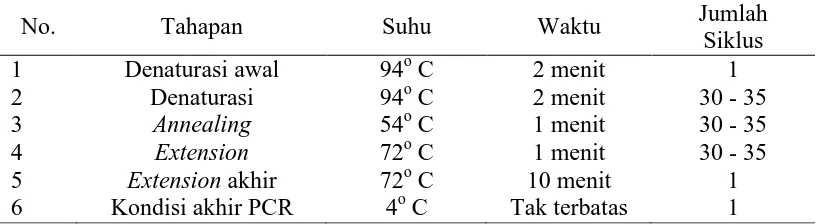
Tabel 1. Proses amplifikasi PCR
No. Tahapan Suhu Waktu Jumlah
Siklus
1 Denaturasi awal 94o C 2 menit 1
2 Denaturasi 94o C 2 menit 30 - 35
3 Annealing 54o C 1 menit 30 - 35
4 Extension 72o C 1 menit 30 - 35
5 Extension akhir 72o C 10 menit 1
6 Kondisi akhir PCR 4o C Tak terbatas 1
Elektroforesis
Gel agarose disiapkan dengan konsentrasi 2%, kemudian dicetak dan
dimasukkan ke dalam bak elektroforesis, loading buffer kemudian disiapkan
dalam parafilm. Loading buffer dan sampel kemudian dimasukkan ke dalam
sumur-sumur elektroforesis. Alat elektroforesis dijalankan dengan
menghubungkannya dengan power supply dan ditunggu ± 1 jam. Gel agarose
kemudian dikeluarkan dari bak elektroforesis kemudian hasil running
elektroforesis difoto menggunakan Geldoc.
Analisis Data
Penentuan ukuran pita hasil PCR
Ukuran fragmen basa (pasangan basa = bp) produk PCR ditentukan
dengan menggunakan UVITEC Cambridge FireReader. Fragmen DNA yang
digunakan sebagai standart ukuran yaitu 1 kb DNA ladder. Data gambar hasil
elektroforesis yang dimasukkan ke dalam program akan dideteksi muncul
tidaknya band dan dinilai berdasarkan marker value yang telah dimasukkan.
Analisis hubungan genetik
Untuk penentuan kekerabatan genetik, produk PCR diskoring berdasarkan
muncul tidaknya pita. Pita yang muncul pada gel diasumsikan sebagai alel.
masing-masing sampel. Berdasarkan ada atau tidaknya pita, profil pita diterjemahkan ke
dalam data biner. Pita yang muncul diberi kode 1 (ada) dan 0 (tidak ada)
(Ferreira dan Grattapaglia, 1998).
Data skoring kemudian diolah menggunakan software DARwin 6.0.13
(Perrier dan Jacquemoud-Collet, 2009) dimana matriks jarak atau ketidaksamaan
genetik untuk semua kombinasi pasangan individu dapat dilakukan dengan dua
tipe analisis deskriptif dari keragaman: (i) Principal Coordinates Analysis
(PCoA), suatu jenis analisis faktorial pada tabel ketidaksamaan untuk
mendapatkan group origin utama dan (ii) Neighbor-Joining Tree (NJTree) untuk
HASIL DAN PEMBAHASAN
Hasil
Beberapa sampel yang diperoleh dari lapangan yang terdiri 19 sampel
daun muda (daun tombak), 13 sampel buah muda (9 jenis virescens dan 4 jenis
nigrescens), 4 sampel akar kecambah, 1 sampel pollen (serbuk sari), dan 1 sampel
bunga betina tanaman kelapa sawit yang dikeluarkan oleh PT. Socfin Indonesia
diperoleh identitas yang dapat dilihat pada Tabel 2.
Uji kualitas DNA dapat dilakukan untuk mengetahui hasil dari isolasi
DNA sampel tanaman kelapa sawit dengan metode Orozco-Castillo et al. (1994)
yang dimodifikasi dengan penambahan Polyvinilpolypirolidone (PVPP) dan
β-mercaptoethanol dapat digunakan untuk proses PCR. Sedangkan hasil dari uji
kuantitas DNA stok dapat dilihat pada Lampiran 7.
Hasil amplifikasi DNA dengan menggunakan marka molekuler terkait
karakter beta karoten pada tanaman kelapa sawit (primer Beta) menunjukkan
pola pita (amplikon) yang beragam dengan persentase polimorfisme yang tinggi
sebesar 100%. Terdapat perbedaan pola pita pada sampel yang berasal dari
individu yang sama tetapi berbeda sumber bagian tanaman yang diisolasi. Pada
penelitian ini diperoleh fragmen pita polimorfik dengan ukuran berkisar ± 155 bp
‒ 3172 bp.
Berdasarkan analisis faktorial PCoA dapat diketahui bahwa keragaman
dari 38 sampel DNA stok kelapa sawit sangat tinggi yaitu sebesar 56.19 % dan
analisis dendogram menunjukkan 38 sampel DNA stok kelapa sawit terbagi
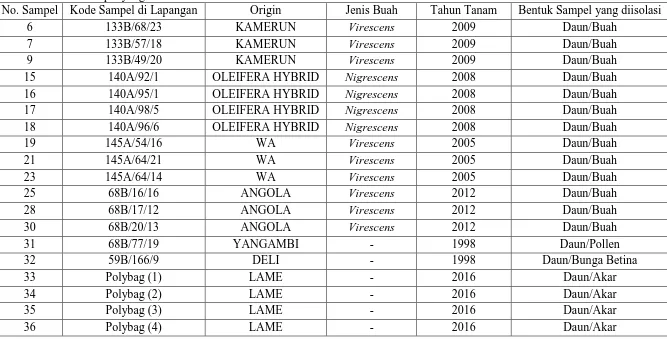
Tabel 2. Identitas sampel yang dikeluarkan oleh PT. Socfin Indonesia
No. Sampel Kode Sampel di Lapangan Origin Jenis Buah Tahun Tanam Bentuk Sampel yang diisolasi
6 133B/68/23 KAMERUN Virescens 2009 Daun/Buah
7 133B/57/18 KAMERUN Virescens 2009 Daun/Buah
9 133B/49/20 KAMERUN Virescens 2009 Daun/Buah
15 140A/92/1 OLEIFERA HYBRID Nigrescens 2008 Daun/Buah
16 140A/95/1 OLEIFERA HYBRID Nigrescens 2008 Daun/Buah
17 140A/98/5 OLEIFERA HYBRID Nigrescens 2008 Daun/Buah
18 140A/96/6 OLEIFERA HYBRID Nigrescens 2008 Daun/Buah
19 145A/54/16 WA Virescens 2005 Daun/Buah
21 145A/64/21 WA Virescens 2005 Daun/Buah
23 145A/64/14 WA Virescens 2005 Daun/Buah
25 68B/16/16 ANGOLA Virescens 2012 Daun/Buah
28 68B/17/12 ANGOLA Virescens 2012 Daun/Buah
30 68B/20/13 ANGOLA Virescens 2012 Daun/Buah
31 68B/77/19 YANGAMBI - 1998 Daun/Pollen
32 59B/166/9 DELI - 1998 Daun/Bunga Betina
33 Polybag (1) LAME - 2016 Daun/Akar
34 Polybag (2) LAME - 2016 Daun/Akar
Uji kualitas DNA
Kualitas DNA dinilai dari uji gel agarose 0.8% hasil running
electrophoresis (elektroforegram). Elektroforegram untuk 38 sampel yang telah
diisolasi DNA dapat dilihat pada Gambar 2 ‒ Gambar 9.
Gambar 2. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun (15, 16, 21, 23, 25, 28, 30) x = sampel lain
Gambar 3. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
B = isolasi DNA berasal dari bagian Buah (7, 9, 15, 16) x = sampel lain
Gambar 4. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun (6 dan 9) B = isolasi DNA berasal dari bagian buah (17, 18, 19) x = sampel lain
x x x x 15D 16D 21D x 23D x 25D x x 28D x 30D
x x x x x x 7B x 9B x x x x x 15B 16B
Gambar 5. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun (15, 16, 25, 30) B = isolasi DNA berasal dari bagian buah (7, 9, 15, 17, 18) * = sampel cadangan
x = sampel lain
Gambar 6. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun ( 33, 34, 35, 36) B = isolasi DNA berasal dari bagian buah (19)
A = isolasi DNA berasal dari bagian akar (33, 34, 35, 36) P = isolasi DNA berasal dari bagian pollen (31)
x = sampel lain
Gambar 7. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun (6, 7, 9, 17, 18, 19, 31) B = isolasi DNA berasal dari bagian buah (23)
x = sampel lain
x x x 15D*16D 25D x x 30D 7B 9B x x 15B 17B*
19B x 33D 34D 35D 36D 33A 34A 35A 36A 31P 17B 18B 19B x
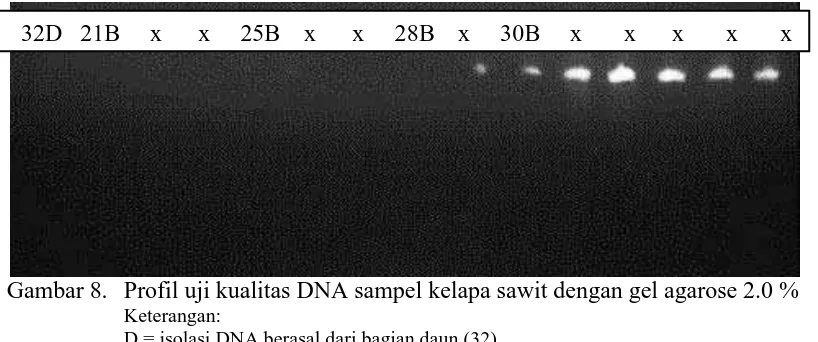
[image:30.595.110.516.85.209.2] [image:30.595.104.520.281.688.2]Gambar 8. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun (32)
B = isolasi DNA berasal dari bagian buah (6, 21, 25, 28, 30) x = sampel lain
Gambar 9. Profil uji kualitas DNA sampel kelapa sawit dengan gel agarose 2.0 % Keterangan:
D = isolasi DNA berasal dari bagian daun (9 dan 32) B = isolasi DNA berasal dari bagian buah (21, 25, 28, 30) P = isolasi DNA berasal dari bagian pollen (31)
x = sampel lain
Dari elektroforegram uji kualitas DNA yang dihasilkan di gel agarose
dapat dilihat pada Gambar 2 – 9, menunjukkan bahwa DNA dari 37 sampel
berhasil diisolasi dan sudah memenuhi syarat untuk dilanjutkan pada proses
amplifikasi mesin PCR. Sampel yang terlihat pada elektroforegram awal memiliki
kualitas yang kurang baik (smear) telah dipurifikasi dan ditunjukkan pada proses
[image:31.595.105.520.76.486.2]elektroforesis selanjutnya, seperti pada pola pita sampel 16D dan 30D pada
Gambar 2 telah terlihat jelas pada Gambar 5.
32D 21B x x 25B x x 28B x 30B x x x x x
[image:31.595.110.519.84.255.2]Keragaan primer spesifik dan ukuran pasangan basa pita
Sebanyak 38 stok sampel DNA telah diamplifikasi menggunakan mesin
PCR dan diseparasi dengan elektroforesis gel agarose 1.2% yang kemudian
divisualisasikan menggunakan UV Transluminator. Elektroforegram produk PCR
menunjukkan bahwa tiga sampel tidak dapat teramplifikasi menggunakan primer
Beta. tiga sampel tersebut adalah 34A, 35A, dan 36A. Terdapat satu sampel yang
menampilkan pita yang tipis yaitu 19B. Sedangkan saat 38 stok sampel yang sama
diamplifikasi menggunakan primer pembanding yaitu primer SSR-3574, semua
sampel dapat teramplifikasi. Elektroforegram sampel yang diampifikasi
menggunakan primer SSR-3574 dapat dilihat pada Lampiran 1 dan 2.
[image:32.595.113.517.361.512.2]
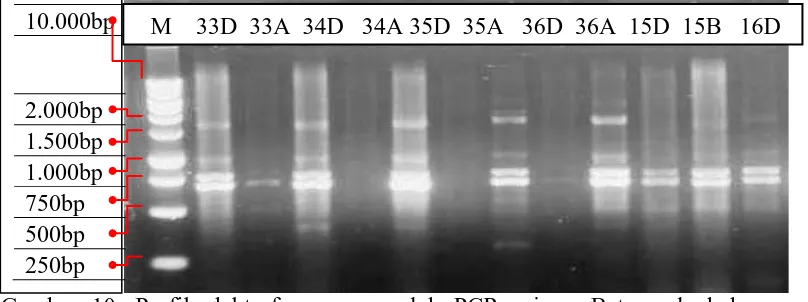
Gambar 10. Profil elektroforegram produk PCR primer Beta pada beberapa sampel kelapa sawit
Keterangan:
M = DNA ladder 1kb (Marker)
A = isolasi DNA berasal dari bagian akar (33, 34, 35, 36) D = isolasi DNA berasal dari bagian daun (15, 16, 33, 34, 35, 36) B = isolasi DNA berasal dari bagian buah (15 dan 16)
10.000bp
2.000bp 1.500bp 1.000bp 750bp 500bp 250bp
Tabel 3. Hasil Amplifikasi Produk PCR Primer Beta Kode
Sampel
Ukuran Pasangan Basa Amplikon Primer Beta
340 bp 428 bp 738 bp 843 bp 1060 bp 1934 bp 2248 bp
33D √ √ √ √
33A √
34D √ √ √ √ √
34A
35D √ √ √ √ √
35A
36D √ √ √ √ √ √
36A
15D √ √ √ √
15B √ √
16D √ √
16B √ √
Keterangan: A = isolasi DNA berasal dari bagian akar (33, 34, 35, 36) D = isolasi DNA berasal dari bagian daun (15, 16, 33, 34, 35, 36) B = isolasi DNA berasal dari bagian buah (15 dan 16)
[image:33.595.111.517.384.539.2]
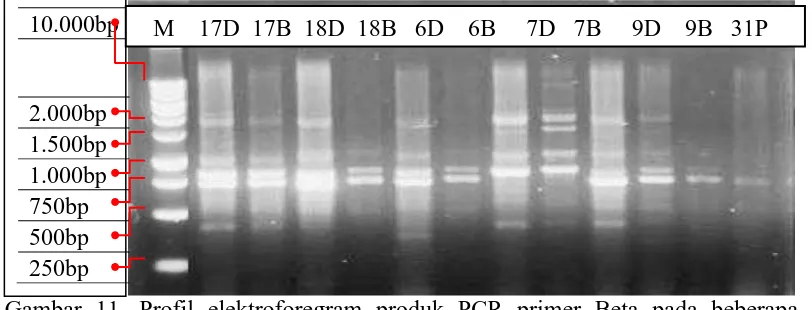
Gambar 11. Profil elektroforegram produk PCR primer Beta pada beberapa sampel kelapa sawit
Keterangan:
M = DNA ladder 1kb (Marker)
D = DNA stok berasal dari bagian daun (6, 7, 9, 17, 18) B = DNA stok berasal dari bagian buah (6, 7, 9, 17, 18) P = DNA stok berasal dari bagian pollen (31)
O = DNA stok berasal dari bagian bunga betina (32) 10.000bp 2.000bp 1.500bp 1.000bp 750bp 500bp 250bp
Tabel 4. Hasil Amplifikasi Produk PCR Primer Beta
Kode Sampel
Ukuran Pasangan Basa Amplikon Primer Beta 340 bp 428 bp 738 bp 843 bp 991 bp 1060 bp 1756 bp 1934 bp 2248 bp
17D √ √ √ √ √ √ √
17B √ √ √ √
18D √ √ √ √
18B √ √
6D √ √ √ √
6B √ √
7D √ √ √ √ √ √
7B √ √ √ √ √
9D √ √ √ √ √
9B √ √ √
31P √ √
32O √
Keterangan: D = DNA stok berasal dari bagian daun (6, 7, 9, 17, 18) B = DNA stok berasal dari bagian buah (6, 7, 9, 17, 18) P = DNA stok berasal dari bagian pollen (31)
O = DNA stok berasal dari bagian bunga betina (32)
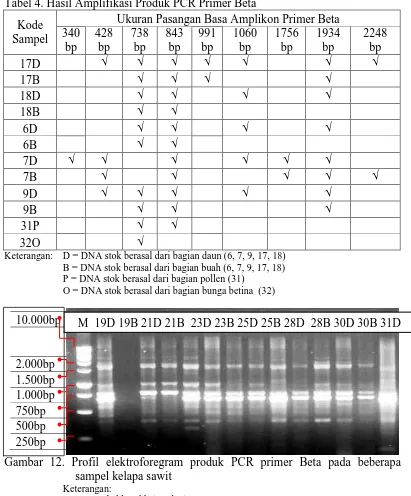
Gambar 12. Profil elektroforegram produk PCR primer Beta pada beberapa sampel kelapa sawit
Keterangan:
M = DNA ladder 1kb (Marker)
D = DNA stok berasal dari bagian daun (19, 21, 23, 25, 28, 30, 31, 32) B = DNA stok berasal dari bagian buah (19, 21, 23, 25, 28, 30) 10.000bp 2.000bp 1.500bp 1.000bp 750bp 500bp 250bp
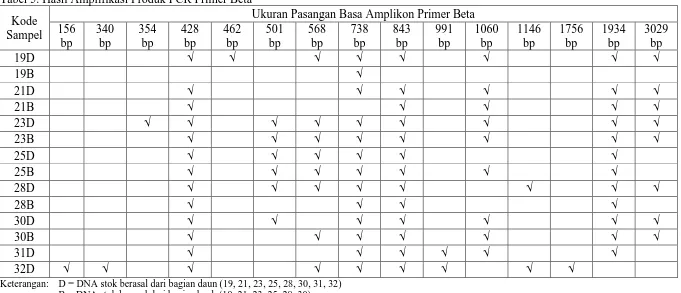
Tabel 5. Hasil Amplifikasi Produk PCR Primer Beta
Kode Sampel
Ukuran Pasangan Basa Amplikon Primer Beta 156 bp 340 bp 354 bp 428 bp 462 bp 501 bp 568 bp 738 bp 843 bp 991 bp 1060 bp 1146 bp 1756 bp 1934 bp 3029 bp
19D √ √ √ √ √ √ √ √
19B √
21D √ √ √ √ √ √
21B √ √ √ √ √
23D √ √ √ √ √ √ √ √ √
23B √ √ √ √ √ √ √ √
25D √ √ √ √ √ √
25B √ √ √ √ √ √ √
28D √ √ √ √ √ √ √ √
28B √ √ √ √
30D √ √ √ √ √ √ √
30B √ √ √ √ √ √ √
31D √ √ √ √ √ √
32D √ √ √ √ √ √ √ √ √
Amplifikasi terhadap 38 sampel kelapa sawit yang dilakukan dengan
primer Beta menghasilkan 16 amplikon yang berukuran ± 156 bp ‒ 3029 bp. Dari
Gambar 10 ‒ 12 dan Tabel 3 ‒ 4, dapat dilihat bahwa terdapat tiga sampel seperti
34A, 35A, dan 36A tidak membentuk amplikon. Sampel-sampel tersebut telah
dilakukan pengulangan PCR dengan primer yang sama agar didapati amplikon
namun tetap tidak teramplifikasi (Lampiran 3).
Persentase polimorfisme primer Beta berada pada 100% polimorfik yang
menunjukkan bahwa keragaman genetik individu yang menjadi sampel adalah
tinggi. Jumlah amplikon terbanyak ada pada sampel 19D (isolasi DNA dari daun
origin WA dan jenis buah virescens), 23D (isolasi dari daun origin WA dan jenis
buah virescens), dan 32D (isolasi dari daun origin DELI). Dari Lampiran 8 ‒ 10
diketahui bahwa data ukuran fragmen basa tertinggi yaitu terdapat pada sampel
19D, 21D, 21B, 23D, 23B, 28B, 30D dan 30B (3029 bp) sedangkan ukuran
fragmen terendah terdapat pada sampel 32D (156 bp).
Analisis hubungan genetik
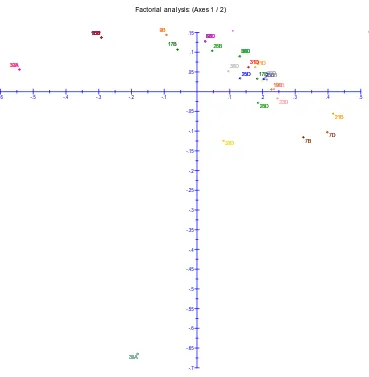
Gambar 13 menunjukkan persentase keragaman genetik berdasarkan
analisis faktorial PCoA (Principal Coordinates Analysis). Melalui software
DARwin 6.0.13, keragaman molekuler dari hasil analisis faktorial menunjukkan
bahwa total keragaman molekuler pada aksis 1 sebesar 33.67 % dan aksis 2
sebesar 22.52 % dengan total 56.19 %. Sebaran genetik sampel-sampel terdapat
Gambar 13. Faktorial analisis PCoA (Principal Coordinates Analysis) aksis 1 (horizontal) dan aksis 2 (vertikal) dengan marka primer Beta
Hasil analisis pengelompokan dengan metode Matrix Dissimilarity Simple
Matching untuk 38 sampel stok DNA kelapa sawit dengan marka primer Beta
menghasilkan dendogram yang menunjukkan bahwa semua sampel dapat
dibedakan dengan jelas antara satu dengan yang lain (Gambar 14)
Factorial analysis: (Axes 1 / 2)
-.6 -.5 -.4 -.3 -.2 -.1 .1 .2 .3 .4 .5
Gambar 14. Profil filogenik Neighbor-Joining dari 38 sampel DNA stok kelapa sawit berdasarkan Matrix Dissimilarity Simple Matching dengan primer Beta
Dari Gambar 14 dapat diketahui bahwa 38 sampel DNA stok kelapa sawit
terbagi menjadi 3 kelompok utama (cluster). Kelompok pertama (22 sampel) yang
terdiri dari origin WA (5 sampel), origin Angola (5 sampel), origin Kamerun
(4 sampel), origin Lame (4 sampel), origin Oleifera Hybrid (3 sampel), dan origin
Yangambi (1 sampel). Kelompok kedua (15 sampel) yang terdiri dari origin
Oleifera Hybrid (5 sampel), origin Lame (4 sampel), origin Kamerun (2 sampel),
Pita spesifik
Dari Gambar 10 ‒ 12 dan Tabel 5 dapat dilihat pada amplikon polimorfik
yang dihasilkan ditemukan pita spesifik yang tidak dijumpai pada sampel yang
lain. Hal ini dapat dilihat pada sampel 19D (isolasi DNA dari daun origin WA dan
jenis buah virescens) dengan ukuran 462 bp, 23D (isolasi DNA dari daun origin
WA dan jenis buah virescens) dengan ukuran 354 bp dan 32D (isolasi DNA dari
daun origin Deli) dengan ukuran 156 bp.
Pembahasan
Berdasarkan hasil uji kualitas DNA yang dilihat secara visual dari gel
agarose 2.0% yang didapat melalui proses elektroforesis pada Gambar 2 ‒ 10
diketahui bahwa 37 sampel yang diuji menunjukkan kualitas yang beragam.
Sampel 7D, 6B, 16B,17D, 18D, 19D, 21D, 23D, 23B, 28D, 33D, 33A, 34D, 35D,
36D memperlihatkan kualitas pita yang baik tanpa memerlukan dilakukannya
purifikasi DNA stok. Sementara itu sampel dengan pita yang terlihat smear pada
awalnya seperti 15D, 15B, 16D, 21B, 25D, 25B, 28B, 30D, 30B, 7B, 9D, 9B,
17B, 18B, 19B, 6D, 31P, 32D perlu dilakukan purifikasi untuk memurnikan DNA
stok agar sampel layak untuk diamplifikasi pada proses PCR selanjutnya. Hal ini
dikarenakan kualitas pita yang smear menunjukkan tingkat kemurnian DNA
rendah yang diakibatkan oleh kontaminasi oleh senyawa lain seperti polisakarida.
Jika dibiarkan maka kontaminasi ini dapat mengganggu proses amplifikasi sampel
pada proses PCR. Hal ini sesuai dengan pernyataan Padmalatha dan Prasad (2006)
yang menyatakan bahwa permasalahan yang umum ditemui dalam proses isolasi
dan purifikasi DNA tanaman adalah degradasi DNA oleh enzim endonuklease,
metabolit sekunder lain yang dapat mengganggu reaksi enzimatik dan didukung
oleh pernyataan Puchooa (2004) yang mengatakan tingkat kemurnian DNA hasil
isolasi juga menjadi salah satu faktor penentu keberhasilan proses amplifikasi
DNA pada proses PCR.
Dari hasil penelitian yang diperoleh dapat dilihat bahwa banyak terdapat
pola pita yang terlihat tipis, redup, maupun smear, misalnya pada Gambar 12.
Hal ini diduga karena adanya senyawa-senyawa polisakarida dan metabolit
sekunder yang masih tersisa pada DNA stok, sesuai dengan pernyataan Wilkins
dan Smart (1996) yang mengatakan senyawa-senyawa kontaminan yang dapat
mengganggu reaksi PCR seperti polisakarida dan metabolit sekunder. Struktur
polisakarida yang mirip dengan asam nukleat akan menyebabkan polisakarida
tersebut akan mengendap bersama dengan asam nukleat. Hal ini bisa juga
disebabkan oleh faktor lain seperti yang dikatakan oleh Handoyo dan Rudiretna
(2000) yang menyatakan perlu diingat bahwa di dalam proses PCR efisiensi
amplifikasi tidak terjadi 100 %, hal ini disebabkan oleh target templat terlampau
banyak, jumlah polimerase DNA terbatas, dan kemungkinan terjadinya
reannealing untai target.
Pada Gambar 10 yang menunjukkan profil amplikon menggunakan primer
Beta didapati beberapa sampel yang tidak teramplifikasi oleh mesin PCR. Sampel
tersebut adalah 34A, 35A, dan 36A yang DNA-nya diisolasi langsung dari bagian
akar. Dari hasil uji kuantitas yang dilakukan menggunakan nanospektrometer
Namun setelah dilakukan proses PCR ulang, ketiga sampel tersebut tetap tidak
teramplifikasi. Hal ini menunjukkan bahwa kontaminasi bukanlah hal yang
menghalangi DNA untuk tidak teramplifikasi menggunakan primer Beta yang
menyandikan parsial fragmen gen lintasan biosintesis beta karoten. Maka dari itu
dapat diambil kesimpulan bahwa hal ini diduga disebabkan oleh beta karoten yang
tidak disintesis pada bagian akar tanaman. Selain itu, primer Beta yang digunakan
adalah primer spesifik yang hanya dapat mengamplifikasi pita dengan sekuen
DNA spesifik.
Perbedaan bagian tanaman yang diisolasi DNAnya dari satu individu yang
sama juga memperlihatkan pola pita yang berbeda. Perbedaan ini terdapat pada
bagian daun dengan buah, daun dengan akar, daun dengan pollen, dan daun
dengan bunga betina. Salah satu contoh sampel yang menunjukkan perbedaan
pola pita yang sangat nyata adalah sampel 31D dan 32D dengan sampel 31P dan
32O. Pada Sampel 31D dan 32D masing-masing membentuk enam dengan ukuran
428 bp ‒ 1934 bp dan sembilan amplikon dengan ukuran 156 bp ‒ 1756 bp.
Sedangkan pada sampel 31P dan 32O pada tiap masing-masingnya hanya
membentuk dua amplikon dengan ukuran 738 bp ‒ 843 bp dan satu amplikon
dengan ukuran 738 bp. Seperti yang telah diduga pada paragraf sebelumnya, hal
ini disebabkan oleh tiap-tiap bagian tanaman membentuk biosintesis beta karoten
yang berbeda. Perbedaan tingkat kandungan metabolit sekunder yang diproduksi
pada tiap bagian tanaman diduga menjadi salah satu penyebab terjadinya
perbedaan amplikon yang dihasilkan. Sebab diketahui bahwa kandungan
metabolit sekunder pada tanaman dapat mengganggu proses amplifikasi pada saat
Pita yang ada pada ukuran 738 bp dan 843 bp dapat teramplifikasi pada 35
sampel menunjukkan bahwa hampir setiap individu tanaman kelapa sawit secara
konsisten memiliki gen yang menyandikan beta karoten pada ukuran pasangan
basa tertentu. Dengan kata lain, primer Beta yang telah didesain oleh Putri et al.
(2007) dapat dijadikan sebagai acuan marka molekuler genetik terhadap karakter
beta karoten untuk tanaman kelapa sawit.
Tanaman kelapa sawit sebagai sampel yang memiliki jenis buah virescens
dan nigrescens menghasilkan amplikon yang berbeda, dimana pada sampel
dengan jenis buah virescens (6, 7, 9, 19, 21, 23, 25, 28, 30) membentuk amplikon
lebih banyak yaitu rata-rata enam amplikon sedangkan sampel dengan jenis buah
nigrescens (15, 16, 17, 18) terdapat sebanyak rata-rata tiga amplikon. Hal ini
diduga disebabkan oleh jenis buah virescens yang memiliki kandungan karotenoid
lebih tinggi dibandingkan dengan jenis buah nigrescens yang mengandung lebih
banyak antosianin. Pernyataan ini didukung oleh Sambanthamurthi et al. (2000)
yang menyatakan bahwa buah nigrescens mengakumulasi antosianin dalam
jumlah yang besar sehingga menyebabkan warnanya ungu gelap hingga hitam
pada bagian ujung buah dan kuning pada bagian dasar saat belum matang, dengan
perubahan warna yang sedikit dari bagian ujungnya saat pematangan. Buah
virescens berwarna hijau saat belum matang, dan menjadi oranye karena
akumulasi karotenoid dan degradasi klorofil yang berhubungan dengan
pematangan buah.
dari pita yang telah diskoring kemudian dianalisis dengan menggunakan software
DAWwin 6.0.13 sehingga didapatkan hasil yaitu dendogram. Dendogram adalah
pohon filogenetik yang menggambarkan pengelompokkan sampel dengan
Operational Taxonomic Unit (OTU) yang berderet rata secara vertikal pada satu
sisi yang berbentuk pohon. Setiap cabang pada dendogram dilakukan analisis
kepercayaan cabang dengan metode bootstrap yang dilakukan sampai 1000 kali.
Pada penelitian ini diketahui bahwa 38 sampel DNA stok kelapa sawit terbagi
menjadi tiga kelompok utama yang masing-masing memiliki sub-kelompok.
Kelompok pertama (sampel 23D, 23B, 28D, 25D, 25B, 30B, 19D, 30D, 21D,
21B, 35D, 34D, 9D, 31D, 17D, 7D, 7B, 36D, 15D, 6D, 18D, dan 33D), kelompok
kedua (sampel 15B, 6B, 16B, 16D, 18B, 31P, 32O, 19B, 33A, 9B, 17B, 36A,
34A, 35A, dan 32D), dan kelompok ketiga (sampel 28B). Hasil analisis faktorial
untuk PCoA juga menunjukkan bahwa keragaman secara molekuler menggunakan
primer Beta adalah sebesar 56.19 %. Hal ini menunjukkan bahwa adanya variasi
genetik di antara sampel yang satu dengan sampel lainnya menyebabkan
perbedaan yang dapat dibedakan atau dikelompokkan dengan jelas. Keragaman
genetik yang tinggi membuka peluang yang besar untuk mendapatkan kultivar
unggul.
Profil amplikon dari 38 sampel yang diamplifikasi menggunakan primer
Beta menunjukkan adanya pita spesifik. Pita spesifik yang tidak ditemukan pada
sampel yang lain hanya terdapat pada sampel 19D (isolasi DNA dari daun origin
WA dan jenis buah virescens) dengan ukuran 462 bp, 23D (isolasi DNA dari daun
origin WA dan jenis buah virescens) dengan ukuran 354 bp dan 32D (isolasi DNA
kesimpulan bahwa primer Beta juga dapat dijadikan sebagai marka molekuler
yang menggambarkan keunikan suatu individu secara genetik yang dapat
dijadikan sebagai identitas individu tersebut. Hal ini sesuai dengan pernyataan
Karsinah (2002) yang mengemukakan dalam penelitiannya menggunakan primer
RAPD terhadap plasma nutfah jeruk bahwa pita-pita spesifik dapat memberi
harapan sebagai identifikasi kultivar. Ditemukannya pita spesifik sebagai
pembeda dengan kultivar lainnya sangat penting dalam identifikasi bibit pada
KESIMPULAN DAN SARAN
Kesimpulan
Hasil amplifikasi DNA dengan menggunakan marka molekuler terkait
karakter beta karoten pada tanaman kelapa sawit (primer Beta) menunjukkan
pola pita (amplikon) yang beragam sebanyak 16 amplikon. Persentase
polimorfisme yang dihasilkan sebesar 100%. Terdapat perbedaan pola pita pada
sampel yang berasal dari individu yang sama tetapi berbeda sumber bagian
tanaman yang diisolasi. Pada penelitian ini diperoleh fragmen pita polimorfik
dengan ukuran berkisar ± 156 bp ‒ 3029 bp. Keragaman molekuler dari 38 sampel
DNA stok kelapa sawit yaitu sebesar 56.19 % dan analisis dendogram
menunjukkan semua sampel terbagi menjadi 3 kelompok utama.
Saran
Perlu dilakukan penelitian lebih lanjut untuk mengetahui tingkat ekpresi
gen pada tiap bagian tanam yang diisolasi dan menganalisis sekuen dari amplikon
primer Beta dari sampel-sampel yang terlihat untuk mendapatkan informasi lebih
banyak terkait parsial fragmen gen yang menyandi lintasan biosintesis beta
TINJAUAN PUSTAKA
Botani Tanaman
Klasifikasi tanaman kelapa sawit menurut Allorerung et al. (2010) adalah
Kingdom: Plantae, Divisi: Embryophyta siphonagama, Kelas: Angiospermae,
Ordo: Monocotyledonae, Famili: Arecaceae (Dahulu Palmae), Sub-famili:
Cocoideae, Genus: Elaeis, Spesies: Elaeis guineensis Jacq.
Sebagaimana tanaman monokotil, kelapa sawit memiliki akar serabut yang
terdiri dari akar utama, akar sekunder, akar tersier, dan akar rambut. Akar utama
(primer) merupakan akar yang pertumbuhannya lurus vertikal ke bawah, searah
pusat bumi. Fungsi utama akar primer adalah menunjang batang agar batang tetap
berdiri kokoh. Akar sekunder dan akar tersier biasanya menyebar secara
horizontal hingga radius yang sama panjang daun, pada zontal hingga radius yang
sama dengan panjang daun, pada kedalaman kurang dari 150 cm, bahkan sebagian
muncul ke permukaan tanah. fungsi utama akar sekunder adalah menjangkau
unsur hara dan air dalam tanah (Hadi, 2004).
Batang kelapa sawit tumbuh tegak (phototrophy) dibalut oleh pangkal
pelepah daun. Batang berbentuk silinderis dan mempunyai diameter 45 - 60 cm
pada tanaman dewasa. Bagian bawah umumnya lebih besar (gemuk) di sebut
bongkol batang atau bowl. Sampai tanaman berumur 3 tahun batang belum terlihat
karena masih terbungkus kelapa yang belum tunas. Tergantung dari varietas dan
kondisi lingkungannya yaitu pupuk yang diberikan, iklim, kerapatan tanam;
Daun terdiri atas tangkai daun (petiole) yang apada kedua tepinya terdapat
dua baris duri (spines). Tangkai daun bersambung dengan tulang daun utama
(rachis), yang jauh lebih panjang dari tangkaiaa dan pada kiri-kanannya terdapat
anak-anak daun (pinna; pinnata). Daun pertama yang keluar pada stadium benih
berbentuk lanset (lanceolate), beberapa minggu kemudian berbentuk daun seperti
bulu (pinnate) atau menyirip. Misalnya pada bibit berumur lima bulan susunan
daun terdiri atas 5 lanset, 4 berbelah dua dan 10 berbentuk buluh . susunan daun
kelapa mirip dengan kelapa (nyiur), yaitu membentuk daun menyirip
(Mangoensoekarjo dan Semangun, 2008).
Susunan bunga terdiri dari karangan bunga yang terdiri dari bunga jantan
(tepung sari) dan bunga betina (putik). Namun, ada juga tanaman kelapa sawit
yang hanya memproduksi bunga jantan. Umumnya bunga jantan dan bunga betina
terdapat dalam dua tandan yang terpisah. Namun, adakalanya bunga jantan dan
betina terdapat dalam tandan yang sama. Bunga jantan selalu masak lebih dahulu
pada bunga betina. Karena itu, penyerbukannya sendiri antara bunga jantan dan
betina dalam satu tandan sangat jarang terjadi. Masa reseptif (masa putik dapat
menerima tepung sari) adalah 3 x 24 jam. Setelah itu, putik akan berwarna hitam
dan mengering (Sastrosayono, 2003).
Hasil utama perkebunan kelapa sawit adalah buah kelapa sawit.
Selanjutnya, buah kelapa sawit di proses (ekstraksi) di pabrik penggiling (mill)
sehingga menghasilkan ekstrak, berupa minyak kelapa sawit yang mentah atau
CPO (Crude Palm Oil) dan minyak kelapa sawit KPO (Kernel Palm Oil). Tandan
bunga betina yang telah menjadi buah disebut tandan buah segar (TBS). Setiap
berdiameter 1,5 - 3 cm. Berat setiap butir buah adalah 10 – 30 g, sehingga satu
TBS pada tanaman dewasa beratnya mencapai 10- 40 kg. Pada umur 3 tahun atau
saat tanaman berbuah untuk pertama kali, berat TBS adalah 3 – 6 kg
(Soehardjo, 1999).
Pada umumnya tanaman kelapa sawit menghasilkan jenis buah nigrescens
atau virescens (Hartley, 1988). Buah nigrescens mengakumulasi antosianin dalam
jumlah yang besar sehingga menyebabkan warnanya ungu gelap hingga hitam
pada bagian ujung buah dan kuning pada bagian dasar saat belum matang, dengan
perubahan warna yang sedikit dari bagian ujungnya saat pematangan. Buah
virescens berwarna hijau saat belum matang, dan menjadi oranye karena
akumulasi karotenoid dan degradasi klorofil yang berhubungan dengan
pematangan buah (Sambanthamurthi et al., 2000).
Dalam kondisi utuh (tidak pecah), biji kelapa sawit bersifat dorman sampai
sekitar enam bulan. Kondisi dorman ini dapat dipatahkan, antara lain dengan
pemanasan biji (Mangoensoekarjo dan Semangun, 2008).
Syarat Tumbuh
Curah hujan yang ideal berkisar 2.000 – 3.500 mm/th yang merata
sepanjang tahun dengan minimal 100 mm/bulan. Di luar kisaran tersebut tanaman
akan mengalami hambatan dalam pertumbuhan dan berproduksi. Curah hujan
antara 1700 – 2.500 mm/th dan 3.500 – 4.000 mm/th menyebabkan tanaman akan
mengalami sedikit hambatan. Di lokasi dengan curah hujan kurang dari
sangat erat kaitannya dengan tinggi tempat di atas permukaan laut (dpl). Tinggi
tempat optimal adalah 200 m dpl, dan disarankan tidak lebih dari 400 m dpl,
meskipun di beberapa daerah, seperti di Sumatera Utara, dijumpai pertanaman
sawit yang cukup baik hingga ketinggian 500 m dpl. Di daerah yang banyak
berawan menyebabkan intensitas matahari yang diterima daun sawit menjadi lebih
rendah. Sebaliknya meskipun curah hujan relatif tinggi tetapi lebih banyak terjadi
sore hingga malam dan perawanan kurang, maka intensitas matahari bias cukup
untuk mendukung fotosintesis yang tinggi. Makin tinggi tempat, suhu makin
rendah dan biasanya disertai perawanan yang lebih lama atau curah hujan yang
tinggi dan makin menjauh dari garis khatulistiwa penyinaran matahari makin
berkurang. Kelapa sawit memerlukan lama penyinaran antara 5 dan 12 jam/hari
(Allorerung et al., 2010).
Kelapa sawit dapat tumbuh pada jenis tanah Podzolik, Latosol,
Hidromorfik Kelabu, Alluvial atau Regosol, tanah gambut saprik, dataran pantai
dan muara sungai. Tingkat keasaman (pH) yang optimum untuk sawit adalah
5,0 – 5,5. Kelapa sawit menghendaki tanah yang gembur, subur, datar, berdrainase
(beririgasi) baik dan memiliki lapisan solum cukup dalam (80 cm) tanpa lapisan
padas. Kemiringan lahan pertanaman kelapa sawit sebaiknya tidak lebih dari 15o
(Kiswanto et al., 2008).
PCR (Polymerase Chain Reaction)
Polymerase Chain Reaction (PCR) merupakan teknik sederhana yang
digunakan untuk memperbanyak molekul DNA secara invitro di dalam
laboratorium. Hasil perbanyakan molekul DNA sangat banyak, karena jumlah
molekul DNA dapat dibuat dalam waktu yang singkat. PCR dapat diaplikasikan
dalam analisis genetik, seperti: diagnosis medis, dan forensik. PCR merupakan
metode yang sangat sensitif sehingga dari satu pasang molekul DNA dapat
diperbanyak menjadi jutaan kali lipat setelah 30 - 40 siklus PCR (Mullis, 1990).
PCR banyak digunakan untuk banyak keperluan karena memiliki beberapa
keuntungan diantaranya cepat, memerlukan DNA dalam jumlah yang sedikit, dan
dapat dilakukan pada tahap dini dengan teknik isolasi DNA sederhana. Hal-hal
yang mempengaruhi keberhasilan dalam reaksi PCR adalah sekuen primer, dNTP,
enzim polymerase, dan suhu annealing (Graham, 1997).
Kemajuan teknologi telah memungkinkan para ilmuan untuk meniru
urutan nukleotida suatu gen dengan cara melakukan amplifikasi DNA dengan
teknik reaksi berantai polimerase (PCR). Amplifikasi DNA dilakukan secara in
vitro (di dalam tabung) dengan menggunakan: (1) enzim DNA polymerase; (2)
dNTP (dinukleotida triphosphat; (3) oligonukleotida primer; dan (4) molekul
DNA cetakan (DNA template) (Yuwono, 2006).
Reaksi PCR secara umum dilakukan dalam empat tahap. Molekul DNA
rantai ganda akan diurai menjadi molekul tunggal dengan pemanasan. Primer akan
menempel pada molekul DNA rantai tunggal pada tempat yang sudah ditentukan.
Selanjutnya enzim polimerase akan memperpanjang primer dengan basa nitrogen
yang tersedia. Tahapan tersebut merupakan cara untuk menggandakan molekul
DNA yang diinginkan (Mullis, 1990). Tahap peleburan (melting) atau denaturasi
(sampai 5 menit) untuk memastikan semua utas DNA terpisah. Pemisahan ini
menyebabkan DNA tidak stabil dan siap menjadi template bagi primer. Tahap
kedua adalah penempelan atau annealing. Primer menempel pada bagian DNA
template yang komplementer uruta basanya. Hal ini dilakukan pada suhu antara
45 – 60°C. Penempelan ini bersifat spesifik. Suhu yang tidak tepat menyebabkan
tidak terjadinya penempelan atau primer menempel di sembarang tempat. Tahap
ketiga adalah pemanjangan atau elongasi. Suhu untuk proses ini tergantung dari
jenis DNA-polimerase yang dipakai. Proses ini biasanya menggunakan
Taq-polimerase dan dilakukan pada suhu 72°C. Durasi tahap ini biasanya 1 menit.
Setelah tahap 3, siklus diulang kembali mulai tahap 1. Tahap 4 menunjukkan
perkembangan yang terjadi pada siklus-siklus selanjutnya. Akibat denaturasi dan
renaturasi, beberapa utas baru menjadi template bagi primer lain dan akhirnya
terdapat utas DNA yang panjangnya dibatasi oleh primer yang dipakai. Jumlah
DNA yang dihasilkan berlimpah karena penambahan terjadi secara eksponensial.
[image:51.595.114.509.497.730.2]Tahapan amplifikasi DNA dapat dilihat pada Gambar 1.
Perlu diingat bahwa di dalam proses PCR effisiensi amplifikasi tidak
terjadi 100 %, hal ini disebabkan oleh target templat terlampau banyak, jumlah
polimerase DNA terbatas, dan kemungkinan terjadinya reannealing untai target.
Ukuran primer yang pendek kemungkinan terjadinya mispriming (penempelan
primer di tempat lain yang tidak diinginkan) tinggi, ini akan menyebabkan
berkurangnya spesifisitas dari primer tersebut yang nantinya akan berpengaruh
juga pada efektifitas dan efisiensi proses PCR (Handoyo dan Rudiretna, 2000).
Genetik Beta Karoten
Didapati nilai duga heritabilitas arti luas yang tinggi sebesar 84,8% untuk
karakter kandungan beta karoten pada tanaman kelapa sawit. Hal ini
mengindikasikan adanya peran efek gen aditif. Dapat diambil kesimpulan bahwa
faktor genetik untuk karakter kandungan beta karoten lebih berperan dari pada
faktor lingkungan (Putri, 2010).
Heritabilitas beta karoten pada gandum durum (Santra et al., 2005) dan
sorghum (Reddy et al., 2005) bervariasi mulai 67 hingga 99%. Nilai duga
heritabilitas pada selada dan jagung lebih rendah namun tergolong cukup tinggi
yaitu 50,81%. Studi sebelumnya telah disepakati bahwa faktor lingkungan
berpengaruh sangat kecil sekali terhadap karakter karotenoid.
Moehs et al. (2001) menyebutkan dalam penelitiannya bahwa dalam
lintasan metabolisme pembentukan kandungan beta karoten pada buah, yang
berperan adalah enzim kunci yaitu lycopene β-cyclase, yang diduga dikendalikan
varietas tanaman tomat beta karoten rendah dengan varietas beta karoten tinggi
telah diteliti. Gen tunggal yang dinyatakan sebagai gen B, didapatkan untuk
pewarisan beta karoten berdasarkan analisis populasi F1, F2, dan Fa dari
persilangan tersebut. Gen ini diyakini berperan sebagai lycopene untuk
membentuk beta karoten. Gen-gen yang lain diasumsikan fungsinya sebagai
pengubah komponen yang lebih sederhana dari beta karoten seperti zeta karoten
dan phytofluene menjadi lycopene pada saat gen tersebut ada dalam keadaan
heterozigot atau homozigot dominan (Lincoln, 1950).
Rasid et al., (2003) yang melakukan penelitian di Malaysia berhasil
mendapatkan fragmen sekuen DNA sebesar 675 bp melalui RT-PCR yang
menyandi lycopene β-cyclase dari jaringan mesokarp kelapa sawit umur 17
minggu. Disebutkan di dalam penelitannya bahwa dugaan koding sekuen lycopene
β-cyclase kelapa sawit memiliki kesamaan 80% dengan lycopene β-cyclase dari
tanaman Narcisus pseudonarcissus, Sandersonia aurantiaca, Citrus sinensis dan
Capsicum annuum yang berperan dalam pembentukan beta karoten. Penelitian
amplifikasi RT-PCR oleh Rasid et al. (2008) pada E. oleifera telah berhasil
melakukan kloning koding sekuen partial DNA untuk phytoene synthase gene.
Rasid et al. (2011) dalam penelitiannya menemukan bahwa secara
keseluruhan pola ekpresi gen target lycopene β-cyclase (lcyb) dan
lycopene ε-cyclase (lcye) mengindikasikan beberapa tingkatan regulasi diferensial
dari gen-gen ini dipengaruhi oleh stadia perkembangan maupun tipe jaringan
tanaman kelapa sawit. Dalam jaringan mesokarp, lcyb terkespresi cukup tinggi
pada mesokarp muda (5 minggu setelah anthesis (MSA)). Tingkatan ekspresi lalu
lagi pada 17 dan 19 MSA hingga hampir menyamai tingkatan ekspresi gen pada 5
MSA. Sebaliknya tingkat ekspresi gen lcye rendah pada jaringan mesokarp di
semua stadia.
Primer Spesifik
Primer berfungsi sebagai penginisiasi reaksi polimerase DNA secara
in vitro. Tanpa primer reaksi polimerase DNA tidak akan terjadi meskipun enzim
dan komponen lainnya sedah tersedia. Selain itu, primer bertanggung jawab untuk
mengenali dan menandai fragmen sampel DNA (template DNA) yang akan
diamplifikasi (Zein dan Prawiradilaga, 2013).
Beberapa teknik analisis keanekaragaman genetik seperti RAPD, RFLP,
dan DGGE membutuhkan amplifikasi daerah genom tertentu dari suatu
organisme. Amplifikasi ini membutuhkan primer spesifik (sekuen oligonukelotida
khusus) untuk daerah tersebut. Primer biasanya terdiri dari 10 - 20 nukleotida dan
dirancang berdasarkan daerah konservatif dalam genom tersebut. Makin panjang
primer, makin harus spesifik daerah yang diamplifikasi. Jika suatu kelompok
organisme memang berkerabat dekat, maka primer dapat digunakan untuk
mengamplifikasi daerah tertentu yang sama dalam genom kelompok tersebut.
Beberapa faktor seperti konsentrasi DNA contoh, ukuran panjang primer,
komposisi basa primer, konsentrasi ion Mg, dan suhu hibridisasi primer harus
dikontrol dengan hati-hati agar dapat diperoleh pita-pita DNA yang utuh dan baik
(Suryanto, 2003).
ujung 3’-OH rantai DNA cetakan yang lain. Proses annealing biasanya dilakukan
selama 1 - 2 menit. Setelah dilakukan annealing oligonukleotida primer dengan
DNA cetakan, suhu inkubasi dinaikan menjadi 72oC selam 1,5 menit. Pada suhu
ini DNA polimerase akan melakukan proses polimerase rantai DNA yang baru
berdasarkan informasi yang ada pada DNA cetakan. Setelah terjadi polimerase,
rantai DNA akan membentuk jembatan hidrogen dengan DNA cetakan. DNA
rantai ganda yang terbentuk dengan adanya ikatan hidrogen antara rantai DNA
cetakan dengan rantai DNA yang baru hasil polimerase selanjutnya akan
didenaturasi lagi dengan menaikan suhu inkubasi menjadi 95oC. Rantai DNA
yang baru tersebut selanjutnya akan berfungsi sebagai cetakan bagi reaksi
polimerase berikutnya (Yuwono, 2006).
Memilih primer yang sesuai mungkin adalah faktor tunggal yang paling
penting mempengaruhi polymerase chain reaction (PCR). Amplifikasi spesifik
dari target yang diharapkan membutuhkan primer yang tidak dapat cocok dengan
target lainnya pada beberapa orientasi dan dalam jarak tertentu yang
memungkinkan terjadinya proses amplifikasi yang tidak diinginkan. Proses
mendesain primer spesifik pada umumnya melibatkan dua tahap. Pertama, daerah
apit primer yang diinginkan dapat diciptakan baik secara manual ataupun
menggunakan software tool; lalu dicari database sekuen nukleotida yang sesuai
menggunakan alat seperti BLAST untuk mengamati target-target potensial
PENDAHULUAN
Latar Belakang
Kelapa sawit adalah penghasil minyak nabati terbesar di dunia karena minyaknya
dapat diproduksi baik dari serabut buah maupun inti. Minyak tersebut dapat
digunakan sebagai minyak masak, minyak industri, maupun bahan bakar
(biodiesel). Keunggulannya yang lain diantaranya sifatnya yang tahan oksidasi
dengan tekanan tinggi dan kemampuannya melarutkan bahan kimia yang tidak
larut oleh bahan pelarut lainnya, serta daya melapis yang tinggi membuatnya
dapat digunakan untuk berbagai macam keperluan (Kemendag, 2013).
Peningkatan perhatian konsumen minyak nabati akan nilai nutrisi dan kesehatan
menjadi tuntutan permintaan masyarakat saat ini. Menurut Mangoensoekarjo dan
Semangun (2008), diantara jenis-jenis tanaman penghasil minyak nabati, kelapa
sawit termasuk tanaman yang potensi produksi minyaknya tertinggi. Sastrosayono
(2003) menyatakan potensi produksi minyak nabati kelapa sawit per hektar
mencapai 6 ton per tahun, bahkan lebih. Jika di bandingkan dengan tanaman
penghasil minyak lainnya (4,5 ton per tahun), tingkat produksi ini termasuk tinggi.
Melalui program-program pemuliaan yang mutakhir, potensi ini dapat
ditingkatkan lagi.
Kelapa sawit adalah tanaman penghasil minyak nabati yang dapat diandalkan,
karena minyak yang dihasilkan memiliki berbagai keunggulan dibandingkan
dengan minyak yang dihasilkan oleh tanaman lain. Minyak kelapa sawit
(Mangoensoekarjo dan Semangun, 2008) dan mengandung retinol 15 kali lebih
banyak dari wortel, 300 kali lebih banyak dari tomat
(Malaysian Palm Oil Board, 2003).
Karotenoid itu sendiri pada manusia berfungsi sebagai sumber vitamin A.
Karotenoid diasosiasikan dengan berbagai macam kegunaannya untuk kesehatan,
diantaranya sebagai antioksidan bagi tubuh, meningkatkan sistem imun,
mengurangi resiko beberapa jenis kanker dan penyakit kardiovaskular, juga
mengurangi resiko katarak (Olson, 1999).
Karakter kandungan beta karoten pada tanaman kelapa sawit memiliki nilai
heritabilitas yang tinggi. Berdasarkan penelitian Putri (2010) didapati bahwa nilai
duga heritabilitas arti luas karakter tersebut sebesar 84,8%. Dari hal ini dapat
disimpulkan bahwa karakter kandungan beta karoten pada tanaman kelapa sawit
lebih banyak dipengaruhi oleh faktor genetik ketimbang faktor lingkungan.
Pemuliaan konvensional memiliki beberapa keterbatasan, diantaranya
waktu yang diperlukan untuk memasukkan/introgresi gen-gen yang diinginkan
dan jumlah genotipe yang harus ditangani pada saat awal-awal seleksi yang besar
sehingga membutuhkan tenaga kerja dalam jumlah yang sangat besar. Oleh sebab
itu, diperlukan penggunaan teknologi baru untuk membantu meringankan
pekerjaan pemulia tanaman, salah satunya adalah penggunaan marka molekuler.
Penggunaannya dapat mempersingkat waktu yang dibutuhkan dalam melakukan
seleksi dan menentukan ap