HASIL DAN PEMBAHASAN
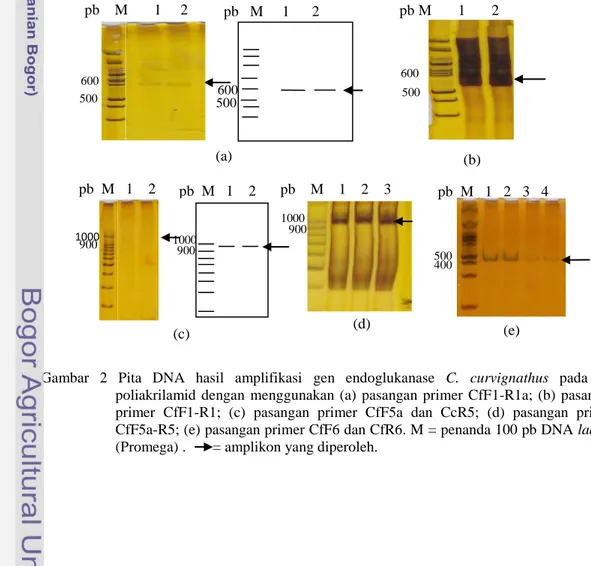
Amplifikasi dan Visualisasi DNA Endo-β-1,4- glukanase C. curvignathusAmplifikasi DNA ekson 1 dan 5 CcEG menggunakan pasangan primer eksternal (Tabel 2) dan masing-masing menunjukkan ukuran amplikon sekitar 600 dan 1000 pb (Gambar 2a dan 2c). Namun hasil PCR dengan menggunakan primer eksternal tersebut menunjukkan pita DNA yang tipis (Gambar 2a dan 2c). Pita DNA (amplikon) yang tipis menunjukkan konsentrasi DNA yang diperoleh masih rendah.
Hasil PCR ekson 1 dan 5 CcEG dengan konsentrasi amplikon yang rendah tersebut, kembali diamplifikasi dengan primer internal (Tabel 2) dan masing-masing menunjukkan ukuran amplikon sekitar 600 dan 1000 pb dengan konsentrasi yang tinggi (Gambar 2b dan 2d). Peningkatan konsentrasi ini terjadi karena sumber DNA merupakan hasil PCR dari amplifikasi primer eksternal yang telah dipurifikasi. Penggunaan primer internal yang dibuat di sebelah dalam primer eksternal
merupakan teknik yang disebut Nested
Polymerase Chain Reaction (Nested PCR).
Dengan Nested PCR, amplifikasi akan lebih spesifik pada DNA target, sehingga konsentrasi DNA meningkat (Lin et al. 2010).
Amplifikasi DNA ekson 6 CcEG meng-gunakan pasangan primer CfF6-R6 menunjukkan ukuran amplikon sekitar 500 pb. Namun amplikon menunjukkan variasi konsentrasi DNA (Gambar 2e). Hasil amplifikasi dengan peningkatan konsentrasi Mg2+ 1.5 mM menjadi 2.0 mM sampel yang sama (Cc 46 LSI), pada nomer 1 dan 2 menghasilkan konsentrsai DNA yang tinggi dibanding nomer 3 dan 4 (Gambar 2e) yang tanpa peningkatan konsentrasi Mg2+. Hal ini terjadi karena peningkatan konsentrasi Mg2+ mampu mengoptimasi amplifikasi karena pe-ngaruhnya dalam penempelan primer (Innis & Gefland 1990). Hasil sekuen ekson-intron 1, 5, dan 6 CcEG yang diperoleh berupa kromatrogram basa nukleotida (Lampiran 1 dan 2).
Gambar 2 Pita DNA hasil amplifikasi gen endoglukanase C. curvignathus pada gel poliakrilamid dengan menggunakan (a) pasangan primer CfF1-R1a; (b) pasangan primer CfF1-R1; (c) pasangan primer CfF5a dan CcR5; (d) pasangan primer CfF5a-R5; (e) pasangan primer CfF6 dan CfR6. M = penanda 100 pb DNA ladder (Promega) . = amplikon yang diperoleh.
(a) 500 600 pb M 1 2 600 500 pb M 1 2 (c) 1000 900 pb M 1 2 900 pb M 1 2 1000 (e) pb M 1 2 3 4 500 400 (d) 1000 pb M 1 2 3 900 (b) 600 500 pb M 1 2
5
Sekuen Ekson-Intron Gen Endo-β-1,4- glukanase C. curvignathus
Hasil sekuen ekson-intron 1, 5, dan 6 gen endo-β-1,4-glukanase C. curvignathus diedit dan di-alignment dengan sekuen CfEG3, RsEG, dan NtEG (Tabel 3). Ukuran gen endo-β-1,4-glukanase C. curvignathus yang diperoleh menggunakan pasangan primer CfF1a-R1 (Gambar 3a), CfF5-R5 (Gambar 3b), dan CfF6-R6 (Gambar 3c) yaitu masing-masing sebesar 594 (Gambar 4a, Tabel 4), 872 (Gambar 4b Tabel 4), dan 465 pb (Gambar 4c, Tabel 4). Total sekuen DNA CcEG yang didapat adalah 1931 pb.
Hasil sekuen ekson 5 CcEG yang diperoleh penelitian ini melengkapi sekuen ekson 5 CcEG penelitian Normasari (2011) dan melengkapi sekuen ekson 6 (Tabel 4 Lampiran 4).
Komposisi GC sekuen ekson CcEG sebesar 51.4% sedangkan sekuen intron sebesar 42.3%. Berdasarkan Alignment nukleotida juga diketahui Intron 1, 5, dan 6 CcEG diawali dengan basa GT (pada ujung 5’-intron) dan diakhiri dengan basa AG (pada ujung 3’-intron). Hasil ini seperti intron gen
amy dari Drosophila (Inomata et al. 1997) dan
intron 2 dan 4 CcEG (Normasari 2011).
Gambar 3 Contig gen endoglukanase C. curvignathus dengan menggunakan pasangan primer (a) CfF1-R1, (b)CfF5a-R5 dan (c) CfF6-R6. Huruf kapital = urutan basa ekson basa yang digaris bawah= primer. Basa nukleotida ambigu; D= A/G/T; H= A/C/T; K= G /T; M= A/C; R= A/G; S= C/G; W= A/T; Y= C/T
(a) CfR1 <<<<<<<<<<<<<<<<<<<< ATGAGGGTCTTCTTTTGCCTTCTGTCTGCGCTCGCGCTTTGCCAAGGTAACCAGTTCACCACTACACTTGG GTTAGAGTTCACTGTGGTAATTTCACACTATTCCCAACAGGATCACACACCTACAGCTATACTCACGAATA ACGCAAGTTCACCTAGTTATACGGATCAGAACCATAAGCTTACATTACCGTAACAGGTTGCTGCAAACTGT ACGATCTCCAAAGCCGCATACATTGTCTCTCTCGCTCTCTCTCTCTCTCACACACGCGCGCACGCACAGAA TAGAACAAAACCAACATCAATGGATACCATAGACAGGGATATAAATGCACCGGTTTATGACACAACGCCCG GATCGCGCTTCATGTTCGAAAATGCTGCAATTCCAAACTGAACACACACGGTAACTTGGCTCAATGGAAAT GTTATCCATTGTCGTTTCATGTCCTACACTTCATCATAAAGACACTCTCCGATGCATATTCCTACAAGGGG GAGACGTTCCCATAATGTGCCCTGTGTTACAGCTGCTTACGACTACAAGACAGTACTGAAGAATTCGCTGC TTTTCTACGAGGCTCAGCGATCGG CfF1 >>>>>>>>>>>>>>>>>>>>>>> (b) >>>>>>>>>>>>>>>>>>>>>>> > CfF6 CfR5 <<<<<<<<< CfF5a >>>>>>>>>>>>>>>>>>> CCAAGCAGCTTTTCGACTTCGCCAACAATTATCGCGGCAAATACAGCGATTCCATCACCGACGCGAAGAAT TTCTATGCGTAAGTGTACTACACTACCCACTCAATTTGCWATACGTACAGCGCTCTTACRCACGYCGSGCT GTCACMACTACCTAAACTYGGCGTTTAGAACTGCWTGAKGCTYGTACATTACACACACCTCCTTTGTGCGC AGGATCCTACRAATTACCACTCGGCCCAGCTGCCKGTCAATTACTCAGTCAGGCACATTCACATATYTAAA GGATACATATAAACACGTTGCAGTGATTACRGGGAAGTTTTACCCTGTTCCGTACGACTACGCTCTATCAT AATAAAAAAACATTAATTTHATCTTCACGTTTAADGTAATATCTTCCACGGGADGTATCACAGKGTGAAGG ATATCGCARACTTCTGCCGCAACATATGTYTACKGCATGACGTTACGTTTCATTCTTTCCACTGAAAACAA CTGTCATATAATGCACATCGCCCCTTTATTAGCCTCTCTTTAACGAATTTTGTGCAAAAGATTCAAATCTA CACAGTTATTCGTCCAGAACTGTGAGACATTTTTCTTCATTATGTGTTCCTGCAATGCCACACTCTGTACT CTGCTACAAATATATTCACACAGAGGAGCAGACTACCGCCTGCATACTCAGTACTATGTTCCTAGCACGAT AAATCTGATACGCTGAATGGCATCACAACGTCAAAATCTCCATATTCACCACCGTCACAAACACAAATCAA CCATTTCTAATCATCTGATGTAGGTCTGGAGACTACAAGGATGAGTTAGTATGGGCAGCCGCATGGCTCTA CAGGGCGACCA (c) AGTATGGGCAGCCGCATGGCTCTACAGGGCGACCAACGACAACGCCTATCTCACTAAAGCTGAGTCGCTGT ACAACGAATTCGGCCTTGGAAGCTGGAATGGTGCCTTCAACTGGGATAACAAGATCTCCGGTGTACAGGTC AGACACGTGAAGAAATTCATGCTTACAAAAGTCACGGACAAAACAACAAAGCTACAGATATTAATACATAC CACAAGCCACACAATCACTGTGTCAGTTTCAGTCACTAGCTGAGTGATAGTTCTTCAGCCATGGAAGTGTA GGCGTCATACAGTAATAATCATTCACAAAAGAAAACTGAAAATCCTGGACAATATAGGTGCCGAAACAGCA ATAGGCACCATGTTCCTAAACGTTTCGCCCTCTATCATATTTACTTGAACTTCCACAGGTTCTACTGGCCA AGCTCACAAGCAAGCAGGCATACAAGGACAAGGTACAGG CfF6 >>>>>> CfR6 <<<<<<<<<<<<<<<<<<<<< CcR5
<<<<<<<<<<<<<<<<<<<
CfR5 <<<<<<<<<<<<<<<<<<<<Tabel 4 Panjang sekuen ekson-intron 1, 5, dan 6 endo-β-1,4-glikanase C. curvignathus No. Sekuen
ekson
Primer yang digunakan Total sekuen (pb) Panjang ekson (pb) Panjan g intron (pb) Informasi tambahan 1 1 CfF1-R1 (primer internal) 592 46 483 Overlap 61 pb
intron 2*
2 5 CfF5a-R5(primer internal) 863 79 725 Overlap 26 pb
ekson 5** dan 35 pb ekson 6*** 3 6 CfF6-R6 (primer eksternal) 465 139 274 Overlap 52 pb ekson 7**** *) Overlap sekuen 1 CcEG dengan sekuen CcEG ekson 2 Normasari (2011); ** Overlap hasil 2 CcEG dengan CcEG ekson 5 Normasari (2011); *** Overlap hasil 2 CcEG dengan sekuen hasil ekson 6 CcEG penelitian ini, **** Overlap hasil 3 CcEG dengan sekuen CcEG ekson 7 Nurgianti (2011) (Lampiran 4)
Gambar 4 Sekuen ekson-intron 1 (a), 5 dan 6 (b) gen endoglukanase C. curvignathus. Huruf kapital = urutan basa ekson; huruf kecil = urutan basa intron; basa dengan latar kuning = awal dan akhir intron; basa yang dengan garis bawah = overlap antar
contig. Basa nukleotida ambigu; Basa nukleotida ambigu; D= A/G/T; H= A/C/T;
K= G /T; M= A/C; R= A/G; S= C/G; W= A/T; Y= C/T .
Intron 1
Ekson 1
Ekson 2
ATGAGGGTCT TCTTTTGCCT TCTGTCTGCG CTCGCGCTTT GCCAAGgtaa 50 ccagttcacc actacacttg ggttagagtt cactgtggta atttcacact 100 attcccaaca ggatcacaca cctacagcta tactcacgaa taacgcaagt 150 tcacctagtt atacggatca gaaccataag cttacattac cgtaacaggt 200 tgctgcaaac tgtacgatct ccaaagccgc atacattgtc tctctcgctc 250 tctctctctc tcacacacgc gcgcacgcac agaatagaac aaaaccaaca 300 tcaatggata ccatagacag ggatataaat gcaccggttt atgacacaac 350 gcccggatcg cgcttcatgt tcgaaaatgc tgcaattcca aactgaacac 400 acacggtaac ttggctcaat ggaaatgtta tccattgtcg tttcatgtcc 450 tacacttcat cataaagaca ctctccgatg catattccta caagggggag 500 acgttcccat aatgtgccct gtgttacagC TGCTTACGAC TACAAGACAG 550 TACTGAAGAA TTCGCTGCTT TTCTACGAGG CTCAGCGATC GG 592
(a)
(b)Intron 5
Ekson 5
Ekson 6
Intron 6 Ekson 7CCAAGCAGCT TTTCGACTTC GCCAACAATT ATCGCGGCAA ATACAGCGAT 50 TCCATCACCG ACGCGAAGAA TTTCTATGCg taagtgtact acactaccca 100 ctcaatttgc watacgtaca gcgctcttac rcacgycgsg ctgtcacmac 150 tacctaaact yggcgtttag aactgcwtga kgctygtaca ttacacacac 200 ctcctttgtg cgcaggatcc tacraattac cactcggccc agctgcckgt 250 caattactca gtcaggcaca ttcacataty taaaggatac atataaacac 300 gttgcagtga ttacrgggaa gttttaccct gttccgtacg actacgctct 350 atcataataa aaaaacatta attthatctt cacgtttaad gtaatatctt 400 ccacgggadg tatcacagkg tgaaggatat cgcaracttc tgccgcaaca 450 tatgtytack gcatgacgtt acgtttcatt ctttccactg aaaacaactg 500 tcatataatg cacatcgccc ctttattagc ctctctttaa cgaattttgt 550 gcaaaagatt caaatctaca cagttattcg tccagaactg tgagacattt 600 ttcttcatta tgtgttcctg caatgccaca ctctgtactc tgctacaaat 650 atattcacac agaggagcag actaccgcct gcatactcag tactatgttc 700 ctagcacgat aaatctgata cgctgaatgg catcacaacg tcaaaatctc 750 catattcacc accgtcacaa acacaaatca accatttcta atcatctgat 800 gtagGTCTGG AGACTACAAG GATGAGTTAG TATGGGCAGC CGCATGGCTC 850 TACAGGGCGA CCAACGACAA CGCCTATCTC ACTAAAGCTG AGTCGCTGTA 900 CAACGAATTC GGCCTTGGAA GCTGGAATGG TGCCTTCAAC TGGGATAACA 950 AGATCTCCGG TGTACAGgtc agacacgtga agaaattcat gcttacaaaa 1000 gtcacggaca aaacaacaaa gctacagata ttaatacata ccacaagcca 1050 cacaatcact gtgtcagttt cagtcactag ctgagtgata gttcttcagc 1100 catggaagtg taggcgtcat acagtaataa tcattcacaa aagaaaactg 1150 aaaatcctgg acaatatagg tgccgaaaca gcaataggca ccatgttcct 1200 aaacgtttcg ccctctatca tatttacttg aacttccaca gGTTCTACTG 1250 GCCAAGCTCA CAAGCAAGCA GGCATACAAG GACAAGGTAC AGG 1293
7
Analisis Homologi Nukleotida dengan BLASTN Gen Endo-β-1,4- glukanase Rayap C. curvignathus
Hasil alignment gen endo-β-1,4-glukanase parsial C. curvignathus (CcEG) dengan C.
formosanus (CfEG3), R. speratus (RsEG), dan N. takasagoensis (NtEG) menunjukkan posisi
ekson 1, 5, dan 6 CcEG berada pada ekson 1, 5, dan 6 CfEG dan RsEG, sedangkan pada NtEG berada pada ekson 2, 6, dan 7. Ukuran panjang ekson 5, 6 CcEG sama dengan ekson 5, 6 CfEG3 dan RsEG serta ekson 6 dan 7 NtEG (Gambar 5). Panjang Ekson 1 CcEG sama dengan panjang CfEG, namun berbeda dengan ekson 1 RsEG dan ekson 2 NtEG (Gambar 5). Hasil alignment ekson 1, 5, dan 6 CcEG dengan CfEG, NtEG, dan RsEG menunjukkan perbedaan basa paling sedikit dengan CfEG dibandingkan dengan NtEG dan RsEG (Lampiran 3).
Hasil analisis homologi nukleotida dengan BLASTN ekson 1, ekson 5 dan ekson 6 gen endoglukanase parsial C. curvignathus
homolog dengan CfEG (Tabel 5). Sekuen ekson 1,5, dan 6 CcEG secara keseluruhan menghasilkan persen homolog 98% dengan C.
formosanus (Tabel 5). Kedua rayap ini
memiliki genus yang sama yaitu Coptotermes (Ahmad 1958).
Analisis Homologi asam amino dengan BLASTP, Jarak Genetik dan Struktur protein Endo-β-1,4-glukanase Parsial Ra-yap C. curvignathus
Hasil deduksi sekuen ekson 1, 5, dan 6 rayap C. curvignathus gen endo-β-1,4-glukanase parsial menghasilkan sebanyak 95 asam amino putative dengan asam amino pertama adalah metionin (Gambar 6). Metionin merupakan asam amino hasil dari translasi kodon start (kodon penginisiasi awal) berupa basa ATG di utas DNA atau AUG di RNA (Griffiths et al. 1999). Hasil analisis homologi asam amino putative ekson 1, 5, dan 6 endoglukanase parsial C.
curvignathus melalui BLASTp homolog 94%
dengan asam amino putative C. formosanus (Tabel 6). Hasil alignment asam amino ekson 1, 5, dan 6 CcEG dengan CfEG3, RsEG, dan NtEG memperlihatkan bahwa asam amino
putative CcEG memilki perbedaan yang
paling sedikit dengan CfEG dibanding dengan RsEG dan NtEG, yaitu 1 asam amino berbeda (Gambar 7).
Perhitungan jarak genetik dan pembuatan pohon filogeni berdasarkan asam amino
putative gen endo-β-1,4-glukanase parsial C. curvignathus ekson 1, 5 dan 6 menunjukan
CcEG lebih dekat dengan asam amino putative gen endoglukanase C. formosanus jika dibandingkan dengan asam amino putative gen endoglukanase N. takasagoensis dan R.
speratus dilihat dari nilai jarak terkecil yaitu
sebesar 0,011 (Tabel 7 dan Gambar 8). Nilai jarak genetik yang terjauh dari asam amino
putative gen endo-β-1,4-glukanse parsial C. curvignathus adalah asam amino putative dari N. takasagoensis yaitu sebesar 0.168 (Tabel
7). Hal ini menunjukkan bahwa C. curvignathus lebih dekat kekerabatannya
dengan C. formosanus dibandingkan dengan
N. takasagoensis. Rayap C. curvignathus dan C. formosanus termasuk ke dalam famili
Rhinotermitidae sedangkan N. takasagoensis termasuk ke dalam famili yang berbeda yaitu Termitideae (Ahmad 1958).
Struktur tiga dimensi endoglukanase parsial dibuat dengan program Cn3D. Struktur 3 dimensi yang terbentuk terdiri dari helix alpa dan lembar beta (Gambar 9). Analisis asam amino putative dengan BLASTp juga menunjukkan asam amino
putative CcEG termasuk dalam kelompok Glycosyl Hydrolase Family 9 (GHF9) karena
homolog 99% dengan GHF 9 C. formosanus (Lampiran 5). Hal ini memperlihatkan kemiripan tingkat asam amino putative dari CcEG terhadap NtEG, CfEG dan RsEG yang juga termasuk dalam GHF 9 (Tokuda et al. 1999, Nakashima et al. 2001)
Rayap C. curvignathus berkerabat dekat dengan C. formosanus, R. speratus karena berasal dari famili yang sama yaitu Rhinotermitidae dibandingkan dengan N.
takasagoensis yang bersal dari famili
Termitideae. Namun C. curvignathus lebih dekat dengan C. formosanus dibandingkan dengan R. speratus karena berasal dari genus yang sama, yaitu Coptotermes (Ahmad 1958). Rayap famili Rhinotermitidae merupakan rayap tingkat rendah sedangkan rayap famili Termitidae merupakan rayap tingkat tinggi (Khrisna & Weesner 1970). Endoglukanase rayap diekspresikan pada saluran pencernaan rayap. Ekspresi endoglukanase (EG) pada rayap tingkat tinggi dan rayap tingkat rendah memiliki perbedaan. Ekspresi EG N. takasagoensis (NtEG) yang termasuk rayap
tingkat tinggi terjadi di usus tengah. Sedangkan ekspresi EG pada rayap tingkat rendah berbeda pada tiap spesies. Ekspresi EG rayap tingkat rendah seperti RsEG terjadi di kelenar saliva/usus depan dan CfEG terjadi di saliva usus depan dan usus tengah (Tokuda et
Gambar 6 Skema posisi ekson dan intron NtEG (AB019146), RsEG (AB008778), CfEG (AB058670), dan gen endoglukanase C. curvignathus (hasil penelitian ini). = ekson ; = intron. Angka di bawah kotak menunjukkan posisi ekson dan angka di atas segitiga menunjukkan posisi intron. Angka di dalam kotak dan segitiga menunjukkan panjang basa; tanda ( o)= Studi ini, tanda (*) = Normasari 2011; (**)= Nurgianti
N. takasagoens (Nakashima et al. 2001) 1467 1183 933 454 247 832 276 1682 1942 1 2 3 4 5 6 7 8 9
1
2
3
4
5
6
7
8
9
10
59 108 162 96 183178
163
165 140 431 R. speratus (Tokuda et al.1999)1
2
3
4
5
6
7
8
9
58 162 96 183 178 163 165 140 320 C. formosanus (Tokuda et al.1999)1
2
3
4
5
6
7
8
9
46 162 96 183 178 163 165 140 2141
2
3
4
5
6
7
8
9
46o 162* 96* 183* 125* 79o 163o 165** 140** 214** 118* 842* 187* 483o 725 o 503** 546** 274o C. curvignathus8
9
No
. Nukleotida Deskripsi Acc number
Persen Homologi (%) E-value 1 Ekson 1 CcEG C. formosanus
endo-beta-1,4-glucanase (EG3a-1) gene, partial cds
HQ698632.1 98 6e-14 2 Ekson 5
CcEG
C. formosanus CfEG4 mRNA
for endo-b-1,4-glucanase, complete cds AB058671.1 99 5e-31 3 Ekson 6 CcEG C.formosanus
endo-beta-1,4-glucanase mRNA, complete cds EU853671.1 98 6e-74 4 Ekson 1,5,
dan 6
C. formosanus CfEG4 mRNA
for endo-b-1,4-glucanase, complete cds
AB058671.1 98 5e-117
9
Tabel 5 Hasil analisis homologi berdasarkan nukleotida (BLAST N) gen endo-β-1,4-glukanase rayap C. curvignathus 10 20 30 40 50 60 ATGAGGGTCTTCTTTTGCCTTCTGTCTGCGCTCGCGCTTTGCCAAGCCAAGCAGCTTTTC M R V F F C L L S A L A L C Q K Q L F 70 80 90 100 110 120 GACTTCGCCAACAATTATCGCGGCAAATACAGCGATTCCATCACCGACGCGAAGAATTTC D F A N N Y R G K Y S D S I T D A K N F 130 140 150 160 170 180 TATGCGTCTGGAGACTACAAGGATGAGTTAGTATGGGCAGCCGCATGGCTCTACAGGGCG Y A S G D Y K D E L V W A A A W L Y R A 190 200 210 220 230 240 ACCAACGACAACGCCTATCTCACTAAAGCTGAGTCGCTGTACAACGAATTCGGCCTTGGA T N D N A Y L T K A E S L Y N E F G L G 250 260 270 280 290 AGCTGGAATGGTGCCTTCAACTGGGATAACAAGATCTCCGGTGTACAG S W N G A F N W D N K I S G V Q
Gambar 7 Asam amino putative ekson 1, 5, dan 6 gen endo-β-1,4-glukanase rayap C.
curvignathus. = kodon start (ATG) dan metionin (M), Angka di baris ke-1 = jumlah
nukleotida; huruf di baris ke-2 = nukleotida; huruf di baris ke-3 = asam amino putative
C. curvignathus MRVFFCLLSALALCQKQLFDFANNYRGKYSDSITDAKNFYASGDYKDELVWAAAWLYRAT 60 C. formosanus ... 60 R. speratus .K..V...Q... 60 N. takasagoensis ....L...R...R...A..R... 60 C. curvignathus NDNAYLTKAESLYNEFGLGSWNGAFNWDNKISGVQ 95 C. formosanus ...T... 95 R. speratus ...T...N... 95 N. takasagoensis ...T..NT...D....QN.G.GL...S.V.... 95
Gambar 8 Alignment Asam amino putative ekson 1, 5, dan 6 gen endo-β-1,4-glukanase rayap C.
C. curvignathus C. formosanus R. speratus N. takasagoensis C. curvignathus C. formosanus 0.011 R. speratus 0.053 0.042 N. takasagoensis 0.168 0.158 0.158 CcEG 156 CfEG3 156 RsEG 156 NtEG 156 97 0.02 No. Asam amino putative Deskripsi Jumlah asam amino
Acc number Kehomologian
(%) E-value 1 Ekson 1 Endo-beta-1,4-glukanase C. formosanus 15 ADV16268.1 100 6e-8 2 Ekson 5 Endogenous cellulase R. flavipes 25 AAU20853.2 100 8e-18 3 Ekson 6 Endo-β-1.4-glukanase C. formosanus 54 BAB40695.1 98 2e-28 4 Ekson 1, 5, 6 Endo-β-1.4-glukanase C. formosanus 95 BAB40695.1 94 6e-47
Tabel 7 Jarak genetik asam amino putative gen endoglukanase C. curvignathus ekson 1, 5 dan 6 dengan asama amino putative C. formosanus, R. speratus dan N. takasagoensis
Tabel 6 Hasil analisis homologi berdasarkan asam amino putative (BLAST P) ekson 1, 5 dan 6 gen endo-β-1,4-glukanase rayap C. curvignathus
A
B
Gambar 10 Struktur 3 dimensi endo-β-1,4-glukanase parsial rayap C. curvignathus dari asam amino putative 5 dan 6. A = helix alpa; B = lembar beta.
Gambar 9 Pohon filogeni Endo-β-1,4-glukanase parsial rayap C. curvignathus (CcEG terhadap asam amino putative C. formosanus (CfEG), R. speratus (RsEG), dan N.