IDENTIFIKASI GEN SITOKROM B (Cyt b) BABI HUTAN PADA KORNET SAPI DI YOGYAKARTA DENGAN METODE POLYMERASE
CHAIN REACTION
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm)
Program Studi Farmasi
Oleh :
Laurensia Kusumaningtyas Theodorus NIM: 178114026
FAKULTAS FARMASI UNIVERSITAS SANATA DHARMA
YOGYAKARTA 2021
ii
IDENTIFIKASI GEN SITOKROM B (Cyt b) BABI HUTAN PADA KORNET SAPI DI YOGYAKARTA DENGAN METODE POLYMERASE
CHAIN REACTION
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm)
Program Studi Farmasi
Oleh :
Laurensia Kusumaningtyas Theodorus NIM: 178114026
FAKULTAS FARMASI UNIVERSITAS SANATA DHARMA
YOGYAKARTA 2021
iii
Persetujuan Pembimbing
IDENTIFIKASI GEN SITOKROM B (Cyt b) BABI HUTAN PADA KORNET SAPI DI YOGYAKARTA DENGAN METODE POLYMERASE
CHAIN REACTION
Skripsi yang diajukan oleh : Laurensia Kusumaningtyas Theodorus
NIM : 178114026
telah disetujui oleh
Pembimbing Utama
iv
Pengesahan Skripsi Berjudul
IDENTIFIKASI GEN SITOKROM B (Cyt b) BABI HUTAN PADA KORNET SAPI DI YOGYAKARTA DENGAN METODE POLYMERASE
CHAIN REACTION
Oleh :
Laurensia Kusumaningtyas Theodorus 178114026
Dipertahankan di hadapan Panitia Penguji Skripsi Fakultas Farmasi
Universitas Sanata Dharma Pada tanggal: 15 Juli 2021
Mengetahui Fakultas Farmasi Universitas Sanata Dharma
Dekan
(Dr. apt. Yustina Sri Hartini)
Panitia Penguji Tanda Tangan
1. Damiana Sapta Candrasari, S.Si., M.Sc. ………... 2. Dr. apt. Christine Patramurti ………...
v
PERNYATAAN KEASLIAN KARYA
Saya menyatakan dengan sesungguhnya bahwa skripsi yang saya tulis ini tidak memuat karya atau bagian karya orang lain, kecuali yang telah disebutkan dalam kutipan dan daftar pustaka, dengan mengikuti ketentuan sebagaimana layaknya karya ilmiah.
Apabila di kemudian hari ditemukan indikasi plagiarisme dalam naskah ini, maka saya bersedia menanggung segala sanksi sesuai peraturan perundang-undangan yang berlaku.
Yogyakarta, 15 Juni 2021 Penulis
vi
LEMBAR PERNYATAAN PERSETUJUAN
PUBLIKASI KARYA ILMIAH UNTUK KEPENTINGAN AKADEMIS Yang bertanda tangan di bawah ini, saya mahasiswa Universitas Sanata Dharma :
Nama : Laurensia Kusumaningtyas Theodorus Nomor Mahasiswa : 178114026
Demi pengembangan ilmu pengetahuan, saya memberikan kepada Perpustakaan Universitas Sanata Dharma karya ilmiah saya yang berjudul :
“Identifikasi Gen Sitokrom b (Cyt b) Babi Hutan Pada Kornet Sapi Di Yogyakarta Dengan Metode Polymerase Chain Reaction”
beserta perangkat yang diperlukan (bila ada). Dengan demikian saya memberikan kepada Perpustakaan Universitas Sanata Dharma hak untuk menyimpan, me-ngalihkan dalam bentuk media lain, mengelolanya dalam bentuk pangkalan data, mendistribusikan secara terbatas, dan mempublikasikannya di Internet atau media lain untuk kepentingan akademis tanpa perlu meminta ijin dari saya maupun memberikan royalti kepada saya selama tetap mencantumkan nama saya sebagai penulis.
Demikian pernyataan ini yang saya buat dengan sebenarnya. Dibuat di Yogyakarta
Pada tanggal : 23 Juli 2021 Yang menyatakan
vii ABSTRAK
Daging babi hutan merupakan bahan pangan hewani yang diharamkan bagi umat muslim dan menjadi masalah di Negara Indonesia karena dicampur dengan daging sapi khususnya dalam suatu produk olahan daging sapi seperti kornet. Hal ini bermasalah karena produk olahan tersebut tidak dapat dibedakan secara makroskopis. Penelitian ini bertujuan untuk mengidentifikasi ada atau tidaknya kandungan babi hutan menggunakan marker sitokrom b DNA Mitokondria pada produk kornet sapi di Yogyakarta dengan metode Polymerase Chain Reaction (PCR). PCR merupakan teknik amplifikasi potongan DNA (Deoxyribonucleic Acid) yang diinginkan secara in vitro pada daerah spesifik yang dibatasi oleh dua buah primer oligonukleotida dengan bantuan enzim polymerase.
Penelitian ini merupakan penelitian deskriptif kualitatif dengan teknik purposive sampling. Isolasi DNA dilakukan pada subyek uji yakni 5 sampel produk kornet sapi dengan merk berbeda yang diperoleh dari supermarket wilayah Yogyakarta. Analisis produk PCR menggunakan teknik elektroforesis. Hasil penelitian menunjukkan bahwa proses amplifikasi tidak terjadi pada fragmen DNA berukuran 398 bp pada lima sampel kornet sapi sehingga dapat disimpulkan bahwa kandungan gen sitokrom b babi hutan (Sus scrofa) tidak teridentifikasi pada kelima sampel kornet sapi yang diperoleh dari supermarket wilayah Yogyakarta.
viii ABSTRACT
Wild boar meat is an animal food that is prohibited by Muslims and is a problem in Indonesia because it is mixed with beef, especially in processed beef products such as corned beef. This is problematic because the processed product cannot be differentiated macroscopically. This study aims to identify the presence or absence of wild boar using Mitochondrial DNA cytochrome b markers in corned beef products in Yogyakarta with the Polymerase Chain Reaction (PCR) method. PCR is a technique of amplification of the desired DNA (Deoxyribonucleic Acid) fragments in vitro in a specific area that is bounded by two oligonucleotide primers with the help of polymerase enzyme.
This research is a qualitative descriptive study with purposive sampling technique. DNA isolation was carried out on test subjects, namely 5 samples of corned beef products with different brands obtained from supermarkets in Yogyakarta area. Analysis of PCR products using electrophoresis techniques. The results showed that the amplification process did not occur in the 398 bp DNA fragment in five samples of corned beef, so it could prove that the content of the cytochrome b gene of wild boar (Sus scrofa) was not identified in five samples of corned beef obtained from supermarkets in Yogyakarta.
ix DAFTAR ISI
HALAMAN COVER ... i
HALAMAN JUDUL ... ii
HALAMAN PERSETUJUAN PEMBIMBING ... iii
HALAMAN PENGESAHAN ... iv
PERNYATAAN KEASLIAN KARYA ... v
LEMBAR PERNYATAAN PERSETUJUAN ... vi
ABSTRAK ... vii
ABSTRACT ... viii
DAFTAR ISI ... ix
DAFTAR TABEL ... xi
DAFTAR GAMBAR ... xii
DAFTAR LAMPIRAN ... xiii
BAB I ... 1 A. Latar Belakang ... 1 B. Rumusan Masalah ... 3 C. Keaslian Penelitian ... 3 D. Tujuan Penelitian ... 4 E. Manfaat Penelitian ... 4 BAB II ... 5 A. Kornet ... 5
B. Babi hutan (Sus scrofa) ... 5
C. DNA Mitokondria ... 6
D. Gen Sitokrom b ... 7
E. Polymerase Chain Reaction (PCR) ... 8
F. Landasan Teori ... 10
G. Hipotesis ... 11
BAB III... 12
A. Jenis dan Rancangan Penelitian ... 12
B. Variabel dan Definisi Operasional ... 12
C. Bahan penelitian ... 13
D. Alat Penelitian ... 14
E. Tata Cara Penelitian ... 14
F. Analisis Hasil ... 18
BAB IV ... 19
A. Optimasi Preparasi Sampel ... 19
B. Isolasi DNA ... 20
C. Analisis Kualitatif Isolat DNA... 22
D. Identifikasi Gen Sitokrom b... 23
E. Analisis Produk PCR ... 27
BAB V ... 31
x
B. Saran ... 31
DAFTAR PUSTAKA ... 32
LAMPIRAN ... 36
xi
DAFTAR TABEL
Tabel I. Kondisi PCR ... 17 Tabel II. Fragmen DNA hasil produk PCR ... 28
xii
DAFTAR GAMBAR
Gambar 1. Struktur DNA Mitokondria (Butler, 2005) ... 7
Gambar 2. Wilayah Target Gen Sitokrom b Babi Hutan (NCBI, 2013) ... 8
Gambar 3. Hasil Elektroforesis Isolat DNA Sampel Tanpa Perlakuan ... 20
Gambar 4. Hasil Elektroforesis Isolat DNA Sampel dengan Perlakuan ... 20
Gambar 5. Wilayah Target Gen Sitokrom b Sapi (NCBI, 2009) ... 24
Gambar 6. Hasil Optimasi Suhu Annealing Produk PCR Kontrol Daging Babi .. 26
Gambar 7. Hasil Optimasi Suhu Annealing Produk PCR Kontrol Daging Sapi ... 26
Gambar 8. Hasil Analisis Kualitatif Produk PCR dengan Primer Reverse Sapi ... 27
xiii
DAFTAR LAMPIRAN
Lampiran 1. Perhitungan Jumlah Sampel ... 37
Lampiran 2. Hasil elektroforesis isolat DNA ... 38
Lampiran 3. Hasil elektroforesis Produk PCR ... 39
Lampiran 4. Informasi Pemakaian FavorPrep™ ... 40
Lampiran 5. Informasi Produk DNA Ladder ... 43
Lampiran 6. Informasi Produk Nucleic Acid Gel Stain ... 45
1 BAB I PENDAHULUAN
A. Latar Belakang
Indonesia merupakan negara dengan mayoritas penduduknya beragama Islam dan kehalalan adalah persyaratan mutlak bagi setiap muslim untuk mengkonsumsi makanan (Zulfahmi, 2015). Menurut Undang-Undang Republik Indonesia Nomor 33 Tahun 2014 Pasal 17 dan 18 dinyatakan bahwa dalam Proses Produk Halal (PPH) bahan baku, bahan olahan, bahan tambahan, dan bahan penolong yang berasal dari bangkai, darah dan babi diharamkan atau dilarang untuk dikonsumsi umat Islam. Dengan demikian, seluruh produk pangan yang mengandung unsur babi di dalamnya diharamkan dalam Islam untuk dimakan, padahal sejumlah produk makanan berbahan dasar daging ditemukan tercampur dengan daging babi, terutama pada makanan olahan daging sapi seperti kornet (Puspitasari dkk., 2019). Kandungan daging babi pada produk olahan seperti kornet menjadi susah karena tidak dapat dibedakan secara makroskopis (Andriyani dkk., 2019). Hal ini terjadi karena sering kali produk olahan tersebut sudah dihancurkan dan dicampur dengan berbagai bahan lain (Puspitasari dkk., 2019).
Menurut Fibriana dkk. (2012) kasus makanan berbahan dasar daging dicampur dengan babi marak terjadi di Indonesia sejak tahun 80-an sampai sekarang. Pencampuran daging babi tersebut bertujuan untuk menurunkan harga produksi menjadi relatif lebih murah dibandingkan jika menggunakan bahan daging sapi asli. Penggantian daging sapi dengan daging babi sebagai bahan dasar atau bahan tambahan tersebut sebagian besar tidak diinformasikan kepada konsumen. Oleh karena itu, deteksi cemaran babi pada produk makanan perlu dilakukan untuk melindungi konsumen (Puspitasari dkk., 2019). Salah satu jenis metode pendeteksian yang umum dilakukan dalam mendeteksi komponen babi dan turunannya adalah metode berbasis DNA yaitu PCR (Polymerase Chain Reaction) (Fadhlurrahman dkk., 2015) baik konvensional PCR, Multipleks PCR, maupun yang terbaru adalah Real Time PCR (Rachmawati dkk., 2018).
Penelitian yang dilakukan (Wijaya dkk., 2018) menemukan bahwa tiga merk kornet sapi yang diperoleh dari supermarket di sekitar wilayah Kedungmudu negatif mengandung babi menggunakan PCR. Berikutnya, ada pula penelitian yang dilakukan (Rahayu dkk., 2020) yaitu satu produk kornet sapi dan satu dendeng dari total empat sampel (dua produk kornet sapi dan dua produk dendeng) diperoleh dari market ditemukan mengandung babi hutan dilihat dari nilai Ct (threshold cycle) dibawah 30 menggunakan Real Time PCR. Teknik PCR dipilih sebagai alat identifikasi karena mempunyai akurasi tinggi dalam mendeteksi ada tidaknya campuran daging babi dalam daging segar maupun produk daging olahan (Wardani and Sari, 2015).
PCR merupakan teknik amplifikasi potongan DNA yang diinginkan secara in vitro pada daerah spesifik yang dibatasi oleh dua buah primer oligonukleotida dengan bantuan enzim polymerase (Wardani and Sari, 2015). Penggunaan DNA mitokondria (mtDNA) dalam analisis PCR dapat meningkatkan sensitivitas karena setiap sel memiliki sekitar seribu mitokondria dan setiap mitokondria memiliki sepuluh salinan DNA (Jain dkk., 2007). Gen-gen yang paling sering digunakan sebagai penanda jenis hewan atau daging diantaranya adalah sitokrom b (cyt b), 12S dan 16S subunit ribosom RNA dan daerah displacement loop (D-loop). Gen sitokrom b yang dipilih pada penelitian ini karena gen sitokrom b dapat digunakan untuk membedakan material yang berasal dari jenis hewan yang berbeda. Kekhasan dari gen sitokrom b diantaranya yaitu adanya daerah yang hampir sama untuk semua jenis hewan tetapi juga terdapat daerah yang spesifik untuk setiap jenis hewan. Kedua daerah tersebut berada dalam satu gen sehingga dalam penggunaannya untuk membedakan beberapa jenis hewan relatif lebih akurat (Primasari, 2011). Hal ini yang membuat peneliti tertarik untuk melakukan penelitian tentang identifikasi gen sitokrom b babi hutan (Sus scrofa) pada kornet sapi di Yogyakarta dengan metode Polymerase Chain Reaction.
B. Rumusan Masalah
Apakah terdapat kandungan gen sitokrom b babi hutan (Sus scrofa) pada produk kornet sapi di Yogyakarta dengan metode Polymerase Chain Reaction (PCR)?
C. Keaslian Penelitian
Terdapat beberapa penelitian terkait kandungan gen sitokrom b babi hutan (Sus scrofa) dengan metode Polymerase Chain Reaction (PCR) yang telah dilakukan para peneliti sebelumnya, yakni sebagai berikut :
1) Penelitian yang dilakukan oleh Fitrilia dkk. (2017) dengan judul “Deteksi Kandungan Gen Sitokrom b (Cyt b) Babi pada Bakso yang Beredar di Kota Jambi Menggunakan Teknik Polymerase Chain Reaction”. Penelitian ini bertujuan untuk mendeteksi adanya kandungan gen sitokrom b babi bakso yang beredar di kota Jambi. Hasil penelitian menunjukkan bahwa sampel bakso yang beredar di Kota Jambi yang dideteksi menggunakan teknik Polymerase Chain Reaction tidak ditemukan gen sitokrom b sebagai penanda gen babi.
2) Penelitian yang dilakukan oleh Wijaya dkk. (2018) dengan judul “Deteksi Daging Babi Pada Tiga Merk Kornet Sapi Berdasarkan Gen Cytochrome b dengan Metode PCR”. Penelitian ini bertujuan untuk mengidentifikasi campuran daging babi pada tiga macam merk kornet sapi berdasarkan primer spesifik P14. Hasil penelitian menunjukkan bahwa tiga merk kornet sapi yang diperoleh dari supermarket di sekitar wilayah Kedungmudu negatif mengandung babi.
Berdasarkan hasil penelusuran pustaka, penelitian mengenai identifikasi gen sitokrom b babi hutan pada produk kornet sapi di Yogyakarta dengan metode Polymerase Chain Reaction (PCR) belum pernah dilakukan. Penelitian ini berbeda dari penelitian sebelumnya dalam hal subyek penelitian dan tempat pengambilan sampel.
D. Tujuan Penelitian
Tujuan dari penelitian ini adalah untuk mengidentifikasi kandungan gen sitokrom b babi hutan (Sus scrofa) pada produk kornet sapi di Daerah Istimewa Yogyakarta dengan metode Polymerase Chain Reaction (PCR).
E. Manfaat Penelitian 1. Manfaat Teoritis
Penelitian ini diharapkan dapat memberikan informasi tambahan dan memperkaya pengetahuan ilmiah terutama di bidang kefarmasian tentang identifikasi gen sitokrom b babi hutan pada produk kornet sapi di Daerah Istimewa Yogyakarta dengan metode Polymerase Chain Reaction (PCR).
2. Manfaat Praktis
Penelitian ini diharapkan dapat menjadi bahan referensi bagi praktek kefarmasian untuk mencegah kandungan gen sitokrom b babi hutan pada produk olahan daging sehingga tetap aman untuk dikonsumsi masyarakat tertentu yang memiliki pantangan terhadap daging babi. Selain itu, penelitian ini diharapkan dapat menjadi pertimbangan bagi penelitian selanjutnya, baik dari segi variabel, metode dan subyek penelitian yang digunakan.
5
BAB II
TINJAUAN PUSTAKA
A. Kornet
Corned Beef atau Kornet adalah salah satu jenis produk olahan daging sapi yang banyak digunakan dalam resep masakan Indonesia. Kornet daging sapi diolah dengan cara diawetkan dalam air garam (brine), yaitu air yang dicampur dengan larutan garam jenuh. Kemudian dimasak dengan cara simmering, yaitu direbus dengan api kecil untuk menghindari hancurnya tekstur daging sapi.
Tujuan pembuatan kornet daging sapi adalah untuk tetap dapat memperoleh produk daging sapi yang berwarna merah, awet dan praktis. Bahan dasar pembuatan kornet adalah daging sapi yang digiling. Daging tersebut kaya protein yang mempunyai kemampuan untuk mengikat air dan membentuk emulsi yang baik. Bahan tambahan yang diperlukan adalah garam dapur, nitrit, alkali fosfat, bahan pengisi, air, lemak, gula, dan bumbu (Cahyono dkk., 2018).
B. Babi hutan (Sus scrofa)
Babi hutan merupakan mamalia berkuku (hoofed) yang mempunyai tulang belakang dan berjalan dengan menggunakan empat kaki (Swindle, 2007). Klasifikasi taksonomi dari babi hutan adalah sebagai berikut :
Kingdom : Animalia Filum : Chordata Kelas : Mammalia Subkelas : Theria Infrakelas : Eutheria Ordo : Artiodactyla Famili : Suidae Subfamili : Suinae Genus : Sus (Irekhore, 2012)
Babi hutan adalah hewan omnivora. Kelompok omnivora mengonsumsi tumbuhan dan hewan. Seekor babi hutan memiliki moncong hidung, mata dan buntut kecil yang mungkin berombak, kaku atau lurus. Babi hutan memiliki tubuh yang gempal, kaki pendek, dan bulu yang kasar. Ada empat jari pada masing-masing kakinya, dengan dua jari tengah besar yang digunakan untuk berjalan. Babi hutan dengan nama ilmiah Sus scrofa berwarna coklat kehitaman dan memiliki bulu yang agak tebal pada seluruh badan dengan berat berkisar antara 50-350 kg (Irekhore, 2012; Widowati, 2013).
C. DNA Mitokondria
Mitokondria adalah organel yang ada di hampir semua sel eukariotik dalam jumlah mulai dari satu salinan hingga beberapa ribu per sel (Desler dkk., 2011). Fungsi utama dari mitokondria adalah untuk regenerasi ATP (Adenosine Triphosphate) dengan fosforilasi oksidatif (Lackie dkk., 2007). Mitokondria sering disebut sebagai pembangkit energi dalam sel karena ATP yang dihasilkan digunakan oleh sel sebagai sumber energi untuk berbagai aktivitasnya (Wandia, 2001). Mitokondria memiliki molekul DNA tersendiri dengan ukuran kecil yang susunannya berbeda dengan DNA inti (Solihin, 1994).
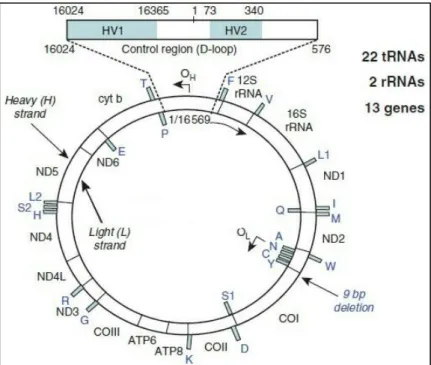
Genom mitokondria adalah molekul DNA untai ganda (double stranded DNA molecule) berbentuk sirkuler dengan panjang basa 15,7 sampai 19,5 kb. Pada sebagian besar mamalia, mtDNA (DNA mitokondria) memiliki panjang basa sekitar 16 kb yang berisi 13 gen penyandi protein yakni 3 subunit sitokrom oksidase (CO I-III), 7 subunit NADH-dehidrogenase (ND 1-6 dan ND 4L), 2 subunit ATP-ase (6 dan 6L), dan sitokrom b (Cyt b); 2 gen rRNA yakni 12S dan 16S; dan 22 tRNA yang semuanya terlibat dalam proses fosforilasi oksidatif (OXPHOS). Daerah penyandi (coding) meliputi 90% dari seluruh genom mitokondria dan sisanya 10% merupakan daerah bukan penyandi (non coding). Daerah bukan peyandi (non coding) utama terletak pada wilayah displacement-loop (D-displacement-loop). Bagian lain dari daerah bukan penyandi terletak pada gugus tRNA antara gen CO I dan ND 2 (Ladoukakis and Zouros, 2017; Wandia, 2001).
7
Gambar 1. Struktur DNA Mitokondria (Butler, 2005)
DNA mitokondria (mtDNA) diwarisi secara keseluruhan dari ibu tanpa rekombinasi (Butler, 2005). Molekul mtDNA merupakan molekul polimer rantai ganda, yang terdiri dari rantai heavy (H) strand dan rantai light (L) strand. H-strand kaya akan purin (adenin dan guanin), sedangkan L-H-strand didominasi pirimidin (timin dan sitosin) (Hidayat, 2017). Jumlah molekul DNA mitokondria (mtDNA) di dalam sel bisa sangat bervariasi. Setiap sel dapat mengandung ratusan mitokondria sehingga mtDNA yang ada pada setiap sel bisa mencapai ribuan. Hal ini menyebabkan tingkat keberhasilan mtDNA menjadi lebih besar dibandingkan DNA nukleus sebagai marker pada sampel biologis yang mungkin telah rusak karena panas atau lembab (Butler, 2005).
D. Gen Sitokrom b
Gen sitokrom b merupakan gen yang terletak di DNA Mitokondria pada daerah penyandi tepatnya pada heavy strand untuk menyandi protein sitokrom b. Gen ini banyak digunakan untuk meneliti hubungan spesies dari genus atau famili yang sama. Sitokrom b termasuk satu dari 11 komponen protein-protein yang disebut kompleks III. Kompleks III dalam mitokondria berperan dalam proses fosforilasi oksidatif untuk menghasilkan ATP sebagai sumber energi untuk berbagai aktivitas dalam sel (Gao dkk., 2015; Wandia, 2001; Widayanti dkk.,
2004). Analisis mtDNA sitokrom b efektif dalam mengidentifikasi spesies asal untuk sampel biologis (Butler, 2005). Gen sitokrom b dapat digunakan sebagai penanda genetik karena berdasarkan posisinya, gen ini mempunyai region yang lebih kekal dan beragam sehingga dalam identifikasi spesies tidak menimbulkan keambiguan (Farias dkk., 2001). Genom utuh mitokondria dari Sus scrofa (babi hutan) memiliki panjang sekuen 16.613 bp (base pair). Target gen sitokrom b mitokondria dari Sus scrofa berada pada rentang wilayah 15.399-15.796 (NCBI, 2013).
Gambar 2. Wilayah Target Gen Sitokrom b Babi Hutan (NCBI, 2013)
E. Polymerase Chain Reaction (PCR)
Polymerase Chain Reaction (PCR) merupakan teknik amplifikasi potongan DNA yang diinginkan secara in vitro pada daerah spesifik yang dibatasi oleh dua buah primer oligonukleotida dengan bantuan enzim polymerase (Wardani dan Sari, 2015). Secara prinsip, PCR merupakan proses yang diulang-ulang antara 20-30 kali siklus. Setiap siklus terdiri atas tiga tahap yaitu :
1) Denaturasi (denaturation)
Selama proses denaturasi, DNA untai ganda akan membuka menjadi dua untai tunggal. Hal ini disebabkan karena suhu denaturasi yang tinggi menyebabkan putusnya ikatan hidrogen DNA diantara basa-basa yang komplemen. Pemisahan ini menyebabkan DNA tidak stabil dan siap menjadi cetakan (template) bagi primer. Denaturasi biasanya dilakukan pada suhu 90-95˚C.
9
2) Penempelan primer (annealing)
Pada tahap penempelan primer (annealing), primer akan menuju ke daerah spesifik yang komplemen dengan urutan primer. Pada proses annealing ini, ikatan hidrogen akan terbentuk antara primer dengan urutan komplemen pada cetakan (template). Proses ini biasanya dilakukan pada suhu 50-60˚C. Selanjutnya, DNA polimerase akan berikatan sehingga ikatan hidrogen tersebut akan menjadi sangat kuat dan tidak akan putus kembali apabila dilakukan reaksi polimerase selanjutnya.
3) Pemanjangan (elongasi/extention)
Umumnya, elongasi ini terjadi pada suhu 72˚C. Primer yang telah menempel tadi akan mengalami perpanjangan pada sisi 3’nya dengan penambahan dNTP yang komplemen dengan template oleh DNA polimerase.
Selain ketiga proses tersebut, biasanya PCR didahului dan diakhiri oleh tahapan berikut.
1) Pra-denaturasi
Tahap ini dilakukan selama 1-9 menit di awal reaksi untuk memastikan kesempurnaan denaturasi dan mengaktifasi DNA polimerase.
2) Final elongasi
Biasanya dilakukan pada suhu optimum enzim (70-72˚C) selama 5-15 menit untuk memastikan bahwa setiap untai tunggal yang tersisa sudah diperpanjang secara sempurna. Proses ini dilakukan setelah siklus PCR terakhir.
(Irianto, 2017) Dengan menggunakan metode PCR dapat meningkatkan jumlah urutan DNA ribuan bahkan jutaan kali dari semula (Irianto, 2017). Target PCR yaitu asam nukleat (DNA) untai ganda yang diekstraksi dari sel dan didenaturasi menjadi asam nukleat untai tunggal (Nollet dan Toldrá, 2011). Metode PCR dapat dilakukan dengan menggunakan komponen dalam jumlah yang sangat sedikit. Berikut terdapat beberapa komponen utama dalam proses PCR yaitu :
1) Cetakan DNA (DNA Template) yaitu fragmen DNA yang akan dilipatgandakan.
2) Primer yaitu suatu urutan nukleotida pendek (15-25 basa nukleotida) digunakan untuk mengawali sintesis rantai DNA. Primer yang berikatan dengan segmen awal sekuen DNA template yang ingin diamplifikasi disebut primer forward, sedangkan primer yang berikatan dengan segmen akhir pada sekuen DNA template yang ingin diamplifikasi disebut primer reverse.
3) Deoksiribonukleotida trifosfat (dNTP), terdiri atas dATP, dCTP, dGTP, dCTP. 4) Enzim DNA polimerase yaitu enzim yang mengkatalisis reaksis sintesis DNA
PCR melibatkan banyak siklus yang masing-masing mempunyai tiga tahapan berulang.
5) PCR buffer merupakan buffer yang digunakan untuk PCR dan umumnya tersusun atas KCl, Tris-Cl dan MgCl2.
(Irianto, 2017) F. Landasan Teori
Daging merupakan bahan pangan yang dikonsumsi masyarakat sebagai sumber protein. Namun, daging seperti daging babi hutan tidak bisa dikonsumsi oleh semua kalangan karena dapat menyebabkan gangguan kesehatan pada beberapa golongan serta adanya pantangan untuk agama tertentu. Menurut Puspitasari dkk. (2019), sejumlah produk makanan berbahan dasar daging ditemukan tercampur dengan daging babi hutan, terutama pada makanan olahan daging sapi salah satunya adalah kornet. Pada penelitian yang dilakukan oleh Rahayu dkk. (2020), terdapat satu produk kornet sapi dan satu produk dendeng dari total empat sampel positif mengandung daging babi hutan berdasarkan nilai Ct (threshold cycle) dibawah 30 menggunakan metode Real Time PCR. Gen sitokrom b yang terdapat dalam DNA mitokondria merupakan salah satu marker yang potensial untuk mendeteksi spesies dari genus atau famili yang sama. DNA mitokondria memiliki jumlah salinan DNA yang tinggi, sehingga cocok untuk analisis dengan jumlah DNA terbatas atau DNA yang mudah terdegradasi.
Pengidentifikasian gen sitokrom b mtDNA (DNA mitokondria) dapat dilakukan dengan metode PCR. PCR merupakan teknik amplifikasi potongan DNA yang diinginkan secara in vitro pada daerah spesifik yang dibatasi oleh dua
11
buah primer oligonukleotida dengan bantuan enzim polymerase (Wardani dan Sari, 2015). Menurut Tanabe (cit Fibriana dkk., 2012) PCR mempunyai sensitivitas yang tinggi karena berhasil mendeteksi gen sitokrom b babi pada sampel daging segar dan daging olahan. Oleh karena itu, metode ini dapat digunakan untuk mendeteksi kandungan daging babi pada daging olahan seperti kornet.
G. Hipotesis
Terdapat kandungan gen sitokrom b babi hutan yang dibuktikan dengan terbentuknya pita dengan panjang 398 bp pada produk kornet sapi di Yogyakarta dengan metode PCR.
12 BAB III
METODE PENELITIAN
A. Jenis dan Rancangan Penelitian
Jenis penelitian yang digunakan adalah deskriptif kualitatif dimana sampel akan dideteksi dengan metode PCR dan rancangan penelitian yang dilakukan merupakan penelitian observasional karena tidak dilakukan intervensi pada variabel bebas.
B. Variabel dan Definisi Operasional 1. Variabel Utama
a) Variabel bebas dalam penelitian ini adalah isolat DNA dari kornet daging sapi yang diperoleh dari supermarket wilayah Yogyakarta.
b) Variabel tergantung dalam penelitian ini adalah produk PCR yang dideteksi dengan elektroforesis.
2. Variabel pengacau
a) Variabel pengacau terkendali dalam penelitian ini adalah ketepatan desain primer dan kemurnian isolat DNA.
b) Variabel pengacau tidak terkendali dalam penelitian adalah proses pembuatan sampel daging kornet sapi, bahan baku yang digunakan untuk mengolah produk kornet sapi dan kualitas bahan baku.
3. Definisi Operasional
a) Kornet sapi adalah produk olahan daging sapi yang beredar di supermarket wilayah Yogyakarta.
b) Gen sitokrom b babi hutan merupakan gen target yang akan diidentifikasi menggunakan PCR.
c) PCR merupakan metode enzimatis untuk melipatgandakan atau mengamplifikasi DNA dalam waktu singkat secara in vitro.
d) Primer adalah potongan DNA yang urutan nukleotidanya komplemen dengan urutan nukleotida dalam cetakan DNA (DNA template).
13
f) Elektroforesis adalah teknik pemisahan berdasarkan pergerakan molekul (ion, molekul, makromolekul) dalam medan listrik dan digunakan untuk analisis kualitatif produk PCR.
g) Suhu annealing adalah suhu yang digunakan pada tahap annealing agar primer menuju daerah spesifik dan menempel dengan tepat pada fragmen DNA target.
h) Jumlah siklus amplifikasi adalah jumlah pengulangan tahapan denaturation, annealing dan extension dalam PCR agar memperoleh produk PCR yang optimum.
C. Bahan penelitian 1. Bahan Penelitian
Bahan-bahan yang digunakan dalam penelitian ini adalah daging babi hutan segar (kontrol positif babi), daging sapi segar (kontrol positif sapi) dan sampel kornet daging sapi. Primer forward universal: 5’ GAC CTC CCA GCT CCA TCA AAC ATC TCA TCT TGA TGA AA 3’, primer reverse sapi: 5’ CTA GAA AAG TGT AAG ACC CGT AAT ATA AG 3’, primer reverse babi hutan: 5’ GCT GAT AGT AGA TTT GTG ATG ACC GTA 3’ diadopsi dari Matsunaga dkk. (1999), kit isolasi DNA (FavorPrep Tissue Genomic DNA Extraction Mini Kit) yang terdiri dari FATG1 buffer, FATG2 buffer, Proteinase K, W1 Buffer, wash buffer, dan elution buffer, Tris-Borate-EDTA Buffer (10x), gel agarosa, etanol absolut, water for injection (WFI), FluoroVue™ Nucleic Acid Gel Stain (10.000x) SMOBiO, DNA ladder 100 bp (SMOBiO), loading dye, ExcelTaq™ 5X PCR Master Dye Mix (berisi Taq DNA Polymerase, reaction buffer, MgCl2, dNTP), akuabides (ddH2O).
2. Kriteria subyek uji
Subyek uji yang digunakan dalam penelitian ini adalah kornet daging sapi sebanyak 5 sampel dengan merk berbeda, belum melewati masa kadaluwarsa, dan berlabel halal yang diperoleh dari supermarket wilayah Yogyakarta.
D. Alat Penelitian
Alat yang digunakan dalam penelitian ini adalah Bio-Rad-T100™ thermal cycler, elektroforesis (Sigma Aldrich), kit isolasi DNA (FavorPrep™ Tissue Genomic DNA Extraction Mini Kit) yang berisi FATG mini column, collection tube, elution tube, dan micropestle, microsentrifuge tube, tabung PCR, yellow tip, blue tip, mikropipet (Soccorex Swiss), vortex (Fisher Scientific), water bath, microwave, centrifuge, mortar stamper, neraca analitik digital, UV Transiluminator (Fischer Scientific), kamera, alat-alat gelas.
E. Tata Cara Penelitian 1. Pengambilan Sampel
Pengambilan sampel kornet daging sapi dilakukan oleh peneliti. Sebanyak 5 sampel kornet daging sapi dengan merk yang berbeda diperoleh dari Daerah Istimewa Yogyakarta dengan teknik purposive sampling, kemudian dilakukan replikasi 3 kali untuk setiap sampel.
2. Optimasi preparasi sampel
a. Preparasi Sampel tanpa perlakuan. Preparasi sampel tanpa perlakuan dilakukan sebelum isolasi DNA yaitu masing-masing sampel kornet daging sapi ditimbang sebanyak 1 gram kemudian dimasukan ke dalam mortar dan dihaluskan menggunakan stamper.
b. Preparasi Sampel dengan perlakuan. Preparasi sampel dengan perlakuan dilakukan sebelum isolasi DNA yaitu masing-masing sampel kornet daging sapi ditimbang sebanyak 5 gram ke dalam gelas beaker 50 ml kemudian ditambahkan sedikit air yang dihangatkan pada suhu 80˚C lalu diaduk. Sampel dipindahkan ke gelas beaker 500 ml kemudian gelas beaker sebelumnya dibilas menggunakan air hangat dan dituangkan ke gelas beaker 500 ml. Larutan sampel ditambahkan air hangat sekitar 300 ml kemudian diletakkan di atas water bath selama 2 jam sambil diaduk. Selanjutnya, larutan sampel didinginkan di suhu ruangan kemudian disaring. Sampel yang telah diberi perlakuan tersebut kemudian ditimbang 50 mg dan dihaluskan menggunakan mortar dan stamper.
15
3. Preparasi kontrol positif dan negatif
Preparasi kontol positif dan negatif sebelum isolasi DNA perlu dilakukan. Daging babi hutan segar sebagai kontrol positif babi dan daging sapi segar sebagai kontrol positif sapi dihaluskan menggunakan mortar dan stamper. 4. Isolasi DNA
Isolasi DNA dilakukan dengan menggunakan Tissue Genomic DNA Extraction Mini Kit. Daging babi hutan segar (kontrol positif babi), daging sapi segar (kontrol positif sapi), dan sampel kornet daging sapi masing-masing yang sudah dipreparasi sebelumnya ditimbang 25 mg dan dipindahkan ke tabung mikrosentrifuge 1,5 mL kemudian dihaluskan menggunakan mikropestel. Sebanyak 200 µl FATG1 buffer ditambahkan ke dalam sampel dan dihaluskan menggunakan mikropestel. Proteinase K sebanyak 20 µl ditambahkan ke dalam campuran sampel, divorteks lalu diinkubasi pada suhu 60˚C selama 3 jam. Sel dilisiskan dengan cara menambahkan FATG2 buffer sebanyak 200 µl, divorteks dan diinkubasi kembali pada suhu 70˚C selama 10 menit. Setelah diinkubasi, ditambahkan etanol absolut sebanyak 200 µl lalu divorteks. FATG mini column dipasangkan dengan 2 ml collection tube dan sampel dipindahkan ke dalam FATG mini column tersebut kemudian disentrifugasi pada kecepatan 17.000 xg selama 1 menit. Collection tube sebelumnya dibuang dan FATG mini column dipasangkan dengan 2 ml collection tube yang baru. Berikutnya FATG mini column ditambahkan 400 µl W1 buffer dan disentrifugasi pada kecepatan 17.000 xg selama 1 menit kemudian collection tube tersebut dibuang. FATG mini column dipasangkan dengan 2 ml collection tube yang baru lalu ditambahkan 750 µl Wash buffer dan disentrifugasi pada kecepatan 17.000 xg selama 1 menit. Collection tube tersebut dibuang dan dipasangkan lagi 2 ml collection tube terakhir yang baru pada FATG mini Column dan disentrifugasi pada kecepatan 17.000 xg selama 3 menit untuk mengeringkan kolom. FATG mini column yang sudah dikeringkan kemudian dipasangkan dengan 1,5 ml tabung mikrosentrifuge lalu ditambahkan 100 µl elution buffer yang telah dipanaskan ke tengah membran FATG mini column sampai benar-benar terserap. Selanjutnya disentrifugasi pada
kecepatan 17.000 xg selama 2 menit untuk mengelusi DNA. DNA yang telah berhasil dielusi tersebut kemudian disimpan pada suhu -20˚C.
5. Penyiapan Gel Agarosa
Pada penelitian ini, terdapat dua konsentrasi gel agarosa yang digunakan yaitu 1,0% dan 1,5%. Gel agarosa 1,0% digunakan untuk mengidentifikasi kemurnian isolat DNA, sedangkan gel agarosa 1,5% digunakan untuk mengidentifikasi produk PCR. Pembuatan gel agarosa 1,0% dilakukan dengan cara melarutkan 0,25 g agarosa dalam 1x larutan TBE sebanyak 25 mL, lalu dipanaskan menggunakan microwave sekitar 5 menit hingga larut. Sebanyak 2,5 µL larutan Nucleic Acid Gel Stain ditambah lalu diaduk hingga homogen. Larutan tersebut dituang ke dalam cetakan yang telah diberi sisiran pada bagian tepi dan gel dibiarkan mengeras. Setelah mengeras, sisiran dicabut hingga terbentuk sumur-sumur sebagai tempat dimasukkannya sampel yang akan diidentifikasi. Pembuatan gel agarosa 1,5% dilakukan dengan cara melarutkan 0,375 g agarosa dalam 1x larutan TBE sebanyak 25 mL, lalu dipanaskan menggunakan microwave sekitar 5 menit hingga larut. Sebanyak 2,5 µL larutan Nucleic Acid Gel Stain ditambahkan lalu diaduk hingga homogen. Larutan tersebut dituang ke dalam cetakan yang telah diberi sisiran pada bagian tepi dan gel dibiarkan mengeras. Setelah mengeras, sisiran dicabut hingga terbentuk sumur-sumur sebagai tempat dimasukkannya sampel yang akan diidentifikasi.
6. Analisis Kualitatif Isolat DNA
Analisis kualitatif isolat DNA dilakukan dengan metode elektroforesis. Gel agarosa 1,0% ditempatkan ke dalam gel tray lalu diberi larutan buffer 1x TBE hingga permukaan gel terendam. Isolat DNA sebanyak 5,0 µl dicampurkan loading dye sebanyak 1,0 µl di atas plat tetes. Campuran tersebut dimasukkan ke dalam sumur-sumur gel menggunakan mikropipet dan salah satu sumuran gel diisi dengan 100 bp DNA ladder sebanyak 3,0 µL sebagai marker. Elektroforesis dijalankan pada tegangan 110 volt selama 30 menit. Hasil elektroforesis diamati di bawah lampu UV Transilluminator dan didokumentasikan menggunakan kamera. Panjang pita akan diketahui dengan cara menarik garis lurus masing-masing pita sampel DNA dengan posisi pita DNA ladder.
17
7. Optimasi PCR
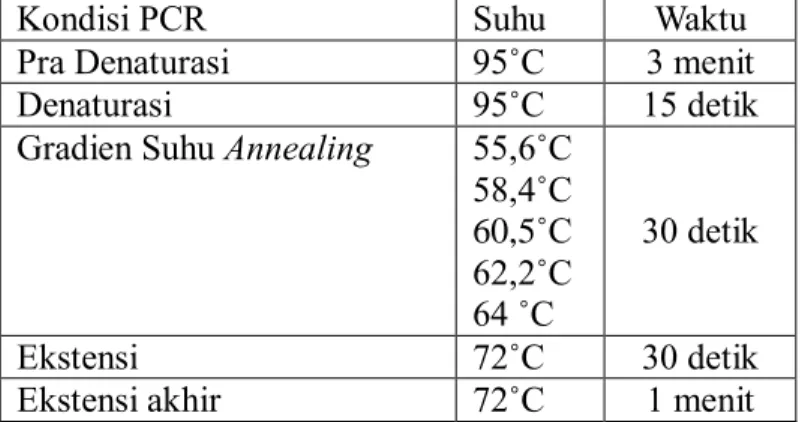
Gradien suhu yang digunakan yaitu 55,6°C;58,4°C;60,5°C;62,2°C; dan 64°C. Kondisi PCR yang digunakan dicantumkan pada Tabel I. Siklus amplifikasi dari tahap denaturasi hingga ekstensi diulangi sebanyak 30 kali.
Tabel I. Kondisi PCR
Kondisi PCR Suhu Waktu
Pra Denaturasi 95˚C 3 menit
Denaturasi 95˚C 15 detik
Gradien Suhu Annealing 55,6˚C 58,4˚C 60,5˚C 62,2˚C 64 ˚C 30 detik Ekstensi 72˚C 30 detik
Ekstensi akhir 72˚C 1 menit 8. Identifikasi Gen Sitokrom b
Identifikasi gen sitokrom b dilakukan dengan menggunakan primer forward universal: 5’ GAC CTC CCA GCT CCA TCA AAC ATC TCA TCT TGA TGA AA 3’, primer reverse sapi: 5’ CTA GAA AAG TGT AAG ACC CGT AAT ATA AG 3’, primer reverse babi hutan: 5’ GCT GAT AGT AGA TTT GTG ATG ACC GTA 3’ diadopsi dari Matsunaga dkk. (1999). Sebanyak 10 µl SMOBio ExcelTaq 5X PCR Master Dye Mix TP1200, 0,5 µl forward primer, 0,5 µl reverse primer babi, 1 µl DNA template, 3.0 µl isolat DNA dan ditambahkan nuclease free water hingga volume total sebanyak 30 µl untuk mengidentifikasi kandungan babi. Sedangkan untuk yang mengandung sapi (negatif mengandung babi), sebanyak 10 µl SMOBio ExcelTaq 5X PCR Master Dye Mix TP1200, 0,5 µl forward primer, 0,5 µl reverse primer sapi, 1 µl DNA template, 3.0 µl isolat DNA dan ditambahkan nuclease free water hingga volume total sebanyak 30 µl. Amplifikasi dilakukan dengan menggunakan mesin thermal cycler. Kondisi PCR yang digunakan yaitu pra-denaturasi pada suhu 95˚C selama 3 menit, dilanjutkan dengan denaturasi pada suhu 95˚C selama 15 detik, annealing pada suhu 58,4˚C selama 30 detik, ekstensi pada suhu 72˚C selama 30 detik. Siklus amplifikasi ini dilakukan sebanyak 30 kali dan diakhiri dengan ektensi akhir (final extension) pada suhu 72˚C selama 1 menit.
9. Analisis Produk PCR Menggunakan Teknik Elektroforesis
Analisis produk PCR dilakukan dengan elektroforesis menggunakan gel agarosa 1,5% sebagai fase diam. Gel agarosa 1,5% ditempatkan ke dalam gel tray lalu diberi larutan buffer 1x TBE hingga permukaan gel terendam. Produk PCR diambil sebanyak 5,0 µl lalu dimasukan ke dalam sumur-sumur gel menggunakan mikropipet. Salah satu sumuran gel diisi dengan 3,0 µl DNA ladder sebagai marker kemudian elektroforesis dijalankan pada tegangan 110 volt selama 30 menit. Hasil elektroforesis dievaluasi dengan lampu UV Transilluminator dan didokumentasikan menggunakan kamera.
F. Analisis Hasil
Hasil yang dianalisis adalah ada atau tidaknya kandungan gen sitokrom b babi hutan. Apabila terdapat kandungan gen sitokrom b babi hutan maka akan terbentuk pita berukuran 398 bp, namun jika tidak terdapat kandungan gen sitokrom b babi hutan maka akan terbentuk pita berukuran 274 bp.
19 BAB IV
HASIL DAN PEMBAHASAN
A. Optimasi Preparasi Sampel
Pada penelitian ini, kornet sapi digunakan sebagai sampel, daging sapi segar sebagai kontrol positif sapi diperoleh dari supermarket dan daging babi segar sebagai kontrol positif babi diperoleh dari pasar yang berada di wilayah Yogyakarta. Sampel kornet sapi yang diambil yaitu sebanyak lima merk berbeda menggunakan teknik Purposive Sampling. Menurut Gay dkk. (2012), purposive sampling merupakan suatu teknik pengambilan sampel dengan menentukan kriteria sampel yang diyakini dapat mewakili suatu populasi.
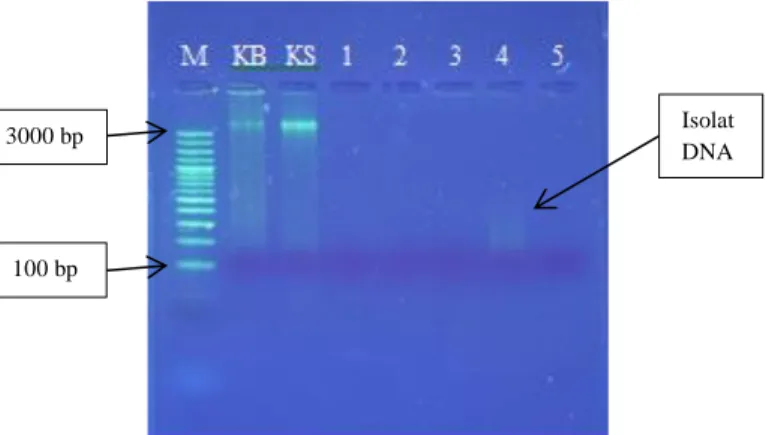
Sampel kornet sapi terlebih dahulu dipreparasi sebelum memasuki tahapan isolasi DNA. Kontrol positif berupa daging babi segar dan daging sapi segar juga dipreparasi untuk mempermudah proses lisis saat dilakukan isolasi DNA. Replikasi sampel sebanyak tiga kali dilakukan dengan tujuan untuk memastikan hasil yang diperoleh akurat (Rau dkk., 2018). Kornet sapi merupakan produk daging sapi yang melalui tahapan pengolahan salah satunya adalah perlakuan kimiawi (penambahan pengawet nitrit). Hal ini bisa mempengaruhi hasil isolasi DNA. Optimasi preparasi sampel dilakukan untuk mengetahui proses preparasi terbaik yang digunakan pada proses isolasi DNA (Cahyono dkk., 2018; Erwanto, 2018). Preparasi sampel dengan perlakuan dilakukan untuk memisahkan kandungan nitrit yang ada pada sampel kornet sapi. Tujuan dilakukan pemisahan ini agar kandungan nitrit tidak mengganggu proses isolasi DNA. Hasil isolasi DNA sampel yang dipreparasi dengan perlakuan (Gambar 3) menunjukkan DNA yang lebih terlihat dibandingkan dengan isolasi DNA sampel yang dipreparasi tanpa perlakuan (Gambar 2). Dengan demikian, preparasi sampel dengan perlakuan merupakan metode preparasi yang paling baik untuk proses isolasi DNA sampel kornet.
Gambar 3. Hasil Elektroforesis Isolat DNA Sampel Tanpa Perlakuan Keterangan :
M : DNA ladder sebagai marker
1-5 : Isolat DNA produk kornet sapi tanpa perlakuan KB : Kontrol positif babi (isolat DNA daging babi segar) KS : Kontrol positif sapi (isolat DNA daging sapi segar)
Kondisi elektroforesis : Fase diam gel agarosa 1%; fase gerak larutan TBE 1x; tegangan 110 Volt, waktu running 30 menit; volume injeksi 6 μL; DNA
ladder 3 μL
Gambar 4. Hasil Elektroforesis Isolat DNA Sampel dengan Perlakuan Keterangan :
M : DNA ladder sebagai marker
1-5 : Isolat DNA produk kornet sapi dengan perlakuan KB : Kontrol positif babi (isolat DNA daging babi segar) KS1 : Kontrol positif sapi (isolat DNA daging sapi segar) KS2 : Kontrol positif sapi (isolat DNA daging sapi segar)
Kondisi elektroforesis : Fase diam gel agarosa 1%; fase gerak larutan TBE 1x; tegangan 110 Volt, waktu running 30 menit; volume injeksi 6 μL; DNA
ladder 3 μL
B. Isolasi DNA
Isolasi DNA bertujuan untuk memisahkan DNA dari partikel-partikel lain seperti lipid, protein, polisakarida, dan zat lainnya (Hariyadi dkk., 2018). Prinsip utama dari isolasi DNA yaitu pengancuran (lisis), ekstraksi atau pemisahan DNA dari bahan padat seperti selulosa dan protein, serta pemurnian
3000 bp Isolat DNA 100 bp 3000 bp 100 bp Isolat DNA
21
DNA (Hidayati dkk., 2016). Lisis merupakan perusakan dinding sel, dan proses lisis ini bisa dilakukan dengan cara fisik maupun kimia. Pemurnian DNA merupakan proses untuk memisahkan DNA dari lisat sel (protein, karbohidrat, lipid) dan kontaminan lain (Wardani dan Sari, 2015).
Isolasi DNA dilakukan menggunakan FavorPrep™ Tissue Genomic DNA Extraction Mini Kit. Tahap awal isolasi DNA adalah proses perusakan atau penghancuran membran dan dinding sel yang bertujuan untuk mengeluarkan isi sel (Hidayati dkk., 2016). Pada penelitian ini, proses lisis menggunakan FATG1 buffer, FATG2 buffer dan penambahan Proteinase K. Menurut Mulyani dkk. (2011), buffer lisis diperlukan untuk memecah membran sel agar dapat mengeluarkan DNA genom. Protein dihilangkan menggunakan proteinase K. Enzim proteinase K berperan untuk menghancurkan protein (Kamaliah, 2017). Penggunaan vorteks dalam proses isolasi DNA bertujuan untuk membantu proses lisis (Mulyani dkk., 2011). Selain itu, inkubasi dilakukan pada suhu 60˚C selama 3 jam kemudian suhu dinaikan hingga 70˚C selama 10 menit serta perlu dibolak balik bertujuan untuk mendukung degradasi protein dan larutan buffer dapat melisiskan sel dengan sempurna (Mulyani dkk., 2011). Protein yang telah rusak bersama komponen hasil lisis lainnya dipisahkan melalui sentrifugasi (Irianto, 2017). Sampel disentrifugasi dengan kecepatan tinggi agar komponen yang berukuran lebih besar atau lebih berat akan mengendap membentuk sedimen pada bagian bawah tabung (Langga dkk., 2012). Menurut Bettelheim dan Landdesberg (cit., Hutami dkk., 2018) setelah disentrifugasi, campuran akan terpisah menjadi dua fase yakni fase organik pada lapisan bawah dan fase aqueous (air) berada pada lapisan atas. DNA akan berada pada fase aqueous sementara makromolekul protein, lipid, dan polisakarida akan berada pada fase organik.
Pemurnian DNA dalam proses isolasi dilakukan dengan penambahan etanol absolut. Hal tersebut menyebabkan penurunan kelarutan asam nukleat dalam air sehingga DNA mengalami presipitasi membentuk endapan DNA pada bagian bawah tabung (Hutami dkk., 2018). Endapan DNA dicuci menggunakan dua jenis buffer yang terdapat pada kit isolasi yaitu W1 buffer dan wash buffer. Menurut Mulyani dkk. (2011), pencucian bertujuan untuk menghilangkan residu
etanol dan senyawa lain yang ikut mengendap bersama dengan DNA. DNA dielusi menggunakan elution buffer. Larutan elution buffer berfungsi untuk menghasilkan purifikasi DNA (DNA yang benar-benar murni) (Utami dkk., 2013). Isolat DNA yang telah diperoleh disimpan di dalam freezer bersuhu -20˚C (Marwayana, 2015). Kemurnian dari isolat DNA dapat ditentukan dari ada tidaknya komponen lain seperti protein dan RNA dengan menggunakan elektroforesis (Hidayati dkk., 2016).
C. Analisis Kualitatif Isolat DNA
Isolat DNA diuji secara kualitatif dengan elektroforesis menggunakan gel agarosa 1% dalam larutan TBE 1x (Tris Borate EDTA) yang ditambahkan Nucleic Acid Gel Stain. Elektroforesis merupakan suatu teknik untuk memisahkan molekul menggunakan medan listrik berdasarkan ukuran molekul. Kecepatan migrasi molekul DNA tergantung pada ukuran molekul DNA, kerapatan media gel yang dilalui DNA, dan arus listrik. Molekul DNA bermuatan negatif sehingga akan bergerak menuju kutub positif dalam medan listrik (Irianto, 2017; Novitasari dkk., 2014). Nucleic Acid Gel Stain berfungsi sebagai pewarna agar DNA dapat tervisualisasi di bawah UV Transilluminator (Hidayati dkk., 2016; Utami dkk., 2013). Nucleic Acid Gel Stain dirancang dengan tujuan menggantikan ethidium bromida (EtBr) yang bersifat mutagen dan karsinogenik (Crisafuli dkk., 2015; Maftuchah dkk., 2014). Isolat DNA dicampurkan dengan loading dye yang mengandung gliserol dan bromofenol biru sebelum melakukan proses elektroforesis untuk menambah densitas DNA dan sebagai penanda migrasi DNA (Fatchiyah dkk., 2011; Novitasari dkk., 2014).
Hasil elektroforesis yang tervisualisasi di bawah UV transilluminator berupa pita-pita DNA. Pita tebal dan terang secara kualitatif mengindikasikan konsentrasi isolasi DNA yang dihasilkan tinggi sedangkan pita yang tipis mengindikasikan konsentrasi DNA yang dihasilkan kecil (Hidayati dkk., 2016). Hasil elektroforesis pada Gambar 3 menunjukkan terbentuknya pita tunggal dan tebal pada isolat DNA kontrol daging babi maupun daging sapi. Sedangkan pada isolat DNA produk kornet sapi terlihat smear pada sampel nomor 4. Genom yang
23
smear pada hasil elektroforesis disebabkan karena tidak utuhnya DNA yang terisolasi. Smear tersebut disebabkan karena DNA yang diperoleh rusak atau sudah terfragmentasi (Mulyani dkk., 2011; Novitasari dkk., 2014). Jumlah DNA yang diekstraksi dalam daging olahan akan berkurang, khususnya pada produk olahan yang telah melalui proses autoklaf berkisar antara 7-9 μg / 300 mg karena makanan olahan seperti kornet melibatkan perlakuan autoklaf. Pada prinsipnya, sterilisasi autoklaf menggunakan panas dan tekanan dari uap air. Suhu didalamnya dapat mencapai 115-125˚C dan tekanan uapnya mencapai 2-4 atm, sehingga pada autoklaf semakin tinggi tekanannya, maka suhu yang dihasilkan semakin tinggi. Tekanan yang diberikan menyebabkan suhu yang dihasilkan >100˚C dan suhu ini mengakibatkan terjadinya degradasi pada DNA. Kemampuan DNA yang terdegradasi untuk mengikat pewarna dalam elektroforesis gel akan berkurang (Calderón-Franco dkk., 2020; Chandrika dkk., 2010; Farmawati dkk., 2015; Rahayu dkk., 2020). Dengan demikian, tidak ada pita DNA yang muncul pada produk kornet sapi.
D. Identifikasi Gen Sitokrom b
Gen sitokrom b merupakan gen yang terletak pada DNA mitokondria dan memiliki variasi urutan sekuen antara satu spesies dengan spesies lainnya sehingga gen ini banyak digunakan untuk meneliti hubungan spesies dari genus atau famili yang sama (Farias dkk., 2001; Gao dkk., 2015). Amplifikasi gen sitokrom b pada penelitian ini dilakukan dengan metode PCR. Komponen-komponen yang diperlukan dalam proses amplifikasi dengan metode PCR yaitu template DNA, primer, enzim DNA polymerase, dNTPs (Deoxynucleotide triphosphates, buffer PCR dan konsentrasi Mg2+(Fatchiyah dkk., 2011).
Keberhasilan amplifikasi DNA tergantung dari ketepatan primer yang digunakan (Prakoso dkk., 2016). Proses amplifikasi gen sitokrom b dilakukan dengan menggunakan primer forward universal: 5’ GAC CTC CCA GCT CCA TCA AAC ATC TCA TCT TGA TGA AA 3’, primer reverse sapi: 5’ CTA GAA AAG TGT AAG ACC CGT AAT ATA AG 3’, primer reverse babi hutan: 5’ GCT GAT AGT AGA TTT GTG ATG ACC GTA 3’ diadopsi dari Matsunaga
dkk. (1999). Identifikasi adanya kandungan gen sitokrom b babi menggunakan primer forward universal dan primer reverse spesifik babi serta ditunjukkan dengan terbentuknya pita berukuran 398 bp. Sedangkan untuk mengindentifikasi tidak adanya kandungan babi dan memastikan bahwa sampel mengandung sapi menggunakan primer forward universal dan primer reverse spesifik sapi serta ditunjukkan dengan terbentuknya pita berukuran 274 bp. Daerah penempelan primer untuk gen sitokrom b babi terletak pada urutan basa 15.399-15.796 (Gambar 1), sedangkan daerah penempelan primer gen sitokrom b sapi terletak pada urutan basa 14.571-14.844 (Gambar 4). Primer berfungsi sebagai pembatas fragmen DNA target yang akan diamplifikasi. Setiap satu fragmen target, memerlukan sepasang primer yang sesuai dengan DNA target (Maitriani dkk., 2015).
Gambar 5. Wilayah Target Gen Sitokrom b Sapi (NCBI, 2009)
Pembuatan DNA template merupakan proses pertama yang dilakukan dalam metode PCR. DNA template adalah DNA untai ganda yang membawa basa fragmen atau gen yang akan digandakan. Urutan basa ini disebut juga urutan target (target sequence) (Utami dkk., 2013). Pada proses PCR, terdapat tiga tahapan penting yaitu denaturasi, penempelan primer (annealing), dan ekstensi. Pada tahap denaturasi, DNA untai ganda akan membuka menjadi dua untai tunggal. Suhu yang digunakan pada tahap ini adalah 95˚C. Hal ini dikarenakan suhu yang tinggi dapat menyebabkan putusnya ikatan hidrogen yang menghubungkan dua untai DNA.
Annealing merupakan tahap terjadinya penempelan primer pada DNA template. Pencarian kondisi optimal dari suhu annealing dilakukan sebab primer
25
dapat menempel pada DNA template apabila suhu yang digunakan merupakan suhu optimum, sehingga suhu yang digunakan dalam tahap annealing merupakan faktor penting keberhasilan amplifikasi DNA dengan metode PCR. Jika suhu annealing terlalu tinggi maka penempelan primer pada DNA template akan lepas sehingga produk PCR tidak terbentuk, sebaliknya jika suhu annealing terlalu rendah maka akan terjadi penempelan primer pada DNA template tidak spesifik sehingga menghasilkan produk PCR yang juga tidak spesifik (Hidayati dkk., 2016; Irianto, 2017). Primer yang digunakan pada penelitian ini mengacu pada Matsunaga dkk. (1999). Pada penelitian tersebut dilakukan dengan metode multipleks PCR sedangkan pada penelitian ini menggunakan metode unipleks PCR. Metode multipleks PCR menggunakan beberapa pasang primer untuk mengamplifikasi lebih dari satu sekuen target DNA sehingga kondisi optimal PCR pada suhu annealingnya akan berbeda dengan metode unipleks PCR yang hanya membutuhkan satu pasang primer untuk mengamplifikasi satu sekuen target DNA yang dituju (Butler, 2005). Selain itu, reagen mix PCR pada penelitian Matsunaga dkk. (1999) mengandung taq DNA polimerase yang berbeda dengan taq DNA polimerase yang terdapat dalam reagen mix PCR pada penelitian ini. Dengan demikian, hal-hal tersebut yang mendasari perlunya dilakukan optimasi kondisi PCR pada suhu annealing.
Hasil optimasi suhu annealing untuk kontrol positif daging babi ditunjukkan pada Gambar 5 dan untuk kontrol positif sapi segar pada Gambar 6. Amplifikasi gradien PCR dari fragmen gen sitokrom b spesifik babi maupun fragmen gen sitokrom b spesifik sapi menggunakan suhu annealing yang bervariasi dari suhu 55˚C-64˚C. Amplifikasi gradien PCR dilakukan untuk menentukan suhu annealing yang optimum untuk pengujian dengan menggunakan primer yang telah dirancang (Che Man dkk., 2012).
Hasil elektroforesis pada Gambar 5 menunjukkan bahwa terbentuk pita tebal dan tunggal pada seluruh variasi suhu annealing dengan ukuran 398 bp, sehingga suhu annealing untuk penempelan primer reverse spesifik gen sitokrom b babi optimum untuk semua variasi suhu. Hasil elektroforesis pada Gambar 6 menunjukkan bahwa terbentuk pita tebal dan tunggal pada variasi suhu 60,5˚C,
58,4˚C, 55,6˚C dengan ukuran 274 bp, sehingga suhu annealing untuk penempelan primer reverse spesifik gen sitokrom b sapi pada variasi suhu 60,5˚C, 58,4˚C, 55,6˚C. Dengan demikian, suhu annealing yang optimum dan digunakan pada penelitian ini yaitu 58,4˚C.
Tahap ekstensi merupakan tahap sintesis DNA. Tahap ekstensi dilakukan pada suhu 72˚C. Suhu ini adalah suhu yang optimal bagi Taq DNA polymerase untuk mensintesis pita-pita DNA baru dengan cara memanjangkan rantai primer (Bintang, 2018). Hasil dari produk PCR yang telah diamplifikasi dianalisis menggunakan elektroforesis.
Gambar 6. Hasil Optimasi Suhu Annealing Produk PCR Kontrol Daging Babi Keterangan:
M : DNA ladder
Kondisi elektroforesis : Fase diam gel agarosa 1,5%; fase gerak larutan TBE 1X; tegangan 110 Volt, waktu running 30 menit; volume injeksi 5 μL
Gambar 7. Hasil Optimasi Suhu Annealing Produk PCR Kontrol Daging Sapi Keterangan:
27
Kondisi elektroforesis : Fase diam gel agarosa 1,5%; fase gerak larutan TBE 1X; tegangan 110 Volt, waktu running 30 menit; volume injeksi 5 μL
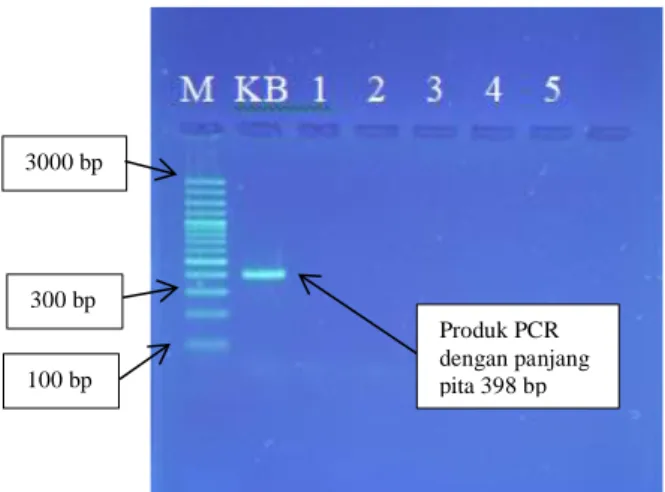
E. Analisis Produk PCR
Hasil produk PCR dianalisis secara kualitatif dengan elektroforesis menggunakan gel agarosa 1,5%. Konsentrasi gel agarosa pada analisis produk PCR lebih besar daripada konsentrasi gel pada analisis isolat DNA. Hal ini disebabkan molekul yang ukurannya lebih kecil akan bermigrasi lebih cepat melalui pori-pori gel dan ukuran DNA produk PCR relatif lebih kecil sehingga rongga untuk molekul bermigrasi saat elektroforesis harus lebih rapat (Irianto, 2017). DNA ladder sebagai marker juga perlu ditambahkan saat elektroforesis. Marker adalah segmen DNA yang spesifik dan telah diketahui ukurannya. Marker berfungsi untuk mengetahui ukuran DNA hasil amplifikasi sehingga amplifikasi dengan PCR dapat diketahui berhasil atau tidak sesuai target (Utami dkk., 2013).
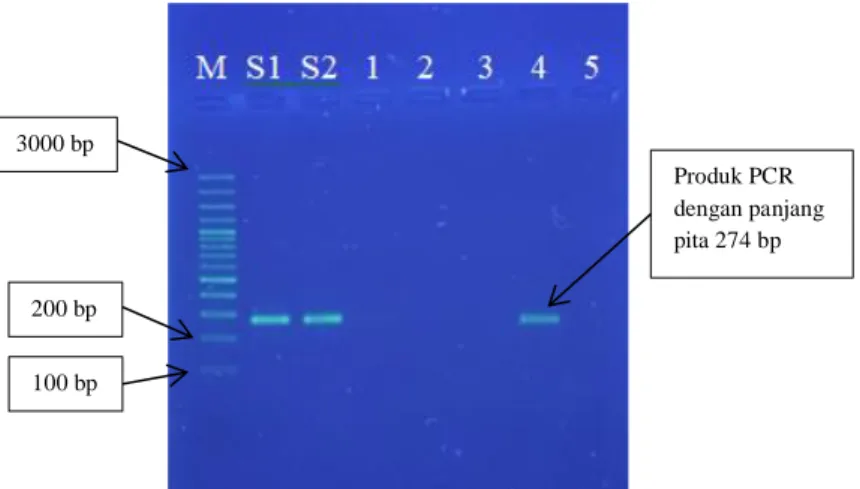
Gambar 8. Hasil Analisis Kualitatif Produk PCR dengan Primer Reverse Sapi Keterangan :
M : DNA ladder (marker)
S1 : Kontrol sapi sebagai pembanding (produk PCR) S2 : Kontrol sapi sebagai penegas (produk PCR) 1-5 : Produk PCR dari isolat DNA kornet sapi
Kondisi elektroforesis : Fase diam gel agarosa 1,5%; fase gerak larutan TBE 1x; tegangan 110 Volt, waktu running 30 menit; volume injeksi 5 μL; DNA
ladder 3 μL Produk PCR dengan panjang pita 274 bp 3000 bp 100 bp 200 bp
Gambar 9. Hasil Analisis Kualitatif Produk PCR dengan Primer Reverse Babi Keterangan :
M : DNA ladder (marker)
KB : Kontrol positif babi (produk PCR) 1-5 : Produk PCR dari isolat DNA kornet sapi
Kondisi Elektroforesis : Fase diam gel agarosa 1,5%; fase gerak larutan TBE 1x; tegangan 110 Volt, waktu running 30 menit; volume injeksi 5 μL; DNA ladder 3 μL
Produk PCR dari isolat DNA dinyatakan positif mengandung babi apabila hasil visualisasi tersebut membentuk pita DNA tunggal dan tebal dengan ukuran 398 bp sesuai dengan kontrol positif babi. Sebaliknya, produk PCR dari isolat DNA dinyatakan mengandung sapi atau negatif mengandung babi apabila hasil visualisasi tersebut membentuk pita DNA tunggal dan tebal dengan ukuran 274 bp sesuai dengan kontrol positif sapi. Hasil elektroforesis produk PCR yang menggunakan primer reverse sapi ditunjukkan pada Gambar 7. Hasil elektroforesis menunjukkan terbentuknya pita berukuran 274 bp pada daging sapi dan sampel kornet sapi. Sedangkan, hasil elektroforesis produk PCR yang menggunakan primer reverse babi ditunjukkan pada Gambar 8. Hasil elektroforesis menunjukkan terbentuknya pita berukuran 398 bp pada daging babi. Ketebalan pita DNA hasil amplifikasi dipengaruhi oleh jumlah DNA yang terisolasi. Semakin banyak DNA yang terisolasi maka semakin tebal pita DNA begitupun sebaliknya (Wijaya dkk., 2018).
Tabel II. Fragmen DNA hasil produk PCR Sampel Kode
Sampel
Primer Teramplifikasi Ukuran pita Daging sapi S1 Reverse sapi 274 bp Daging sapi S2 Reverse sapi 274 bp
Kornet 1 1 Reverse sapi - -
Produk PCR dengan panjang pita 398 bp 3000 bp 100 bp 300 bp
29
Kornet 2 2 Reverse sapi - -
Kornet 3 3 Reverse sapi - -
Kornet 4 4 Reverse sapi 274 bp
Kornet 5 5 Reverse sapi - -
Daging babi KB Reverse babi 398 bp
Kornet 1 1 Reverse babi - -
Kornet 2 2 Reverse babi - -
Kornet 3 3 Reverse babi - -
Kornet 4 4 Reverse babi - -
Kornet 5 5 Reverse babi - -
Pada tabel II disajikan data hasil pengamatan fragmen DNA produk PCR. DNA yang teramplifikasi dengan primer reverse sapi membentuk pita berukuran 274 bp adalah kontrol positif daging sapi dan sampel kornet sapi nomor 4. Pada sampel 1,2,3 dan 5 tidak menunjukkan terjadinya amplifikasi karena tidak terbentuk pita sejajar dengan kontrol positif sapi yaitu 274 bp. Hal ini disebabkan karena DNA terdegradasi akibat proses pemanasan menggunakan suhu >100˚C (Che Man dkk., 2007). Pemanasan menggunakan suhu tinggi >100˚C yang dipengaruhi karena adanya tekanan dari uap air dalam proses autoklaf pada produk daging akan menyebabkan degradasi DNA (Rahayu dkk., 2020). Selain suhu dan tekanan, durasi saat mengolah daging juga mempengaruhi kualitas DNA. Semakin lama durasi dan semakin tinggi suhu yang digunakan untuk mengolah daging maka dapat menyebabkan degradasi DNA, akibatnya DNA pada produk daging olahan terfragmentasi dan menyebabkan DNA menjadi sulit untuk teramplifikasi (Erwanto, 2018). Di samping itu, persentase daging sapi yang ada pada setiap sampel kornet serta proses pembuatan dari setiap pabrik berbeda-beda sehingga hal ini menjadi penyebab terjadinya amplifikasi pada sampel 4 namun tidak pada sampel 1,2 3, dan 5. DNA yang teramplifikasi dengan primer reverse babi membentuk pita berukuran 398 bp adalah kontrol positif daging babi sesuai sekuen target gen sitokrom b babi. Pada sampel produk kornet 1 sampai 5 tidak ada pita yang terbentuk dengan panjang berukuran 398 bp. Proses amplifikasi pada sampel 1 sampai 5 tidak terjadi karena tidak ada primer yang menempel pada DNA template sehingga disimpulkan bahwa gen sitokrom b babi tidak teridentifikasi pada 5 sampel produk kornet sapi.
Hasil ini berbeda dengan penelitian yang dilakukan oleh Wijaya dkk. (2018). Metode yang digunakan pada penelitian Wijaya dan penelitian ini sama
yaitu metode PCR konvensional. Namun, pada penelitian ini tidak semua sampel kornet sapi teramplifikasi dengan primer reverse sapi ataupun primer reverse babi, sedangkan pada penelitian Wijaya dkk. (2018) semua sampel kornet sapi dapat teramplifikasi. Faktor yang dapat mempengaruhi perbedaan antara penelitian ini dan penelitian yang dilakukan oleh Wijaya dkk. (2018) adalah jumlah kandungan daging sapi pada produk kornet sapi yang berbeda-beda dan pengolahan kornet sapi yang menyebabkan DNA pada beberapa sampel telah terdegradasi akibat proses autoklaf sebelum dilakukannya isolasi DNA. Dengan demikian, hal ini merupakan kondisi yang tidak terduga dan tidak mampu diatasi (force major).
Metode isolasi DNA yang digunakan oleh Wijaya dkk. (2018) adalah metode Phenol Chloroform Isoamil Alcohol (PCIA) yang disebut juga metode ekstraksi DNA konvensional (tanpa kit), sedangkan pada penelitian ini digunakan metode isolasi DNA dengan kit atau disebut juga metode Double Spin Column. Perbedaan antara kedua metode ini yaitu pada metode PCIA, esktrak DNA yang diperoleh berupa pellet transparan dapat teramati secara langsung sehingga mempermudah peneliti untuk memastikan keberadaan ekstrak DNA sedangkan metode Double Spin Column menggunakan membran silika untuk memerangkap DNA yang telah keluar dari sel (Marwayana, 2015). Namun, perbedaan metode isolasi tidak mempengaruhi hasil isolat DNA kornet sapi secara signifikan karena pada proses isolasi DNA menggunakan kedua metode ini tetap melalui tahapan penambahan etanol absolut. Menurut Aini dkk. (2011), DNA yang terdegradasi menjadi untai tunggal atau nukleotida bebas larut dalam etanol pada saat proses pencucian sehingga dapat mengurangi jumlah DNA yang diperoleh. Dengan demikian, faktor adanya perbedaan metode isolasi DNA antara metode PCIA dan metode Double Spin Column tidak mempengaruhi hasil isolasi DNA.
31 BAB V
KESIMPULAN DAN SARAN
A. Kesimpulan
Berdasarkan penelitian yang telah dilakukan, dapat ditarik kesimpulan sebagai berikut :
1. Metode PCR menggunakan gen sitokrom b babi memiliki kemampuan untuk membedakan secara spesifik kandungan babi dan sapi pada produk kornet dengan ukuran pita 398 bp untuk babi dan 274 bp untuk sapi.
2. Kelima produk kornet sapi yang diperoleh dari supermarket wilayah Yogyakarta tidak ditemukan adanya kandungan babi.
B. Saran
1. Penelitian lebih lanjut dapat dilakukan dengan menggunakan marker dari mitokondria DNA selain gen sitokrom b
2. Penelitian lebih lanjut dapat dilakukan menggunakan metode PCR seperti droplet digital (ddPCR) yang mampu mendeteksi secara akurat dan sensitif meskipun melalui proses pemanasan dan tekanan dalam daging olahan
DAFTAR PUSTAKA
Aini, A.N., Ria, P., Aminin, A.L.N., 2011. Pemurnian DNA Plasmid Puc19 Menggunakan Kolom Silika dengan Denaturan Urea. Jurnal Sains dan Matematika, 19(2), 47–53.
Andriyani, E., Fais, N.L., Muarifah, S., 2019. Perkembangan Penelitian Metode Deteksi Kandungan Babi untuk Menjamin Kehalalan Produk Pangan Olahan. Journal of Islamic Studies and Humanities, 4(1), 104–126.
Bintang, M., 2018. Biokimia : Teknik Penelitian, 2nd ed. Penerbit Erlangga, Jakarta.
Butler, J.M., 2005. Forensic DNA Typing, Elsevier Academic Press.
Cahyono, H.B., Yuliastuti, R., Amanati, L., 2018. Pengaruh Penggorengan Terhadap Kandungan Nitrit Dalam Kornet. Jurnal Teknologi Proses dan Inovasi Industri, 3(2), 57–62.
Calderón-Franco, D., Lin, Q., van Loosdrecht, M.C.M., Abbas, B., Weissbrodt, D.G., 2020. Anticipating Xenogenic Pollution at the Source: Impact of Sterilizations on DNA Release From Microbial Cultures. Frontiers in Bioengineering and Biotechnology, 8(171), 1–13.
Chandrika, M., Maimunah, M., Zainon, M.., Son, R., 2010. Identification of The Species Origin of Commercially Available Processed Food Products by Mitochondrial DNA Analysis. International Food Research Journal, 17(1), 867–875.
Che Man, Y.B., Aida, A.A., Raha, A.R., Son, R., 2007. Identification of Pork Derivatives in Food Products by Species-Specific Polymerase Chain Reaction (PCR) for Halal Verification. Food Control, 18(7), 885–889.
Che Man, Y.B., Mustafa, S., Khairil Mokhtar, N.F., Nordin, R., Sazili, A.Q., 2012. Porcine-Specific Polymerase Chain Reaction Assay Based on Mitochondrial D-loop Gene for Identification of Pork in Raw Meat. International Journal of Food Properties, 15(1), 134–144.
Crisafuli, F.A.P., Ramos, E.B., Rocha, M.S., 2015. Characterizing the Interaction between DNA and GelRed Fluorescent Stain. European Biophysics Journal, 44(1), 1–7.
Desler, C., Marcker, M.L., Singh, K.K., Rasmussen, L.J., 2011. The Importance of Mitochondrial DNA in Aging and Cancer. Journal of Aging Research, 2011(17), 1–10.
Erwanto, Y., 2018. Molecular Based Method Using PCR Technology on Porcine Derivative Detection for Halal Authentication, in: Abdurakhmonov, I. (Ed.), Genotyping. IntechOpen, pp. 65–84.
Fadhlurrahman, Wardani, A.K., Endrika, W., 2015. Deteksi Gelatin Babi Pada Soft Candy Menggunakan Metode PCR-RFLP Sebagai Salah Satu Pembuktian Kehalalan Pangan. Jurnal Teknologi Pertanian, 16(2), 81–88. Farias, I.P., Ortí, G., Sampaio, I., Schneider, H., Meyer, A., 2001. The
Cytochrome b Gene as a Phylogenetic Marker: The Limits of Resolution for Analyzing Relationships Among Cichlid Fishes. Journal of Molecular Evolution, 53(2), 89–103.