Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
SKRIPSI
Diajukan untuk Memenuhi Sebagian dari Syarat
untuk Memperoleh Gelar Sarjana Sains Program Studi Biologi
Oleh:
Vania Febriyantie
1005103
PROGRAM STUDI BIOLOGI JURUSAN PENDIDIKAN BIOLOGI
FAKULTAS PENDIDIKAN MATEMATIKA DAN ILMU PENGETAHUAN ALAM UNIVERSITAS PENDIDIKAN INDONESIA
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
STUDI KEKERABATAN FENETIK BEBERAPA JENIS
TANAMAN SAWO
Pouteria
(SAPOTACEAE)
MENGGUNAKAN METODE RAPD
Oleh Vania Febriyantie
Sebuah skripsi yang diajukan untuk memenuhi salah satu syarat memperoleh gelar Sarjana pada Fakultas Pendidikan Matematika dan Ilmu Penetahuan Alam
© Vania Febriyantie 2014 Universitas Pendidikan Indonesia
Oktober 2014
Hak Cipta dilindungi undang-undang.
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu LEMBAR PENGESAHAN
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Oleh
Vania Febriyantie
1005103
DISETUJUI DAN DISAHKAN OLEH: Pembimbing I
Dr. Topik Hidayat, M.Si NIP. 197004101997021001
Pembimbing II
Nuryani Y Rustaman, Prof.Dr.M.Pd NIP. 195012311979032029
Pembimbing III
Teguh Triono,Ph.D Tanri Abeng University
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
Ketua Jurusan Pendidikan Biologi FPMIPA UPI
Dr. Riandi, M.Si NIP. 196305011988031002
PERNYATAAN
Dengan ini saya menyatakan bahwa skripsi dengan judul “STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD” ini beserta seluruh isinya adalah benar-benar karya sendiri, dan saya tidak melakukan penjiplakan atau pengutipan dengan cara-cara yang tidak sesuai dengan etika keilmuan yang berlaku dalam masyarakat keilmuan. Atas pernyataan ini, saya siap menanggung sanksi yang dijatuhkan kepada saya apabila kemudian ditemukan adanya pelanggaran terhadap etika keilmuan dalam karya saya ini atau klaim dari pihak lain terhadap keaslian karya saya ini.
Bandung, September 2014
Yang membuat pernyataan
Vania Febriyantie
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE (RAPD)
ABSTRAK
Pouteria duclitan merupakan tanaman yang termasuk keluarga sawo-sawoan
(famili Sapotaceae). Tanaman ini terdistribusi hampir di seluruh negara tropis termasuk Indonesia, salah satunya dapat ditemukan di Kebun Raya Bogor. Namun, beberapa tanaman yang tumbuh di area dan kondisi lingkungan yang sama yaitu Kebun Raya Bogor memiliki karakter morfologi buah yang berbeda di bandingkan dengan Pouteria duclitan lainnya. Tujuan penelitian ini yaitu untuk menganalisis kekerabatan fenetik beberapa jenis tanaman sawo Pouteria (Sapotaceae) menggunakan metode RAPD (Random Amplified Polymorphic
DNA). Hasil penelitian menunjukkan bahwa primer acak OPB-09 memiliki
persentase tingkat polimorfik yang tinggi sebesar 72,42%, dan berhasil memperoleh 52 larik DNA diantaranya 13 larik monomorfik dan 39 larik polimorfik dari ukuran basa 408 - 3500 pb. Analisis fenetik dengan menggunakan
software MEGA 4 berhasil mengklasifikasi keenam sampel tanaman sawo Pouteria dengan ditandai adanya dua kelompok besar yang terbentuk. P. obovata
dan P. campechiana berada dalam satu kelompok besar, sedangkan beberapa tanaman P. duclitan berada dalam satu kelompok yang berbeda. Hal ini mengindikasikan bahwa P. obovata dan P. campechiana berkerabat jauh dengan
P. duclitan. Data Fenogram juga menunjukkan bahwa P. duclitan 112 A yang
memiliki karakter morfologi buah yang berbeda termasuk ke dalam varietas baru, tetapi pada P. duclitan XXV.B.120 status kekerabatannya belum jelas.
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
STUDY PHENETIC OF SEVERAL PLANTS Pouteria (Sapotaceae) USING
RANDOM AMPLIFIED POLYMORPHIC (RAPD) METHOD
ABSTRACT
Pouteria duclitan is a member of Sapotaceae family and distributed almost in
tropical country, including Indonesia. In Indonesia, P.duclitan trees could be found in Bogor Botanical Garden, West Java. Some of P. duclitan trees which grow in the same area and environmental conditions has different morphological of fruits compared with another P.duciltan trees. The purpose of this research is to analyse phenetic relationship between several plants Pouteria (Sapotaceae) using RAPD (Random Amplified Polymorphic DNA) method. The result showed that arbitrary primer OPB-09 has high percentage of polymorphic rate was 72,42% and successfully amplified 52 locci, including 13 locci monomorphic and 39 locci polymorphic of base size 408 – 3500 bp. Phenetic analyse using MEGA 4 software successfully classified several sampels of Pouteria into two major groups formed. P. obovata and P campechiana are in the same group, whereas all of P. duclitan samples are in a different group. This indicates that P. obovata and
P. campechiana distantly related to P duclitan. Furthermore, the phenogram also
demonstrated that P. duclitan 112 A is likely to be a new cultivar, but P. duclitan XXV.B.120 remains unclear.
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu DAFTAR ISI
C. Pertanyaan Penelitian ... 4
D. Tujuan Penelitian ... 4
E. Manfaat Penelitian ... 5
F. Batasan Penelitian ... 5
BAB II BIOSISTEMATIKA SAPOTACEAE MENGGUNAKAN PENANDA MOLEKULAR RAPD ... 7
A. Biositematika………..7
1. Analisis Fenetik………7
2. Analisis Filogenetik ... 8
B. Dasar-dasar Teknik Biologi Molekuler ... 10
1. Esktraksi DNA Tanaman……….10
2. Uji Kuantitatif DNA……….11
3. Uji Kualitatif DNA………...13
4. Teknik Dasar Amplifikasi PCR (Polymerase Chain Reaction)…...16
C. Penanda Molekuler………20
1. Penanda Kodominan (co-dominant markers)………..20
a. RFLP (Restriction Fragment Length Polymorphisms)…………20
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
2. Penanda Dominan (dominant markers)………...24
a. AFLP (Amplified Fragment Length Polymorphism)………..…24
b. RAPD (Random Amplified Polymorphic DNA)….………26
D. Sapotaceae ... 31
1. Pouteria campechiana ... 33
2. Pouteria duclitan ... 35
3. Pouteria obovata ... 37
E. Penelitian Terkait dengan Pouteria………..38
F. Penelitian Terkait dengan Metode RAPD (Random Amplified Polymorphic DNA)………39
BAB III METODE PENELITIAN ... 42
A. Jenis Penelitian ... 42
B. Populasi dan Sampel ... 42
C. Waktu dan Tempat Penelitian ... 42
D. Alat dan Bahan ... 42
E. Prosedur Penelitian ... 42
1. Tahap Persiapan ... 42
2. Tahap Penelitian ... 43
a. Pengambilan Sampel... 43
b. Isolasi DNA Genom Tanaman ... 43
c. Uji Kuantitatif DNA ... 46
d. Uji Kualitatif DNA (Elektroforesis) ... 46
e. Amplifikasi DNA dengan Metode PCR-RAPD ... 46
f. Uji Kualitatif Hasil PCR………..46
3. Analisis Data………..…48
F. Alur Penelitian ... 50
BAB IV HASIL PENELITIAN DAN PEMBAHASAN ... 51
A. Isolasi DNA Genom Tanaman Sawo Pouteria ... 51
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
1. Optimasi Kondisi PCR ... 55
2. Seleksi Primer Acak ... 56
3. Amplfikasi DNA dengan Primer Acak OPB-09 ... 57
4. Data Matriks Hasil Interpretasi Larik dan Analisis Data Polimorfisme DNA...58
5. Nilai Polymorphic Information Content (PIC) Primer OPB-09….61 6. Klaster UPGMA (Unweighted Pair Group Method with Arithmetic Average) ... 62
BAB V KESIMPULAN DAN SARAN ... 68
A.Kesimpulan ... 68
B. Saran ... 68
DAFTAR PUSTAKA ... 69
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu DAFTAR TABEL
Tabel Halaman
2. 1 Urutan Pengenalan untuk Beberapa Enzim Restriksi Endonuklease ... 22
2.2 Perbandingan Beberapa Jenis Penanda Genetik ... 30
3.1 Komposisi Reaksi PCR berdasarkan Williams, et al. (1990) ... 47
4.1 Konsentrasi dan Kemurnian Isolasi DNA Tanaman Sawo Pouteria ... 53
4.2 Jumlah Larik Monomorfik dan Polimorfik keenam Sampel Tanaman Sawo Pouteria ... 59
4.3 Data Matriks Hasil Amplifikasi DNA Tanaman Sawo Pouteria...60
4.5 Perhitungan Nilai PIC dari primer OPB-09………...61
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu DAFTAR GAMBAR
Gambar Halaman
2.1 (A). Pohon Fenetik dan (B). Pohon Kladistik...9
2.2 Contoh Alat Spektrofotometer UV ... 12
2.3 Prinsip Kerja Alat Spektrofotometer UV ... 12
2.4 Berbagai Macam Alat Elektroforesis ... 13
2.5 Prinsip Kerja Elektroforesis ... 14
2.6 Prinsip Kerja PCR (Polymerase Chain Reaction) ... 16
2.7 Thermocycler ... 19
2.8 Langkah Kerja Penanda Molekuler AFLP ... 25
2.9 Skema Prinsip Kerja RAPD ... 28
2.10 Pouteria campechiana, (a) pohon ketika berbuah, (b) daging buah, (c) daun ... 33
2.11 Pouteria duclitan,(a) Habitus, (b) Perbungaan, (c) Pertulangan daun, dan (d) Buah ... 35
2.12 Anatomi Serat Batang Pouteria duclitan (Blanco) ... 36
2.13 Pouteria obovata,(a) kecambah, (b) daun anakan, (c) buah, (d) biji ... 37
3.1 Skema Langkah Kerja Esktraksi DNA Tanaman... 45
3.2 Program Amplifikasi Fragmen DNA menggunakan Thermocylcer ... 48
3.3 Diagram Alur Penelitian ... 50
4.1 Elektroforegram DNA enam sampel tanaman sawo Pouteria dari jaringan daun muda ... 51
4.2 Elektroforegram Hasil Amplifikasi DNA Tanaman Sawo PouteriaMenggunakan Primer Acak OPB-09 ... 58
4.3 Ilustrasi Interpretasi Elektroferogram Hasil Ampilifikasi keenam sampel Tanaman Sawo Pouteria menggunakan primer OPB-09 ... 59
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu DAFTAR LAMPIRAN
Lampiran Halaman
1 Daftar Alat dan Bahan………. 77
2 Protokol Pembuatan Larutan Stok……….. 80
3 Cara Menghitung Panjang Molekul DNA Hasil
Amplifikasi……….. 82
4 Surat Perizinan Pengambilan Sampel 84
5 Foto Kegiatan……….. 88
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu
1 BAB I
PENDAHULUAN
A. Latar Belakang
Indonesia merupakan negeri khatulistiwa yang terdiri dari bentangan luas
lautan dan sekitar 13.000 pulau-pulau yang berjajar dari ujung Sabang sampai
Merauke. Iklim tropis menjadikan tanah Indonesia subur dan berpotensi
ditumbuhi berbagai macam jenis tumbuhan. Indonesia memiliki hutan tropis
terbesar ke-3 setelah Brazil dan Republik Demokrasi Kongo (Zaire). Hutan
tropis ini sangat kaya akan kenekaragaman hayati yang unik (Forest Watch
Indonesia, tanpa tahun) khususnya pada keanekaragaman floranya.
Sebagai negara tropis, Indonesia memiliki banyak jenis buah-buahan, baik
yang asli dari Indonesia maupun jenis-jenis introduksi. Kelompok sawo-sawoan
atau famili Sapotaceae merupakan salah satu kelompok buah-buahan yang
tumbuh dan berkembang dengan baik di Indonesia. Keanekaragaman jenis
sawo-sawoan di Indonesia merupakan gabungan antara jenis-jenis asli Indonesia dan
jenis-jenis introduksi dari Asia dan Amerika yang dibawa masuk ke Indonesia
berabad-abad lampau (Triono, 2000).
Buah sawo sering dijadikan bahan pangan, diantaranya adalah sawo Belanda
(Pouteria campechiana) dan sawo Manila (Manilkara zapota). Buah ini sangat
digemari oleh banyak kalangan karena buahnya yang matang memiliki aroma
dan rasa yang manis, tekstur buahnya pun halus. Selain itu buah sawo juga
bermanfaat untuk kesehatan karena memiliki zat antioksidan yang tinggi (Kong
2
Tanaman-tanaman yang termasuk ke dalam Famili Sapotaceae sangat
memberi manfaat bagi kebutuhan hidup manusia. Buahnya yang bermanfaat
untuk sumber pangan, kayunya juga dapat dijadikan sebagai bahan bangunan dan
alat-alat furniture. Masyarakat Jawa sering memanfaatkan kayu Palaquium
javanensis (sinonim dari Palaqium amboinense) untuk membuat alat musik
gamelan dan kerajinan tangan khas daerah mereka (Moon et al., 2011). Manfaat
lainnya adalah produksi getah perca yang sangat melimpah pada ekstraksi daun
dan getah batang pohon genus Palaquium terutama Palaquium gutta Burck dan
Palaquium oblongifolium Burck (Karliati et al., 2011). Getah perca yang
dihasilkan berguna untuk perekat pelapis kayu (Karliati et al., 2011), bahan baku
permen karet dan membungkus kabel di bawah laut (Moon et al., 2011).
P.duclitan merupakan salah satu jenis tanaman sawo yang termasuk ke
dalam famili Sapotaceae, terdistribusi luas di Sumatra, Kalimantan, Bali,
Lombok, Jawa dan sekitarnya (Moon et al., 2011). Kayunya seringkali
dimanfaatkan untuk membuat pahatan kayu dan figura. Namun, menurut
penelitian Krisdianto (2000) kualitas kayu P. duclitan kurang kuat dan tidak
tahan lama, sehingga jarang sekali digunakan untuk bahan bangunan (Moon et
al., 2011)
P.duclitan juga turut menghiasi area Kebun Raya Bogor. Telah dilakukan
pengamatan pendahuluan mengenai karakterisik morfologi beberapa individu
P.duclitan yang tumbuh satu area dan kondisi yang sama yaitu di Kebun Raya
Bogor. Hasilnya salah satu individu P.diclitan cenderung memiliki perbedaan
pada karakter morfologi buah. Namun, dari segi morfologi daun, bunga, tipe
percabangan batang secara keseluruhnya sama. Perbedaan sifat yang teramati
(fenotipe) seperti bentuk morfologi buah, warna buah dan rasa buah dapat
dipengaruhi oleh banyak faktor (multifactorial) baik genetik maupun lingkungan
(Campbell et al., 2010). Dalam pandangan yang terintegrasi tentang pewarisan
sifat dan variasi gen, fenotipe suatu organisme mencerminkan keseluruhan
3
tersebut diduga individu P.duclitan tersebut memiliki keanekaragaman genetik
yang berbeda dan kemungkinan dapat dikategorikan ke dalam varietas yang
berbeda. Sesuai dengan Undang-undang No.29 Tahun 2000 Pasal 1 ayat 3
tentang perlindungan varietas tanaman yaitu sekelompok tanaman dari suatu
jenis atau spesies ditandai oleh bentuk tanaman, pertumbuhan tanaman, daun
bunga, biji dan ekpresi genotipe/kombinasi genotipe yang dapat membedakan
dari jenis atau spesies yang sama oleh sekurang-kurangnya satu sifat yang
menentukan dan apabila diperbanyak tidak mengalami perubahan.
Diperlukan klasifikasi secara molekuler sebagai penunjang atau alternatif
untuk mengetahui tingkat keragaman dan perbedaan genetik pada tanaman sawo
ini, sehingga dapat ditemukan seberapa besar tingkat kekerabatan tanaman sawo
P. duclitan yang memiliki karakter morfologi yang berbeda dengan P. duclitan
lainnya. Untuk menganalisis keanekragaman genetik dan kekerabatan genetik
suatu organism e dibutuhkan penanda molekuler. Penanda molekuler beberapa
diantaranya adalah penanda tingkat protein dan penanda tingkat DNA. Penanda
tingkat protein memiliki keterbatasan dalam jumlahnya karena penanda ini masih
dipengaruhi oleh beberapa faktor seperti lingkungan, apabila kondisi
lingkungannya berbeda gen akan mengekspresikan protein yang berbeda pula
sesuai dengan kondisi lingkungannya, sehingga pendanda ini tidak cukup
informatif untuk penelitian yang mencakup seluruh genom (Semagn et al., 2006;
Asins et al., 1995 dalam Karsinah et al., 2002). Penanda molekuler yang
digunakan dalam penelitian ini adalah penanda tingkat DNA. Penanda tingkat
DNA sifatnya lebih stabil, dapat dideteksi di dalam semua jaringan tubuh dan
tidak dipengaruhi oleh lingkungan (Semagn et al., 2006; Zulfahmi, 2013).
Penanda tingkat DNA yang digunakan untuk tanaman khususnya untuk
mendeteksi polimorfisme terdiri dari banyak tipe di antaranya Restriction
Fragment Length Polymorphism (RFLP), Random Amplified Polymorphic DNA
(RAPD), Amplified Fragment Length Polymorphism (AFLP), Inter-Simple
4
Tag Sites (STSs), Cleaved Amplified Polymorphic Sequences (CAPS),
Microsatellites atau Simple Sequence Repeats (SSRs), Expressed Sequence
Tags (ESTs), Single Nucleotide Polymorphisms (SNPs), dan Diversity Arrays
Technology (DArT).
Pada penelitian ini digunakan penanda RAPD (Random Amplified
Polymorphic DNA). Penanda ini pertama kali diperkenalkan oleh Williams et al
(1990). Namun seiring berjalannya waktu, penanda RAPD menjadi semakin
populer. Penanda ini sering dipakai untuk meneliti dan menganalisis genom
khususnya pada tanaman. Penanda molekular RAPD dipilih karena memiliki
keunggulan diantaranya pada proses pengerjaannya dapat memakai DNA sampel
yang tidak terlalu murni (Bakkapa et al., 2011; Semagn, 2006), membutuhkan
estimasi biaya yang relatif rendah (Bakkapa et al., 2011), langkah pengerjaannya
mudah dan sederhana sehingga tidak membutuhkan waktu yang lama untuk
mendapatkan hasil (Kumar et al., 2009), dan primer dapat digunakan untuk
analisis genom pada berbagai spesies (Semagn et al.¸2006).
Beberapa penelitian yang terkait dengan RAPD dan pemakaian tanaman
sebagai objek penelitiannya adalah genetic mapping berbasis RAPD pada
Passiflora edulis (Carneiro, 2002). Penelitian Zhang et al. (2013) tentang
analisis keanekaragaman genetik Larix gmelinii (Pinaceae), mengkaji genotoksik
efek Boron pada Triticum aestivum L. dan Phaseolus vulgaris L. (Kekec et al.,
2010), analisis keragaman genetik kultivar Citrus sinensis L. Osbeck
menggunakan karakteristik morfologi dan penand RAPD (Malik et al., 2012).
Seperti pada penelitian Majourhat et al. (2009) yang menggunakan Argania
spinosa (Sapotaceae) sebagai objek penelitiannya. Salah satu tujuan
penelitiannya adalah untuk mengetahui tingkat kekerabatan dan keanekaragaman
genetik Argania spinosa yang sebelumnya sudah diidentifikasi beberapa individu
memiliki bentuk morfologi buah yang berbeda yaitu bulat, gelondong dan oval
menggunakan penanda RAPD (Random Amplified Polymorphic DNA) dan SSR
5
Penelitian mengenai karakter molekular pada tanaman sawo Pouteria sudah
dilakukan sebelumnya oleh Rojas et al. (2012) dengan mengambil Pouteria
sapota sebagai objek penelitiannya dan menggunakan data RAPD untuk
menganalisis keragaman genetiknya. Pada jenis Pouteria duclitan masih belum
ditemukan penelitian mengenai karakter molekulernya, sehingga memang
diperlukan penelitian yang berkaitan untuk menambahkan data biologi molekuler
khususnya pada tanaman genus Pouteria.
B. Rumusan Masalah
Rumusan masalah dalam penelitian ini adalah: “Bagaimana hubungan kekerabatan fenetik beberapa jenis tanaman sawo Pouteria (Sapotaceae)
menggunakan metode RAPD?”
C. Batasan Masalah
Agar penelitian ini tidak meluas maka ada beberapa batasan masalah sebagai
berikut.
a. Beberapa spesimen merupakan jenis P. duclitan yang diambil di Kebun
Raya Bogor.
b. Penelitian ini menggunakan beberapa spesies Pouteria lain yang dijadikan
sebagai individu pembanding, yaitu P. obovata dan P. campechiana.
c. Primer acak yang digunakan untuk mengamplifikasi adalah OPB-09 dan
OPB-10.
D. Tujuan
Adapun tujuan yang ingin dicapai dalam penelitian ini adalah menganalisis
hubungan kekerabatan beberapa jenis tanaman sawo Pouteria (Sapotaceae)
menggunakan metode RAPD berdasarkan studi fenetik.
6
Hasil penelitian ini memiliki beberapa manfaat, diantaranya adalah sebagai
berikut.
a. Menemukan hubungan kekerabatan antara beberapa jenis tanaman sawo
Pouteria yang informasinya berguna untuk dilakukanya perkawinan silang
untuk menghasilkan varietas-varietas yang unggul.
b. Menghasilkan data molekuler yang bermanfaat bagi pengembangan
sumber plasma nutfah yang ada di Indonesia khususnya pada tanaman
sawo jenis Pouteria.
c. Sebagai tambahan ilmu dalam bidang biologi khususnya mengenai studi
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu BAB III
METODE PENELITIAN
A. Jenis Penelitian
Jenis penelitian ini adalah penelitian deskriptif yang mengangkat
fenomena alam sebagai salah satu masalah dalam penelitian. Penelitian ini
dapat menerangkan arti dan kejelasan terhadap fenomena tersebut.
B. Populasi dan Sampel
Populasi dalam penelitian ini adalah beberapa tanaman P.duclitan,
P.obovata dan P.campechiana di Kebun Raya Bogor sedangkan sampelnya
adalah DNA beberapa tanaman P.duclitan, P.obovatadan P.campechiana
yang telah diisolasi. Sumber DNA berasal dari jaringan daun muda beberapa
tanamanP.duclitandan tanaman pembanding yaitu P.obovatadan
P.campechiana.
C. Waktu dan Lokasi Penelitian
Penelitian dilaksanakan dari bulan Februari sampai dengan bulan
September 2014. Lokasi penelitian terbagi menjadi dua, yaitu: pengambilan
sampel di Kebun Raya Bogor dan analisis genetik di laboratorium
Mikrobiologi FPMIPA UPI.
D. Alat dan Bahan
Alat dan bahan yang digunakan dalam penelitian terdapat di
Laboratorium Mikrobiologi Universitas Pendidikan Indonesia. Daftar alat
dan bahan yang digunakan tercantum dalam Lampiran 1.
43
Alat-alat yang digunakan dicuci terlebih dahulu dan dikeringkan
kemudian dibungkus dengan plastik tahan panas yang selanjutnya
disterilisasi panas lembab. Begitu juga bahan yang digunakan disterilisasi
menggunakanautoclave dengan suhu 121°C tekanan 1,5 atm selama 10-15
menit. Pembuatan larutan yang digunakan tercantum pada Lampiran 2.
2. Tahap Penelitian a. Pengambilan Sampel
Sampel yang digunakan adalah daun muda dari beberapa tanaman
P.duclitan, P.obovatadan P.campechiana. Daun tersebut disimpan pada
plastic bagdan dimasukkan ke dalam coolbox yang sudah diisi dengan
es batu sebelumnya agar daun tetap segar. Kemudian di dalam
laboratorium, daun tersebut dicuci dengan air mengalir dan dikeringkan
menggunakan tissue. Selanjutnya sampel daun ditimbang seberat
0,1-0,5gram dari setiap tanaman P.duclitan, P.obovata dan P.campechiana
yang tercuplik dan selanjutnya disimpan pada suhu -20°C hingga proses
ekstraksi DNA.
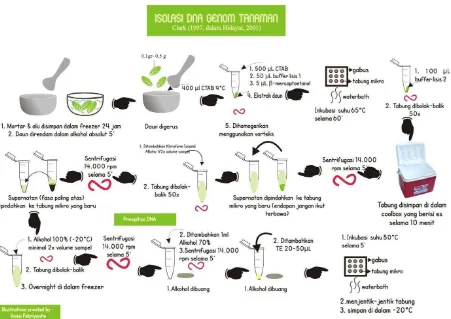
b. Isolasi DNA Genom
Isolasi DNA yang dilakukan berdasarkan metode Clark (1997, dalam
Hidayat, 2001)dengan beberapa modifikasi. Mortar dan alu yang
digunakan disimpan di dalamfrezeer selama 24 jam untuk memudahkan
menggerus daun. CTAB (1 M Tris-HCl pH 8,0; 6 M NaCl; 0,5 M EDTA
pH 8,0; 2% CTAB; 2% SDS; ddH2O steril) yang digunakan terdiri dari
CTAB dingin 4°C dan CTAB hangat 65°C. Sebanyak 0,1-0,5 gram daun
muda masing-masing sampeldirendam dalam alkohol absolut selama 5
menit. Sementara itu, tabung mikro 1,5 mL steril diberi label sesuai
dengan kode masing-masing sampel dan dimasukkan ke dalamnya
500µL CTAB hangat (sebelumnya dilarutkan dulu menggunakan
magnetic stirrer dalam suhu 65°C), buffer lisis 1 (20% SDS)50µL dan β-mercaptoetanol sebanyak 1% dari CTAB hangat yang digunakan.
Penggerusan daun dimulai dengan memasukkan daun muda yang
benar-44
benar halus, dimasukkan 400µL CTAB dingin dan penggerusan
dilanjutkan hingga daun benar-benar halus. Setelah halus, daun tersebut
dimasukkan ke dalam tabung mikro 1,5 ml yang telah diberi label dan
diisi dengan larutan CTAB, buffer lisis 1 serta β-mercaptoetanol. Sampel dihomogenkan dengan menggunakan vortex. Setelah itu sampel
diinkubasi dalam penangas air pada suhu 65°C selama 60 menit.
Selanjutnya ditambahkan buffer lisis 2 (5 M Potassium asetat) ke
dalam tabung mikro yang berisi sampel sebanyak 100µL dan
dihomogenkan dengan cara membolak-balik tabung sebanyak 50 kali.
Simpan tabung mikro tersebut dalam wadah yang berisi es selama 10
menit, lalu disentrifugasi sampel dengan kecepatan 14.000 rpm selama 5
menit. Pindahkan fasa atas (supernatan) ke dalam tabung mikro yang
baru. Ditambahkan larutan kloroform isoamil alkohol sebanyak 0,5 kali
dari volume total sampel dan dihomogenkan dengan cara membolak
balik tabung sebanyak 50 kali. Selanjutnya sampel disentrifugasi lagi
dengan kecepatan 14.000 rpm selama 5 menit. Fasa paling atas
(supernatan) dimasukkan ke dalam tabung mikro yang baru dan
ditambahkan alkohol 100% (-20°C) sebanyak minimal 2 kali volume
total sampel atau hingga 1,5 ml dan dihomogenkan larutan tersebut
dengan cara membolak-balik tabung sebanyak 50 kali. Sampel tersebut
disimpan dalam freezer(-20°C) selama 24 jam.
Setelah 24 jam, dilakukan proses presipitasi DNA. Sampel
disentrifugasi dengan kecepatan 14.000 rpm selama 5 menit dan dibuang
alkoholnya. Ditambahkan 1 ml alkohol 70% dan dibolak-balik tabung
tersebut. Sampel disentrifugasi lagiselama 5 menit dengan kecepatan
14.000 rpm,alkoholnya dibuang dan tabung dibalikkan di atas tisu
hingga tidak ada lagi alkohol di dalam tabung. Selanjutnya sampel
ditambahkan pelarut DNA TE sebanyak 30µL dansampel diinkubasi
dalam penangas air dengan suhu 50°C selama 5 menit agar DNA dapat
larut. Terakhir, sampel siap digunakanuntuk masuk ke tahap pengukuran
(-45
20°C) apabila sampel belum digunakan. Secara singkat langkah kerja
isolasi DNA genom dideskripsikan dalam Gambar 3.1.
46
c. Mengukur Kemurnian dan Konsentrasi DNA
Uji kuantitatif DNA dilakukan menggunakan spektrofotometer.
Komposisi larutannya yaitu 5 µL DNA dan 495 µL ddH2O steril.
Larutan tersebut dihomogenkan terlebih dahulu menggunakan alat
vortex baru kemudian dimasukkan ke dalam kuvet spektrofotometer.
Kemurnian DNA diukur dengan menghitung nilai absorbansi 260 nm
dibagi dengan nilai absorbansi 280 nm. Nilai kemurnian DNA biasanya
berkisar antara 1,8-2,0. Jika kemurnian DNA kurang dari 1,8 maka
indikasi adanya kontaminan dari protein dan UV sedangkan jika
kemurnian DNA lebih dari 2,0 maka indikasi adanya kontaminan
kloroform dan fenol.Konsentrasi DNA dihitung menggunakan rumus :
[DNA] = Å260 x 50 x faktor pengenceran
Keterangan :
Å260 : Nilai absorbansi pada 260 nm
50 : larutan dengan nilai absorbansi 1,0 sebanding dengan 50μg untai ganda DNA per ml
d. Elektroforesis Sampel Hasil Isolasi DNA
Uji kualitatif DNA dilakukan dengan menggunakan elektroforosesis.
DNA sampel yang akan diamplifikasi terlebih dahulu dielektroforesis
agar diketahui ketebalan fragmen DNA tersebut. Proses elektroforesis
dilakukan dengan cara membuat gel agarose terlebih dahulu. Gel
agarose yang digunakan konsentrasinya 1,4% dalam 25 ml TBE.
Sebanyak 2 µL DNA dicampurkan dengan 1 µL Loading dye dan
kemudian dimasukkan ke dalam sumur-sumur gel. Proses elektroforesis
ini menggunakan larutan TBE dan di-runningdengan 100 volt selama 30
menit. DNA hasil elektroforesis diamati pada UV transluminator lalu
didokumentasikan menggunakan kamera yang ditutupi kertas mika.
e. Amplifikasi DNA dengan Metode PCR
Amplifikasi ini dilakukan dengan menggunakan mesin thermocycler
dengan program Gene Amplified PCR system 9700. Primer yang
47
3’dan OPB-10 dengan sikuen data 5’ CTGCTGGGAC 3’, primer ini diadaptasi dari Rojas et al. (2012).
Pencampuran komponen reaksi PCR dilakukan secara cepat dan
teliti. Semua proses pencampuran dilakukan di dalam coolbox untuk
menjaga agar komponen reaksi PCR tidak rusak. Setiap tabung PCR
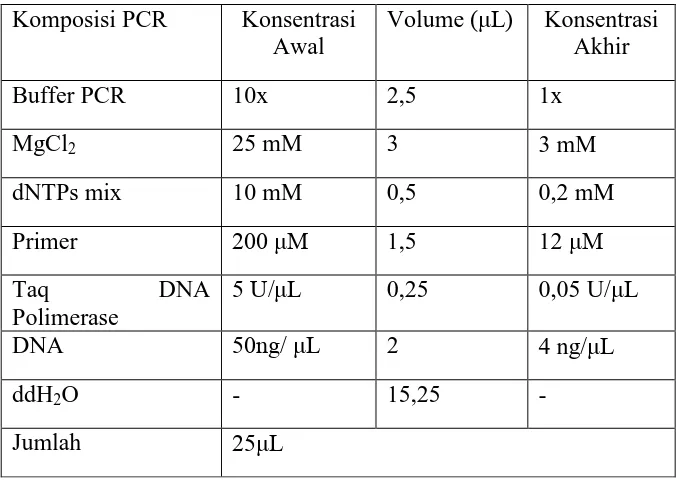
berisi 25 μLkomposisi PCR yang tercantum pada Tabel 3.1.
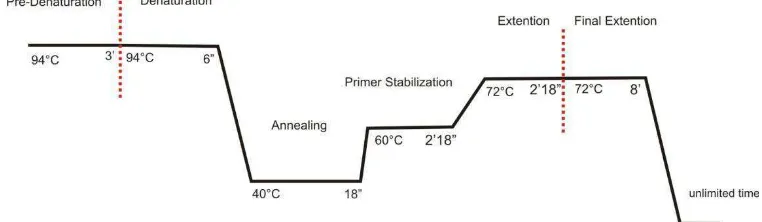
Amplifikasi DNA dilakukan dengan tahap denaturasi awal pada suhu
94°C selama 3 menit dan 60 siklus dengan suhu denaturasi 94°C selama
6 detik, annealingpada suhu 40°C selama 18 detik, stabilisasi primer
pada suhu 60°C selama 2 menit 18 detikdan ekstensi pada suhu 74°C
selama 2 menit 18 detik. Ekstensi akhir dilakukan pada suhu 74°C
selama 8 menit.
Tabel 3.1 Komposisi Reaksi PCR berdasarkan Williams, et al. (1990) yang
mengalami modifikasi
Komposisi PCR Konsentrasi Awal
Volume (μL) Konsentrasi Akhir
Buffer PCR 10x 2,5 1x
MgCl2 25 mM 3 3 mM
dNTPs mix 10 mM 0,5 0,2 mM
Primer 200 μM 1,5 12 μM
Taq DNA
Polimerase
5 U/μL 0,25 0,05 U/μL
DNA 50ng/ μL 2 4 ng/μL
ddH2O - 15,25 -
48
Gambar 3.2 Program Amplifikasi Fragmen DNA menggunakan Thermocylcer
f. Elektroforesis Hasil PCR
Amplikon yang didapat, diuji secara kualitatifdengan melakukan
elektroforesis pada gel agarose 1,4% dalam 40 ml TBE. Sebanyak 10 μL
amplikon dicampurkan dengan 3 uL loading dye dan dimasukkan ke
sumur-sumur gel. Elektroforesis dilakukan dengan tegangan 50 volt selama 2 jam.
Marker DNA yang digunakan adalah DNA Lambda yang dipotong oleh
enzim restriksi EcoRI dan HindIII.
Gel agarose yang telah melalui proses elektroforesis diwarnai dengan
Etidium Bromida (EtBr) selama 10 menit. Kemudian gel agarose yang
sudah diwarnai dengan EtBr dibilas dengan aquades selama 5 menit. Hasil
pewarnaan dilihat dengan sinar ultraviolet dari alat UV transluminator dan
didokumentasikan dengan kamera yang lensanya ditutupi kertas mika
berwarna merah.
3. Analisis Data
Analisis data molekuler dilakukan dengan melihat hasil pita-pita
DNA yang dihasilkan. Hasil pita-pita DNA menjadi dua kategori yaitu pita
dna yang monomorfik dan polimorfik. Kemudian data-data ini dibuat
dalam bentuk matriks dan buat skema interpretasinya, dengan melihat
kehadiran dan ketidakhadiran pita DNA. Jika pita DNA ada maka diberi
nilai 1 sedangkan jika pita DNA tidak ada diberi nilai 0. Selanjutnya data
dalam bentuk matriks tersebut diolah dalam software MEGA 4 dengan
program UPGMA (Unweighted Pair-Group Method with Aritmatic
49
Pita polimorfik yang dihasilkan dari amplifikasi RAPD-PCR
selanjutnya dihitung nilai PIC untuk mengetahui tingkat keinformatifan
pada primernya Nilai PIC dapat dihitung dengan rumus:
PIC = 1-∑
Keterangan:
50
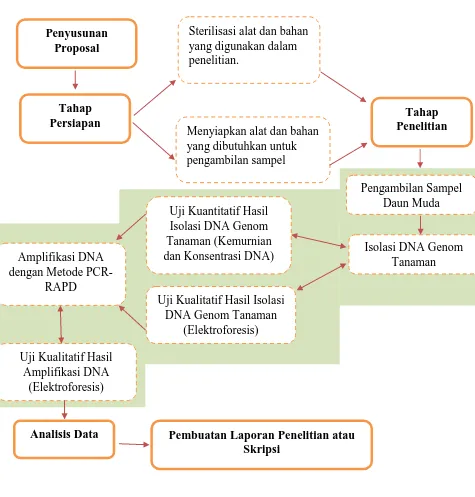
F. Alur Penelitian
Gambar 3.3 Diagram Alur Penelitian
Penyusunan
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu BAB V
KESIMPULAN DAN SARAN
A. Kesimpulan
Data fenogram yang dibentuk oleh klaster UPGMA membagi keenam
sampel yang menjadi dua kelompok besar. Kelompok I terdiri atas P.duclitan
IV.C.83, P.duclitan 112A, P.duclitan XXV/B111. P.duclitan 112 A yang
memiliki bentuk morfologi buah yang bulat terpisah dengan P.duclitan IV.C.83
dan P.duclitan XXV/B111 membentuk satu kelompok kecil di dalam kelompok 1,
diindikasikan bahwa P.duclitan 112 A termasuk ke dalam varietas atau kultivar
yang berbeda, tetapi masih berkerabat dekat dengan individu-individu P.duclitan
lainnya, sedangkan kelompok II terdiri atas P.obovata IV.170, P.duclitan
XXV.B.120 dan P.campechiana IV.D.182A.
P.duclitan XXV.B.120 memiliki larik yang lebih bervariasi dibandingkan
dengan P.duclitan lainnya, menyebabkan individu ini berada di kelompok besar
yang berbeda. P.campechiana dan P.obovata masih berkerabat dekat karena
masih termasuk ke dalam satu kelompok besar, sedangkan P.campechiana dan
P.obovata dengan P.duclitan IV.C.83, P.duclitan XXV/B111, P.duclitan 112A
berkerabat jauh hal ini dibuktikan bahwa masing-masing individu termasuk
kedalam kelompok besar yang berbeda dan karakter morfologinya berbeda.
B. Saran
Untuk penelitian selanjutnya, diperlukan analisis yang lebih mendalam
terhadap tanaman sawo Pouteria dengan melibatkan lebih banyak primer dan
lebih banyak sampel yang diuji, karena semakin banyak primer dan sampel yang
digunakan akan semakin besar peluang untuk mendeteksi polimorfisme DNA dan
Febriyantie, Vania. 2014
STUDI KEKERABATAN FENETIK BEBERAPA JENIS TANAMAN SAWO Pouteria (SAPOTACEAE) MENGGUNAKAN METODE RAPD
Universitas Pendidikan Indonesia | repository.upi.edu | perpustakaan.upi.edu DAFTAR PUSTAKA
Bakkappa, S., Talebi, E. & Subramanya, G. (2011). “Role Of Molecular Marker Conservation”. Internaional Journal of Advanced Biology Research 1(1),
hlm. 1 – 7.[Online]. Tersedia di: V., & Jackson, R. B. (2010). Biology Jilid 1 8thEdition . Jakarta: Erlangga.
Carneiro, M. S., Camargo, L. E. A., Coelho, A. S., Vencovosky, R., Junior, L. P. L., Stenzel, M. C. L. & Veiera, M. L. C. (2002). “RAPD-based genetic linkage maps of yellow passion fruit (Passiflora edulis)”. Genome 45, hlm. 670 –
678.[Online]. Tersedia di:
http://www.nrcresearchpress.com/doi/abs/10.1139/g02-035 (Diakses 15 September 2014)
Cornquist, A. (1981). An Integrated System of Classification of Flowering Plants. New York: Columbia University Press.
Das, S., Misra, R. C., Rout, G. R., Pattanaik, M. C. & Aparajita, S. (2009).
“Relationship of Status of Polymorphic RAPD Bands with GenotypicAdaptation In Early Finger Millet Genotypes”. African Crop Science Journal 17 (2), hlm. 61 – 69. [Online]. Tersedia di: http://www.bioline.org.br/pdf?cs09006(Diakses 15 September 2014)
Department of Primary Indusrty and Fisheries.(1991). A Botanical and Agronomic
Review.Australia: Northern Teritory of Australia. [Online]. Tersedia di:
www.nt.gov.au/d/Content/File/p/Tech.../TB169.pdf(Diakses 18Agustus 2014)
Ditjen Kehutanan .(1972). Daftar Nama Pohon-Pohonan Bali dan Lombok. Bogor: Lembaga Penelitian Hutan.
Fatchiya., Arumingtyas, E. L., Widyati, S., & Rahayu, S. (2011). Biologi Molekuler:
Prinsip Dasar Analisis. Jakarta : Erlangga
Fitrisari, A. (2004). Studi Keragaman Genetik Pada Osphronemus gouramy Dengan
70
(RAPD).Skripsi.Program Biologi.Universitas Pendidikan Indonesia. Bandung:
tidak diterbitkan.
Fisher, M., Husi, R., Prati, D., Peintinger, M., Kleuneun, M. V. & Schmid, B. (2000).
“RAPD Variation Among and within Small and Large Populations of The Rare
Clonal PlantRanunculus Reptans (Ranunculaceae)”. American Journal of
Botany 87 (8), hlm. 1128-1137. [Online]. Tersedia di: www.ncbi.nlm.nih.gov/ (Diakses 30 September2014)
Forest Watch Indonesia. (Tanpa Tahun). Hutan-hutan Indonesia: Apa yang
dipertaruhkan?.[Online]. Tersedia di:
http://www.wri.org/sites/default/files/pdf/indoforest_chap1_id.pdf (Diakses 15 September 2014)
Gupta, P.K., Balyan, H. S., Sharma, P. C. & Ramesh, B.(1996). “Microsatellite in
plant: A new class of molecular marker”. CURRENT SCIENCE 70(1), hlm. 45
– 54.[Online]. Tersedia di: www.currentscience.ac.in.pdf (Diakses 9 Oktober 2014)
Heyne, K. (1987). Tumbuhan Berguna Indonesia Jilid III. Jakarta: Badan Litbang Kehutanan
Hidayat, T dan Pancoro, A. (2006).“Sistematika dan Filogenetika Molekuler”.Kursus Simgkat Aplikasi Peramgkat Lunak PAUP dan MrBayes untuk Penelitian Filogenetika Molekuler SITH-ITB 2006. Bandung: tidak diterbitkan.[Online].
Tersedia di: www.pdfio.net/k-6187732.htm (Diakses 20 September 2014)
Hidayat, T. (2001).Studi Filogenetik Molekulel pada Anacardiaceae berdasarkan Variasi Urutan Daerah “Internal Transcribed Spicer” (ITS). Tesis, Sekolah Pascasarjana, Institut Teknologi Bandung.
Ian, S. (Tanpa tahun).Planchonella obovata (R. Brown) Pierre.[Online]. Tersedia di:http://www.efloras.org/florataxon.aspx?flora_id=2&taxon_id=200017543 (Diakses 12 September 2014)
International Center for Underutilised Crops. (2004). “Pouteria Fruits for TheFuture”. Factsheet 11, hlm. 1 – 2.[Online]. Tersedia di:
www.cropsforthefuture.org/publication/Factsheets/Factsheet-pouteria.pdf(Diakses 29 September 2014)
71
Karliati, T., Febriyato, F., Syafii, W.& Wahyudi, I.(2011).“Karakterisasi Getah Perca
dan Pemanfaatannya sebagai Perekat Kayu Lapis”.Jurnal Ilmu dan Teknologi Kayu Tropis 10 (1), hlm.12-22.[Online]. Tersedia di: www.mapeki.org/.../JITKT%20Vol.10%20No.1%202..(Diakses 29 September 2014)
Kekec, G., Sakcali, M. S. & Uzonur, I. (2010).“Assessment of Genotoxic Effects of
Boron on Wheat (Triticum aestivum L.)and Bean (Phaseolus vulgaris L.) by
Using RAPD Analysis”. Bull Environ Contam Toxicol 84, hlm. 759 –
764.[Online]. Tersedia di: www.ncbi.nlm.nih.gov/.../2...(Diakses 29 September 2014)
Kong, K. W., Khoo, H. E., Prasad, N. K., Chew, L. Y. & Amin, I.(2013). “Total Phenolics and Antioxidant Activities of Pouteria campechiana Fruit
Parts”.Sains Malaysiana, 42 (2), hlm. 123 – 127. [Online]. Tersedia di:
journalarticle.ukm.my/5893/(Diakses 29 September 2014)
Krisdianto.(2000). “Anatomi dan Kualitas Serat Tujuh Jenis Kayu kurang Dikenal dari Jawa Barat”.Pusat Penelitian dan Pengembangan Hasil Hutan, hlmn. 1 –
29.[Online]. Tersedia di:
www.forda-mof.org/index.php/content/.../jurnal/29(Diakses 1 Oktober 2014)
Kumar, P., Gupta, V. K., Misra, A. K., Modi, D. R., & Pandey, B. K.(2009).
“Potential of Molecular Markers in Plant Biotechnology”.Plant Omics Journal
2(4), hlm. 141 – 162.[Online]. Tersedia di:
www.pomics.com/Pradeep_2_4_2009_141_162.pdf(Diakses 10 Oktober 2014)
Kukachka, B. F. (1982). Wood Anatomy of The Neurotropical Sapotaceae.Forest
Product Laboratory Forest Service.Departement of Agriculture
Madison.[Online]. Tersedia
di:www.dtic.mil/cgi-bin/GetTRDoc?AD=ADA108863(Diakses 29 September 2014)
Kusumadewi, Y. (2005). Analisis Variasi Genetik pada Tebu (Saccahrum sp.)Asli
Indonesia Dengan Menggunakan Penanda RAPD.Skripsi., Program Biologi.
Universitas Pendidikan Indonesia.Bandung: tidak diterbitkan.
Langga, I. F.., Restu, M & Kuswinanti, T. (2012). “Optimalisasi Suhu dan Lama
Inkubasi Dalam Ekstraksi DNA Tanaman Bitti (Vitex cofassus Reinw) Serta
Analisis Keragaman Genetik dengan Teknik RAPD-PCR”. J. Sains & Teknologi 12(3), hlm. 265 – 276.[Online]. Tersedia di: pasca.unhas.ac.id/.../0b939e0181da0eccc16cd29267c(Diakses 10 September 2014)
hlm.731-72
738. [Online]. Tersedia di:www.ajebs.com/vol8/26.pdf (Diakses 29 September
2014)
Majourhat, K., Jabbar, Y., Hafidi, A. & Martinez-Gomez, P. (2008). “Molecular Characterization And Genetic Relationships Among Most Common Identified Morphotypes of Critically Endangered RareMoroccan Species Argania spinosa (Sapotaceae) Using RAPD and SSR Markers”. Ann. For. Sci. 65, hlm. 1 –
6.[Online]. Tersedia di:http://hal.inria.fr/docs/00/88/34/63/PDF/hal-00883463.pdf(Diakses 29 September 2014)
Malik, S.K., Rohini, M. R., Kumar, S., Choudary, R., Pal, D. & Chaudhury, R.(2012).
“Assessment of Genetic Diversity in Sweet Orange [Citrus sinensis (L.)Osbeck] Cultivars of India Using Morphological and RAPD Markers”.Agric Res 1(4), hlm. 317 – 324.[Online]. Tersedia di: www.sciencedomain.org/download.php?f=Al...pdf&aid=415(Diakses 18 September 2014)
Marantina, S. S. (2008). Deteksi Fragmen Gen NA Pengkode Neuraminidase Virus
Avian Influenza A Subtipe H5N1 dengan Teknik Reverse Transcription Polymerase Chain Reaction (RT-PCR). Skripsi. Departemen Biologi.
Universitas Indonesia.[Online]. Tersedia
di:lib.ui.ac.id/file?file=digital/...Deteksi%20Fragmen...(Diakses 29 Agustus 2014)
Master, J. (2012). Campoleh (Pouteria campechiana).[Online]. Tersedia di: http://staff.unila.ac.id/janter/2012/01/16/campoleh-pouteria-campechiana/ [Diakses 11 September 2014]
McPherson, Michael J. et al. (1992). Molecular Plant Pathology. New York: Oxford University Press.
Moon, H. K., Ujang, S. I., Park, S. Y., Park, C. H. & Yi, J. S.(2007). Tropical Trees of Indonesia. Korea: Korea Forest Research Institute. [Online]. Tersedia di: 152.99.88.238/KFRICAB/IMG/006/003/155916.pdf(Diakses 13 September 2014)
Moreira, P.A. & Oliveira, D.A. (2011). “Leaf age affects the quality of DNA extracted from Dimorphandra mollis (Fabaceae), a tropical tree species from
the Cerrado region of Brazil”. Genetics and Molecular Research 10 (1), hlm.
353 – 358. [Online]. Tersedia di:
www.ncbi.nlm.nih.gov/pubmed/21365551(Diakses 29 September 2014)
Mueller, U.G. &Wolfenberger, L.L. (1999).“AFLP genotyping and
73
di:https://www.ufpe.br/biolmol/Artigos_Doutorado_2003_Jan/AFLPgenotypin g-review.pdf.(Diakses 29 September 2014)
Nagy, S., Poczai, P., Cernak, I., Gorji, A. M., Hegedus, G. & Taller, J. (2012).
“PICcalc: An Online Program to Calculate Polymorphic Information
Content for Molecular Genetic Studies”. Biochem Genet 50, hlm
670-672. [Online]. Tersedia di:
www.researchgate.net/publication/224933086_PICcalc_An_Online_Program_t o_Calculate_Polymorphic_Information_Content_for_Molecular_Genetic_Stud ies/links/0fcfd50fd9a85c5635000000 (Diakses 1November 2014)
Nurtikasari, R. (2009). Analisis Keragaman Genetik Burung Famili Columbidae
Dengan Penanda Random Amplified Polymrphic DNA (RAPD).Skripsi.Program Biologi.Universitas Pendidikan Indonesia.Bandung:
tidak diterbitkan.
Opperdoes, F. (1997). Phenetics versus Cladistics and the pro's and con's of the various phylogeny inference methods. [Online]. Tersedia di: http://www.icp.ucl.ac.be/~opperd/private/phenetics.html[Diakses 11 September 2014]
Opperdoes, F. (1997b).Construction of a distance tree using clustering with the
Unweighted Pair Group Method with Arithmatic Mean (UPGMA).[Online].
Tersedia di: http://www.icp.ucl.ac.be/~opperd/private/upgma.html[Diakses 11 September 2014]
Pathak, B. C. & Serajuddin, M. (2012). “Genetic Diversity in Barred Spiny Eel
Macrognathuspancalus (Hamilton 1822) Sampled in Ganges River Basin, India”. International Journal of Genetics 2 (1), hlm. 1 – 5.[Online]. Tersedia di: idosi.org/ijg/ijg2(1)12.htm(Diakses 12 Oktober 2014)
Pennington, T.D. (2004). “Sapotaceae. InKubitzki, K. (ed.), The Families and Genera of Vascular Plants”. Springer-Verlag: Berlin. 6, 390–421.[Online]. Tersedia di:www.lrm.nt.gov.au/__data/.../SAPOTACEAE.pdf(Diakses 28 Agustus 2014)
Plowden, C. Chicheley. (1972) A Manual of Plant Names Third Edition. London: George Allen & Unwid Ltd.
74
Pradnyaniti, D.G., Wirajana, I.N. & Yowani, S.C. (2013). “Desain Primer Secara In Silico Untuk Amplifikasi Fragmen Gen RpobMycobacterium tuberculosis Dengan Polymerase Chain Reaction (PCR)”. Jurnal Farmasi Udayana hlm.
124-129.[Online]. Tersedia di:
ojs.unud.ac.id/index.php/jfu/article/download/.../5641(Diakses 10 Oktober 2014)
Raunsay, S. S. (2010). Isolasi Plasmid, Elektroforesis, Spektrofotometri. [Online]. Tersedia di: http://susanai07.student.ipb.ac.id/2010/06/18/isolasi-plasmid-elektroforesis-spektrofotometri/ (diakses 27 September 2013)
Rasnovi, S. (2004). “Konsep Spesies: Mengapa Fenetik atau
Filogenetik?”.Floribunda 2 (5), hlm.138-143. [Online]. Tersedia
di:www.ptti.or.id/floribunda2.html(Diakses 29 September 2014)
Richardson, B. J, Baverstock &Adams.(1986). Allozyme Electro-phoresis.A
Handbook for Animal Sys-tematics and Population Studies. Aca-demic Press,
Inc. San Diego : 410 pp.
Rocas, A Niembro. (Tanpa tahun).“ Pouteria campechiana (Kunth) Baehni”. Part II
Species Description.[Online]. Tersedia
di:www.rngr.net/publications/ttsm/species/PDF.2004-03-16.4509/at.../file(Diakses 29 September 2014)
Rojas, T. J., Andrade-Rodriguez, M., Alia-Tejacal, I., Lopez-Martinez, V., Espinosa-Zaragosa, S., & Esquinca-Aviles, H. (2012). “Molecular Characterization of
Zapote Mamey (Pouteriasapota (Jacq.) Moore & Stearn)”.Rec. Fac. Agron 29,
hlm.339-354.[Online]. Tersedia di:
revfacagronluz.org.ve/PDF/.../v29n3a2012339354.pd..(Diakses 16 April 2014)
Sahoo, L. (tt). Plant Biotechnology Lab Manual.Department of Biotechnology.Indian
Instiute of Technology Guwahati.[Online]. Tersedia
di:www.iitg.ernet.in/scifac/.../Plant%20Biotech%20Lab%20Manual.pdf(Diaks es 29 September 2014)
Sargent, J. R. dan George, S. G. (1975).Methods in Zone Electrophoresis BDH
Chemical LTD. Poole England: 219 pp.
Semagn, K., Bjornstard, A., & Ndjiondjop, M. N. (2006).“An Overview of Molecular Marker Methods for Plants”.African Journal of Biotechnology 5(25), hlm.
2540 – 2568.[Online]. Tersedia di:
75
Silvertown, J. W. & Charlesworth, D. (2001).Introduction to plant population
biology. UK: Blackwell Publisihing.
Sprent, J. I., Chalmers, K. J., Waugh, R., Simons, A. J. & Powell, W.
(1992).“Detection of genetic variation between andwithin populations of
Gliricidia sepium andG. maculata using RAPD markers”.Heredity 69,
hlm.465-472. [Online]. Tersedia di: www.ncbi.nlm.nih.gov/ (Diakses 2 Januari 2014)
Steven, P. (2013). Ericales.[Online]. Tersedia di: http://global.britannica.com/EBchecked/topic/191380/Ericales/276494/Sapotac eae (Diakses 2 Januari 2014)
Stuessy, T. F. (2009). “Paradigms in biological classification (1707–2007): Has anything reallychanged?”. Taxon58 (1), hlm. 68 – 76.[Online]. Tersedia di: www.jstor.org/stable/27756825(Diakses 29 September 2014)
Suaidah, L. (2010). Analisis Variasi Genetik Osphronemus gouramyLac
Menggunakan Penanda Genetik Random Amplified Polymorphic DNA.Skripsi.
Program Studi Biologi. Universitas Pendidikan Indonesia.Bandung: tidak diterbitkan.
Suyono, I. J. (2000).Analisis Struktur Genetik Populasi Nyamuk (Aedes aegypti) dari
Tiga Lokasi di Jawa Barat Menggunakan Metode “Random Amplified
Polymorphic DNA” (RAPD).Tesis. Program Pasca Sarjana. Institut Teknologi Bandung: tidak diterbitkan.
Titrawani.(1996). Biodiversiti Kodok Genus Rana Ditinjau dari Morfologi, Kariotip
dan Pola Protein di Kodya Sawahlunto.Tesis.Program Pasca Sarjana. Institut
Pertanian Bogor.[Online]. Tersedia di:
www.oseanografi.lipi.go.id/.../oseana_xxvi(1)25-31.p...(Diakses 6 September 2014)
Triono, T. (2000). “SAWO-SAWOAN: Suatu Potensi yang Terkesampingkan”.
Prosiding Seminar Hari Cinta Puspa dan Satwa Nasional.hlm. 96 –
106.[Online]. Tersedia di:
elib.pdii.lipi.go.id/katalog/index.php/.../byId/104457 (Diakses 1 Januari 2014).
Vos, P., Hogers, R., Blekeer, M., Reijans, M., Hornes, M., Frijters, A,. Pot, J. Peleman, J. & Kuiper, M. (1995). “AFLP: a new technique for DNA
76
Williams, J. G. K., Kubelik, A. R., Livak, K. J., Rafalski, J. A., & Tingey, S.V.(1990). “DNA Polymorphisms amplified by arbitrary primes are useful as
genetic markers”. Nucleic Acids Research 18(22), hlm. 6531 – 6535.[Online]. Tersedia di: www.ncbi.nlm.nih.gov/(Diakses 19 Agustus 2014)
Yunita, O. & Kresnamurti, A. (2005). “Identifikasi Simplisia yang Dijual sebagai
Strychnos ligustrina Bl. Di Pasar Tradisional Surabaya dengan Metode Random AmplifiedPolymorphic DNA (RAPD)”.Berk.Penel. Hayati 11, hlm.19
– 24.[Online]. Tersedia
di:www.berkalahayati.org/index.php/bph/article/.../404(Diakses 29 September 2014)
Zhang, L., Zhang, H. G. & Li, X. F.( 2013). “Analysis of genetic diversity in Larix
gmelinii (Pinaceae)with RAPD and ISSR markers”.Genetics and Molecular Research 12 (1), hlm.196-207.[Online]. Tersedia di:dx.doi.org/10.4238/2013.January.24.12(Diakses 29 September 2014)
Zulfahmi.(2013). “Penanda DNA untuk Analisis Genetik Tanaman”.Jurnal