i
IDENTIFIKASI GEN PENCIRI SAPI BALI POLLED
MENGGUNAKAN MIKROSATLIT
ILSTS017 DAN ILSTS089
SKRIPSI
DINDA FEBRIANTI ADAM I111 13 533
FAKULTAS PETERNAKAN UNIVERSITAS HASANUDDIN
MAKASSAR 2017
2
IDENTIFIKASI GEN PENCIRI SAPI BALI POLLED
MENGGUNAKAN MIKROSATLIT
ILSTS017 DAN ILSTS089
SKRIPSI
Oleh :
DINDA FEBRIANTI ADAM I111 13 533
Skripsi Sebagai Salah Satu Syarat Untuk Memperoleh Gelar Sarjana pada Fakultas Peternakan Universitas Hasanuddin Makassar
FAKULTAS PETERNAKAN
UNIVERSITAS HASANUDDIN
MAKASSAR
2017
3
PERNYATAAN KEASLIAN
1. Yang bertanda tangan di bawah ini :
Nama : DINDA FEBRIANTI ADAM
NIM : I111 13 533
menyatakan dengan sebenarnya bahwa : a. Karya Skripsi saya adalah asli
b. Apabila sebagian atau seluruhnya dari karya skripsi ini, terutama dalam Bab Hasil dan Pembahasan, tidak asli atau plagiasi maka bersedia dibatalkan dan dikenakan sanksi akademik yang berlaku.
2. Demikian pernyataan keaslian ini dibuat untuk dapat digunakan seperlunya.
Makassar, November 2017
DINDA FEBRIANTI ADAM
4 HALAMAN PENGESAHAN
Judul Skripsi : Idetifikasi Gen Penciri Sapi Bali Polled
Menggunakan Mikrosatelit ILSTS017 dan
ILSTS089
Nama : Dinda Febrianti Adam
Nomor Induk Mahasiswa : I111 13 533
Fakultas : Peternakan
Skripsi ini telah diperiksa dan disetujui oleh:
Prof. Dr. Ir. Lellah Rahim, M.Sc Dr. Muhammad Ihsan A. Dagong, S.Pt., M.Si
Pembimbing Utama Pembimbing Anggota
Prof. Dr. Ir. H. Sudirman Baco. M.Sc Prof. Dr. drh. Hj. Rahmawati Malaka, M.Sc Dekan Fakultas Peternakan Ketua Program Studi Peternakan
5
KATA PENGANTAR
Assalamualaikum Warahmatullahi Wabarakatuh
Alhamdulillah, segala puji syukur patut penulis haturkan kehadirat ALLAH SWT yang senantiasa melimpahkan rahmat dan hidayah-Nya, sehingga penulis dapat menyelesaikan Tugas Akhir / Skripsi ini dengan judul “Identifikasi
Gen Penciri Sapi Bali Polled Menggunakan Mikrosateit ILSTS017 dan ILSTS089” merupakan salah satu syarat untuk memperoleh gelar kesarjanaan
pada Fakultas Peternakan Universitas Hasanuddin.
Pada kesempatan ini dengan segala keikhlasan dan kerendahan hati penulis juga menyampaikan terima kasih yang sebesar-besarnya dan penghargaan yang setinggi-tingginya kepada :
1. Kedua Orang tua yang telah banyak memberi cinta, kasih sayang dan mengiringi setiap langkah penulis dengan doa restu yang tulus serta tak henti-hentinya memberikan dukungan baik secara moril maupun materil selama ini, ayahanda H. Heri Husary Adam dan Ibunda Hj. Fauziah
As’ad, beserta saudara-saudara tercinta Ahmad Fariza Adam, Charisma Alwiyah Adam, Fadia Auliyah Adam, dan Gauza Kamilia Adam, terima
kasih atas segala bantuan, motivasi dan doanya. Penulis juga mengucapkan terima kasih yang sebesar-besarnya untuk seluruh keluarga yang selalu mendoakan dan dukungan yang tak henti-hentinya.
2. Bapak Prof. Dr. Ir. Lellah Rahim, M.Sc selaku pembimbing utama dan bapak Dr. Muhammad Ihsan Andi Dagong, S.Pt., MS selaku pembimbing
6
anggota yang telah memberikan nasehat, arahan, petunjuk dan bimbingan serta dengan sabar dan penuh tanggung jawab meluangkan waktunya mulai dari penyusunan hingga selesainya skripsi ini.
3. Bapak Prof. Dr. Ir. Syamsuddin Garantjang, M.Agr, Prof. Dr. Ir. H.
Sudirman Baco, M.Sc, Prof. Dr. Muhammad Yusuf, S.Pt, dan Ibu Prof. Rr. Sri Rachma A. Bugiwati, M.Sc, Ph.D selaku dosen pembahas mulai
dari seminar proposal hingga seminar hasil penelitian, terima kasih telah berkenan mengarahkan dan memberi saran dalam menyelesaikan skripsi ini. 4. Bapak Prof. Dr. Ir. Ahmad Ramadhan Siregar, MS selaku penasehat
akdemik yang terus memberikan arahan, nasihat dan motivasi selama ini. 5. Prof. Dr. Dwia Aries Tina Palubuhu, M.A, selaku Rektor Universitas
Hasanuddin.
6. Bapak Prof. Dr. Ir. H. Sudirman Baco, M.Sc selaku Dekan Fakultas Peternakan, Bapak Wakil Dekan I, II, III dan seluruh Staf Pegawai Fakultas Peternakan, terima kasih atas segala bantuan kepada penulis selama menjadi mahasiswa.
7. Bapak Prof. Dr. Muhammad Yusuf, S.Pt selaku Ketua Jurusan Produksi Ternak, bapak Dr. Muhammad Ihsan A. Dagong, S.Pt., MS sebagai Sekertaris Jurusan terima kasih yang sebesar-besarnya atas bimbingan, dukungan dan bantuannya kepada Penulis.
8. Semua Dosen-Dosen Fakultas Peternakan Universitas Hasanuddin yang telah memberi ilmunya kepada penulis.
7
9. Seluruh Staf dalam lingkungan Fakultas Peternakan Universitas Hasanuddi nyang selama ini telah banyak membantu dan melayani penulis selama menjalani kuliah hingga selesai.
10. Terima kasih kepada Sahabat HIMABEBS, Chairunnisa Idrus Assegaf,
Radinda Dwi Choirunnisa, Andi Jeniwari Elvina, Majdah Pratiwi, Indra Adighuna, Widi Mashoeri, Nawawi Arfan, Fulki Alen, Dwi Suprapto, Wahyu, dan Gabriel mulai dari Maba selalu bersama, terima kasih atas
kebersamaan, bantuannya dan telah menjadi bagian dari penulis serta menemani hari-hari penulis selama ini sampai mendapatkan Gelar S.Pt 11. Keluarga besar Larfa 13, kalian keluarga yang tak akan pernah penulis
lupakan, terima kasih untuk semua kenangan indah yang mengantarkan penulis meraih gelar sarjana.
12. Sahabat AW Room, Nyii, Suke, Racil, Uli, Naya, Ilha, Meno, Obhel,
dian, Ichong dan Icha, terima kasih yang setinggi-tingginya serta
penghargaan yang sebesar-besarnya atas segala cinta, pengorbanan, bantuan, pengertian, candatawa serta kebersamaan selama ini, waktu yang dilalui sungguh merupakan pengalaman hidup yang berharga dan tak mungkin untuk terlupakan dan terima kasih telah memberiku sedikit tempat di hatimu untuk menjadikanku sahabat dan teriring dengan doa semoga rekan dan sahabatku sukses selalu.
13. Kepada Tim Penelitian Genetika Molekuler, Radinda Dwi, Erwin Jufri,
Hasman, Arman, dan Husni. Terima kasih atas segala kebaikan serta
8
14. Kepada angkatan Merpati 09, L10N, Solandeven 11, FM 12, dan ANT 14. 15. Kakanda Abdullah Magfirah Irfan, Muh. Haekal Pratama, Ibnu Hajar,
Agung Perdana dan Candra Arsandi, terima kasih atas segala perhatian,
bantuan, dukungan dan doanya.
16. Semua pihak yang tidak dapat penulis sebut satu persatu, terima kasih atas bantuannya.
Penulis menyadari bahwa penyusunan skripsi ini masih terdapat kekurangan dan kesalahan. Penulis mengharapkan kritikan dan saran yang sifatnya membangun demi kesempurnaan skripsi ini. Semoga Allah SWT membalas segala kebaikan semua yang penulis telah sebutkan diatas maupun yang belum sempat ditulis. Akhir kata penulis ucapkan banyak terima kasih dan menitip harapan semoga tugas akhir ini bermanfaat bagi kita semua.
Amin yaa robbal a’lamin
Wassalamualaikum Wr.Wb
Makassar, November 2017 Penulis
9 ABSTRAK
DINDA FEBRIANTI ADAM (I111 13 533). Identifikasi Gen Penciri Sapi Bali Polled Menggunakan Mikrosatelit ILSTS017 dan ILSTS089. Dibawah bimbingan Lellah Rahim sebagai Pembimbing Utama dan Muhammad Ihsan A. Dagong
sebagai Pembimbing Anggota.
Penelitian ini bertujuan untuk mengetahui gen penciri sifat tanpa tanduk (Polled) pada sapi Bali dengan menggunakan mikrosatelit lokus ILSTS017 dan ILSTS089. Sampel yang digunakan terdiri dari 11 sampel darah sapi Bali Polled dan 89 sampel darah sapi Bali bertanduk. Variasi genetik diidentifikasi menggunakan DNA mikrosatelit. Hasil Penelitian menunjukkan bahwa lokus ILSTS017 dan ILSTS089 mengahasilkan pita/alel yang beragam (polimorfik). Pada lokus ILSTS017 terdapat 4 jenis alel yaitu D, I, G ,J dan lokus ILSTS089 terdapat 3 jenis alel yaitu A, B, D. Hasil penelitian ini menyimpulkan bahwa alel yang ditemukan tidak dapat digunakan sebagai gen penciri sifat Polled pada sapi Bali
10 ABSTRACT
DINDA FEBRIANTI ADAM (I111 13 533). Identification of The Gene
Identifier of Bali Polled Cattle Using ILSTS017 and ILSTS089 Microsatellite. Under the guidance of Lellah Rahim as the Main Supervisor and Muhammad
Ihsan A. Dagong as the co main Supervisor.
The aim of this study to determine the nature of the gene identifier of polled trait in Bali cattle used ILSTS017 and ILSTS089 microsatellite locus. In total, 100 heads of samples consist of 11 heads of polled Bali and 89 heads horned Bali. Genetic variation was identified by using microsatellite DNA. The results showed that the ILSTS017 and ILSTS089 loci gave various bands / alleles (polymorphs). At ILSTS017 locus there are 4 types of alleles, namely D, I, G, J and ILSTS089 locus alleles there are 3 types namely A, B, D. The Results of this research conclude that alleles found can not be used as a genetic identifier of polled Bali cattle.
x
DAFTAR ISI
Halaman
DAFTAR ISI ... x
DAFTAR TABEL ... xi
DAFTAR GAMBAR ... xii
DAFTAR LAMPIRAN ... xiii
PENDAHULUAN... 1
TINJAUAN PUSTAKA Gambaran Umum Sapi Bali ... 3
Potensi Kekhasan pada Sapi Bali ... 4
Karakteristik Sifat Polled pada Sapi ... 5
Keunggulan Sifat Polled dari Segi Manajemen Pemeliharaan ... 6
Identifikasi Potensi Genetik Sapi Bali Polled ... 6
Identifikasi Gen Pengontrol Sifat Polled ... 9
Keragaman Genetik... ... 9
METODE PENELITIAN Waktu dan Tempat ... 11
Alat dan Bahan ... 11
Tahapan Penelitian... 12
Analisis Data ... 15
HASIL DAN PEMBAHASAN Amplifikasi DNA Mikrosatelit ... 17
Frekuensi Genotipe dan Alel ... 19
Nilai Heterozigositas ... 20
KESIMPULAN DAN SARAN……… 22
DAFTAR PUSTAKA ... 23
LAMPIRAN ... 28 RIWAYAT HIDUP
xi
DAFTAR TABEL
No. Halaman
1. Urutan Basa dan Ukuran Mikrosatelit ... 13
2. Frekuensi Genotipe dan Alel Lokus ILSTS017 ... 19
3. Frekuensi Genotipe dan Alel Lokus ILSTS089 ... 20
4. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas ... 21
Harapan (He) Lokus ILSTS017 ... 5. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas Harapan (He) Lokus ILSTS089 ... 21
1
PENDAHULUAN
Indonesia dikenal sebagai negara kaya akan keanekaragaman hayati dan salah satunya adalah kekayaan berbagai macam ternak, termasuk sapi. Salah satu bangsa sapi lokal Indonesia adalah sapi Bali yang memiliki populasi cukup besar. Hal ini menunjukkan posisi sapi Bali dalam pemenuhan kebutuhan daging nasional sangat strategis sehingga upaya peningkatan populasi dan peningkatan mutu genetik tetap harus diupayakan.
Sapi Polled adalah ternak sapi yang tanduknya tidak tumbuh secara alami. Sapi Bali tanpa tanduk (Polled) ini memiliki keunggulan dari aspek manajemen pemeliharaan. Salah satu aspek manajemen yakni kemudahan dalam pemeliharaan yang memiliki dampak terhadap produktivitas dagingnya. seperti mengurangi resiko terluka yang sering terjadi pada ternak yang disebabkan oleh tanduk, dapat mencegah memar pada karkas dan kerusakan pada kulit. Seleksi terhadap sapi
Polled menjadi sangat penting terutama pada manajemen budidaya ternak yang
modern (Brockmann et al., 2000)
Kemajuan teknologi genetika molekuler memberikan peluang untuk melakukan seleksi berdasarkan gen yang mengontrol sifat-sifat tertentu. Perkembangan sejumlah penanda molekuler memungkinkan untuk melakukan identifikasi terhadap perubahan-perubahan genetik yang terjadi dalam suatu persilangan serta hubungan dengan sifat kuantitatif dan kualitatif ternak. Selain itu, penanda molekuler juga dapat digunakan untuk membedakan antara suatu ras ternak dengan yang lain terutama kaitannya dalam upaya pelestarian dan menjaga
2
kemurnian dari ras tersebut. Salah satu penanda molekuler yang sangat populer saat ini adalah mikrosatelit.
Mikrosatelit merupakan alat bantu yang sangat akurat untuk membedakan genotipe, evaluasi kemurnian benih, pemetaan, dan seleksi genotipe untuk karakter yang diinginkan. Mikrosatelit tergolong sebagai penanda molekuler yang sangat efektif, yakni sekuen DNA yang bermotif pendek dan diulang secara tandem dengan dengan 2 sampai 5 unit basa nukleotida (dikenal sebagai motif) yang tersebar dan meliputi seluruh genom (Lumban dkk., 2013). Analisis mikrosatelit merupakan salah satu penciri genetik yang sudah diaplikasikan secara meluas dalam bidang peternakan. Identifikasi penciri genetik sebagai alat penanda pembeda bangsa merupakan langkah penting sebagai ciri khusus yang bersifat khas pada bangsa ternak tertentu.
Berdasarkan uraian tersebut maka dilakukan suatu penelitian mengenai identifikasi gen penciri sapi Bali Polled menggunakan mikrosatelit ILSTS017 dan ILSTS089.
Tujuan dari penelitian ini adalah untuk mengetahui gen penciri sifat tanpa tanduk (Polled) pada sapi Bali dengan menggunakan mikrosatelit lokus ILSTS017 dan ILSTS089. Kegunaan dari penelitian ini adalah untuk memberikan informasi kepada peneliti terkait gen penciri sapi Bali Polled yang dapat digunakan untuk menentukan keunggulannya, juga kepada peternak dan pemegang kebijakan dalam pengembangan sapi potong lokal khususnya pada sapi tanpa tanduk (Polled).
3
TINJAUAN PUSTAKA
Gambaran Umum Sapi Bali
Sapi Bali merupakan ternak asli Indonesia. Keaslian sapi Bali telah dibuktikan oleh Mohamad et al. (2009) menggunakan pendekatan genetika molekuler berupa analisa DNA mitokondria yang mempunyai potensi genetik dan nilai ekonomis yang cukup potensial untuk dikembangkan sebagai ternak potong. Sapi Bali adalah domestikasi dari banteng (Bibos banteng Syn. Bos sondaicus) yang telah terjadi sejak zaman prasejarah (Purwantara et al., 2012). Namun ada juga yang menduga bahwa sapi Bali adalah asli berasal dari pulau Bali yang dalam perkembangan selanjutnya dapat dipertahankan kemurniannya (Gunawan et
al., 2004).
Proses domestikasi sapi Bali terjadi sebelum 3.500 SM di Indonesia. Banteng liar saat ini bisa ditemukan di Jawa bagian Barat dan bagian Timur, di Pulau Kalimantan, serta ditemukan juga di Malaysia (Payne and Rollinson, 1973). Ciri khas sapi Bali (Bos sondaicus) adalah warna bulunya merah bata dan mempunyai garis belut di sepanjang punggungnya (Guntoro, 2002). Sapi Bali juga mudah beradaptasi di lingkungan yang buruk dan tidak selektif terhadap makanan. Selain itu, sapi Bali cepat beranak, jinak, mudah dikendalikan dan memiliki daya cerna terhadap makanan serat yang baik (Batan, 2006).
Perkembangan sapi Bali di Indonesia sangat cepat dibanding dengan bangsa sapi potong lainnya, hal tersebut menyebabkan bangsa sapi ini lebih diminati oleh petani kecil karena beberapa keunggulan yang dimiliki antara lain, tingkat fertilitas dan reproduksi tinggi, yang ditandai dengan angka konsepsi dapat
4
mencapai 85,9% dan persentase beranak sekitar 70-81% (Murtidjo, 1990 ; Handiwirawan dan Subandriyo, 2004).
Potensi Kekhasan Sapi Bali
Sapi Bali merupakan ternak tipe potong atau pedaging dan sebagai ternak pekerja. Sapi Bali merupakan penghasil daging utama untuk ruminansia besar di Indonesia. Sapi Bali memiliki bentuk tubuh yang relatif lebih kecil jika dibandingkan bangsa sapi potong yang ada (Bos indicus dan Bos taurus). Bentuk tubuh yang kecil diduga disebabkan oleh faktor genetik dan manajemen pemeliharaan yang diberikan oleh peternak (Masudana, 1990).
Sapi Bali memiliki karakteristik fenotipe yang unik dibandingkan dengan sapi lainnya. Menurut Pane (1986) anak sapi jantan hingga sekitar umur 6 bulan berwarna sama dengan sapi betina yaitu merah bata kecoklatan, tetapi dengan semakin tua umurnya akan mulai berubah menjadi coklat kehitaman mulai dari bagian depan tubuh ke belakang, terdapat warna putih pada bagian belakang paha (pantat), bagian bawah (perut), keempat kaki bawah (white stocking) sampai di atas kuku, bagian dalam telinga, dan pinggiran bibir atas pada sapi Bali jantan dan betina (Hardjosubroto dan Astuti, 1993).
Kemampuan sapi Bali beradaptasi pada lingkungan menjadi hal yang penting, karena kemampuan tersebut tidak dimiliki oleh beberapa bangsa sapi lainnya. Sapi Bali dapat memanfaatkan pakan dengan kualitas rendah (Sastradipraja, 1990), mempunyai fertilitas dan conception rate yang sangat baik (Oka dan Dramadja, 1996), dan memiliki daging berkualitas baik dengan kadar lemak rendah (Bugiwati, 2007).
5
Keunggulan yang dimiliki oleh sapi Bali dari segi pertumbuhan yakni, bobot lahir yang dapat mencapai 18,37 Kg untuk jantan dan 18,27 Kg untuk betina. Berat sapih pedet jantan 93,53±21,00 kg dan pedet betina 87,66±12,04 kg, dengan waktu sapih 205 hari. Keunggulan lainnya yakni laju pertumbuhan bobot badan yang cukup tinggi dengan persentase karkas sebesar 51,22%. Berdasarkan keunggulan dari segi pertumbuhan, sapi Bali menjadi pilihan sebagai ternak potong penghasil daging di daerah tropis khususnya di Indonesia (Ismail dan Nuraini, 2014).
Karakteristik Sifat Polled pada Sapi
Tanduk memiliki fungsi sebagai alat perlindungan sapi dari predator dan pada persaingan dalam mencari pakan, terutama pada kehidupan liar. Sebagian pihak memiliki hipotesis bahwa fungsi lain tanduk berhubungan dengan efektivitas reproduksi, dimana ternak betina cenderung memilih sapi yang bertanduk. Ternak sapi yang tanduknya tidak tumbuh secara alami diistilahkan sebagai sapi Polled. Polled merupakan sebuah sifat yang diturunkan melalui pola autosomal dominan (Cargill et al., 2008).
Fenomena tidak tumbuhnya tanduk pada sapi dikategorikan dalam dua kondisi, 1) dikatakan Polled jika tanduk tidak tumbuh secara alami dan 2) kondisi
scurs yakni tidak tumbuhnya tanduk yang disebabkan oleh kegagalan
penggabungan antara inti tulang tanduk dengan tengkorak. Kondisi scurs dapat juga dikatakan sebagai pertengahan antara kondisi sapi bertanduk dengan tidak bertanduk, disebabkan sapi yang bersifat scurs tetap memiliki tanduk namun tidak
6
tumbuh secara sempurna. Hal tersebut menjadi penting untuk membedakan ternak sapi yang bersifat Polled dengan sifat scurs.
Keunggulan Sifat Polled pada Sapi
Keunggulan dari sifat Polled, yakni generasi homozigot pada sapi Polled mengurangi biaya dan waktu untuk pemotongan tanduk dan menghilangkan stres pada ternak. Beberapa negara telah memberlakukan animal walfare terkait
dehorning, sehingga pemuliabiakan terhadap sapi Polled menjadi lebih
menguntungkan. Pada sapi Simmental, telah banyak upaya yang dilakukan untuk menghasilkan bangsa murni Simmental Polled melalui seleksi fenotifik tradisional. Upaya tersebut telah menghabiskan waktu selama 25 tahun (Brockmann et al., 2000).
Beberapa keunggulan lain pada sapi Polled dari segi manajemen pemeliharaan, seperti mengurangi resiko terluka yang sering terjadi pada peternak yang disebabkan oleh tanduk, dapat mencegah memar pada karkas dan kerusakan pada kulit. Seleksi terhadap sapi Polled menjadi sangat penting terutama pada manajemen budidaya ternak yang modern (Brockmann et al., 2000). Dampak paling jelas dari keberadaan tanduk, terlihat pada saat pengangkutan menuju rumah potong hewan, yaitu ditemukan banyak memar pada ternak yang bertanduk, disebabkan oleh adanya persaingan dan persinggungan antar ternak yang terjadi di atas alat angkut (mobil pengangkut sapi).
Identifikasi Potensi Genetik Sapi Bali Polled
Sapi Bali merupakan hasil domestikasi langsung dari Banteng liar (Namikawa et al., 1980; Payne dan Hodges, 1997; Martojo, 2003), pendapat
7
tersebut diperkuat oleh ciri khas (fenotipe) sapi Bali yang sangat mirip dengan Banteng. Sapi Bali yang dikenal saat ini diistilahkan sebagai Bos javanicus (Zulkharnaim et al., 2010). Fenomena sifat Polled pada sapi Bali seharusnya memiliki dasar ilmiah yang menerangkan keabsahan jenis bangsanya. Hasil pengkajian awal menunjukkan bahwa sapi Bali Polled masih sebangsa dengan sapi Bali pada umumnya, sehingga dibutuhkan pengkajian secara molekuler terhadap potensi genetik yang terekspresi pada sifat fenotipe dari sapi Bali Polled. Kemurnian genetik merupakan suatu individu dimana individu tersebut memiliki alel untuk gen yang menduduki lokus pada suatu kromosom yang sama dengan tetuanya. Analisa kemurnian sapi Bali telah dilakukan dengan menggunakan beberapa penanda molekuler (DNA marker), salah satu penanda molekuler yang sangat populer dewasa ini adalah mikrosatelit (Maskur, 2007). Pada saat sekarang ini mikrosatelit adalah pilihan terbaik dan penting untuk karakterisasi rumpun sapi atau populasi (Metta et al., 2004; Rincon et al., 2007). Marka genetik tersebut juga sering digunakan untuk menjawab pertanyaan terkait dengan keragaman genetik dan hubungan genetik diantara populasi sapi (Rincon
et al., 2007; Sun et al., 2008; Chaudhari et al., 2009).
Lokus mikrosatelit disukai karena polimorfisme yang tinggi (banyak alel dalam populasi), kodominan (segregasi homozigot dan heterozigot), relatif melimpah dalam genom (Rincon et al., 2007; Karthickeyan et al., 2008). Mikrosatelit disebut juga sebagai sequence repeats (SSR's), short tandem repeats (STR's), simple sequence tandem repeats (SSTR), variable number tandem
8 repeats (SSR), dan sequence tagged microsatellites (STMS) (Teneva, 2009).
Mikrosatelit merupakan runtutan DNA pendek berulang dengan panjang antara 1-5 bp serta memiliki panjang total sekitar 10-100 bp. Runutan DNA yang berulang meliputi DNA satelit, DNA mini satelit dan DNA mikrosatelit yang dalam genom memiliki jumlah total 15 %. DNA mikrosatelit ditemukan pada prokariot dan eukariot termasuk pada mamalia (Bennet, 2000). Motif yang berulang pada mikrosatelit biasanya adalah di-nucleotide, misalnya ATATAT (Karthickeyan et
al., 2008). Informasi mengenai keragaman DNA mikrosatelit pada sapi-sapi lokal
Indonesia telah dilaporkan sebelumnya (Satriani dkk., 2002; Sarbaini 2004; Winaya dkk., 2007; Abdullah, 2008).
Mikrosatelit cenderung terpusat pada daerah inisiasi transkripsi, dan tidak ditemukan pada posisi intergene dan dalam pseudogenes (Maskur, 2007). Mikrosatelit yang kaya akan basa purin dan pirimidin seperti (CA) n, dapat membentuk Z-Dna di bawah kondisi-kondisi fisiologis (Comings, 1998). Berdasarkan dari fakta-fakta tersebut, didapatkan informasi yang menunjukkan peran yang potensial dari mikrosatelit dalam regulasi gen. Beberapa penelitian menunjukkan adanya asosiasi yang signifikan antara lokus mikrosatelit dengan sifat kuantitatif seperti sifat produksi susu (Kantanen et al., 2000), lemak karkas (Fitzsimmons et al., 1998), perbedaan tingkat fertilitas (Oliveira et al., 2002) dan efisiensi reproduksi (João et al., 2005) pada beberapa bangsa sapi.
Microsatellite typing juga dapat dilakukan dengan cara yang sederhana
dan dapat diinterpretasikan dengan mudah dengan menggunakan teknologi PCR (Polymerase Chain Reaction). PCR atau reaksi berantai polimerase adalah teknik
9
amplifikasi in vitro fragmen gen tertentu yang terletak di antara pasangan oligonukleotida primer spesifik. PCR mampu mempermudah teknologi yang ada dan mempercepat perkembangan teknik baru yang tidak mungkin dilakukan sebelumnnya. Teknik ini secara umum dapat mengurangi sejumlah tahap analisis sehingga memenuhi karakter ekonomisnya (Newton and Graham, 1994).
Identifikasi Gen Pengontrol Sifat Polled
Sifat Polled pada sapi merupakan sebuah sifat autosomal dominan. Lauwerier (2015) berpendapat sifat Polled terjadi disebabkan oleh terjadinya mutasi yang ditentukan oleh sebuah gen tunggal (gen Polled). Lebih lanjut, sifat
Polled dikodekan dengan alel Polled (P), bertanduk (p). Sifat Polled bersifat
dominan terhadap sifat bertanduk. Sapi-sapi tanpa tanduk selalu dalam bentuk homozigot dominan (PP) atau heterozigot (Pp). Sedangkan pada sifat bertanduk hanya akan muncul jika dalam bentuk homozigot resesif (pp). Sehingga untuk menghasilkan sapi Polled, hanya membutuhkan satu pejantan atau induk untuk menghasilkan keturunan Polled. Penelitian mengenai lokus spesifik yang mengontrol kejadian Polled pada beberapa bangsa sapi telah banyak dilakukan. Sebagian besar penelitian tersebut menyimpulkan bahwa kejadian Polled disebabkan oleh terjadinya mutasi yang merubah asam basa DNA sehingga terjadi perubahan asam amino yang terbentuk pada saat proses transkripsi DNA.
Keragaman Genetik
Keragaman genetik adalah variasi karakteristik yang ada diwariskan pada populasi spesies yang sama. Hal tersebut berperan penting dalam evolusi yang memungkinkan spesies beradaptasi dengan lingkungan baru dan untuk
10
menghindari parasit. Hal ini berlaku untuk terak, yang biasanya memiliki tingkat keragaman rendah (Dianti, 2014).
Hendrick (2000) menyatakan bahwa keragaman genetik adalah perbedaan antara individu dalam suatu populasi, antara individu dalam populasi yang berbeda dalam spesies yang sama atau dalam spesies yang berbeda. Keragaman genetik dalam suatu populasi banyak dipengaruhi oleh faktor-faktor seperti seleksi, inbreeding, mutasi dan migrasi. Mutasi dianggap selalu meningkatkan jumlah keragaman genetik, sedangkan migrasi dan inbreeding selalu mengurangi keragaman genetik. Faktor seleksi bisa meningkatkan atau menurunkan keragaman genetik suatu ternak dalam suatu populasi. Bloot et al. (1998) menyatakan bahwa keragaman genetik sangat penting baik pada studi populasi
maupun evolusi. Mempertahankan keanekaragaman genetik berarti
mempertahankan bangsa-bangsa dalam populasi yang mempunyai sifat khusus dan apabila semua hewan seragam maka keamanan, ketersediaan pangan dan hayati akan terancam punah. Nei (1987) menyatakan bahwa keragaman genetik populasi secara kuantitatif dapat menggunakan lokus polimorfik dan rataan heterozigositas. Nei dan Kumar (2000) menyatakan bahwa derajat heterozigositas merupakan rataan persentase lokus heterozigot tiap individu atau rataan persentase individu heterozigot di dalam populasi.
11
METODE PENELITIAN
Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan Februari sampai Agustus 2017. Penelitian ini dilaksanakan di Laboratorium Ternak Potong, Ranch Maiwa Breeding Center dan Laboratorium Terpadu Fakultas Peternakan Universitas Hasanuddin.
Bahan dan Alat
Penelitian ini menggunakan 100 ekor sapi Bali yang terdiri dari 89 sampel darah sapi Bali bertanduk dan 11 sapi Bali Polled dari Laboratorium Ternak Potong dan Ranch Maiwa Breeding Center Fakultas Peternakan Universitas Hasanuddin.
Bahan pendukung antara lain: Primer, bahan ekstraksi DNA (Kit DNA ekstraksi (Thermo Scientific), Proteinase K, ethanol 96% ), bahan PCR (dNTP mix, Enzim Taq DNA polymerase, 10x buffer, 10x TBE buffer), bahan elektoforesis (agarose, Ethidium bromide, Marker DNA 100pb, Loading dye), tissue dan plastik mika.
Alat yang digunakan yaitu : venoject, tabung vakuttainer, mesin PCR (Sensor Quest Germany), centrifuge, alat pendingin, tabung eppendorf besar kecil, gel documention, mikropipet, tip, rak tabung, elektroforesis, autoclave, timbangan, sarung tangan.
12
Tahapan Penelitian Koleksi sampel darah
Sampel darah diperoleh dari Laboratorim Ternak Potong dan Ranch Maiwa Breeding Center Fakultas peternakan. Pengambilan darah melalui vena
jugularis ditampung pada tabung vakuttainer yang telah berisi antikoagulan
EDTA untuk mencegah penggumpalan darah.
Ekstraksi DNA
Ekstraksi DNA dilakukan di Laboratorium Terpadu Fakultas Peternakan. DNA diisolasi dan dimurnikan dengan menggunakan Kit DNA ekstraksi Genjet Genomic DNA Extraction (Thermo Scientific) dengan mengikuti protocol ekstraksi yang disediakan. Sebanyak 200 µl sampel darah dilisiskan dengan menambah 400 µl larutan lysis buffer dan 20 µl proitenase K (10 mg/ml), dicampurkan kemudian diinkubasi pada suhu 56 ºC selama 60 menit di dalam
waterbath shaker. Setelah inkubasi larutan kemudian ditambahkan 200 µl Ethanolabsolute 96% dan disentrifugasi 6.000 x g selama 1 menit.
Pemurnian DNA kemudian dilakukan dengan metode spin column dengan penambahan 500 µl larutan pencuci wash buffer I yang kemudian dilanjutkan dengan sentrifugasi pada 8.000 x g selama 1 menit. Setelah supernatannya dibuang, DNA kemudian dicuci lagi dengan 500 µl wash buffer II dan disentrifugasi pada 12.000 x g selama 3 menit. Setelah supernatannya dibuang, DNA kemudian dilarutkan dalam 200 µl elution buffer dan disentrifugasi pada 8.000 x g untuk selanjutnya DNA hasil ekstraksi ditampung dan disimpan pada suhu -20 ºC.
13
Analisis PCR dengan Penciri DNA Mikrosatelit
Komposisi reaksi PCR dikondisikan pada volume reaksi 25 μl yang terdiri atas 2 μl DNA, 0,3 μl masing – masing primer, 1 μl dNTP, 1 μl Mg2+, 0,1 μl Taq DNA polymerase, 1x buffer dan 15,1 μl H2O. Kondisi mesin PCR dimulai dengan
denaturasi awal pada suhu 94oC selama 2 menit, diikuti dengan 35 siklus berikutnya masing – masing denaturasi 94oC selama 45 detik, dengan suhu annealing lokus DNA mikroatelit lokus ILSTS017 dan ILSTS089 yaitu 60°C selama 30 detik yang dilanjutkan dengan ekstensi : 72°C selama 60 detik, yang kemudian diakhiri dengan satu siklus ekstensi akhir pada suhu 72°C selama 5 menit dengan menggunakan mesin PCR (SensoQuest, Germany).
Analisis produk PCR dan deteksi terhadap alel mikrosatelit dilakukan dengan elektroforesis pada gel poliakrilamida dan pewarnaan dengan perak mengikuti metode Tegelstrom (1992).
Primer Mikrosatelit
Penelitian ini menggunakan Lokus ILSTS017 dan ILSTS089 sebagai penciri mikrosatelit pada penanda DNA. Urutan basa mikrosatelit untuk penanda tersebut disajikan pada Tabel 1.
Tabel 1. Urutan Basa dan Ukuran Mikrosatelit
Lokus Urutan Basa Primer
Suhu Anneling (°C) Ukuran (bp) ILSTS017 F: 5’-GTCCCTAAAATCGAAATGCC-3’ R: 5’-GCATCTCTATAACCTGTTCC-3’ 60 105-125 ILSTS089 F: 5’-AATTCCGTGGACTGAGGAGC-3’ R: 5’-AAGGAACTTTCAACCTGAGG-3’ 60 128
Sumber : Kemp et al., 1995 F = Forward, R = Reverse
14
- Elektroforesis pada Gel Poliakrilamid
Komponen gel poliakrilamida terdiri atas campuran 30% acrylamida dan bis-akrilamida sebanyak 6 ml, 10 x TBE sebanyak 6 ml, H2O sampai mencapai
volume 30 ml, temed sbanyak 20 μl, 10% APS 200 μl. Sampel DNA tersebut kemudian dimasukkan ke dalam sumur gel setelah gel diletakkan pada tangki elektroforesis yang telah berisi larutan penyangga 1 x TBE. Elektroforesis dilakukan pada voltase konstan 250 V selama 120 menit pada suhu ruang.
- Silver stainning (Pewarnaan Perak)
Pewarnaan dengan perak dilakukan melalui serangkaian proses yaitu pewaraan gel dengan larutan stainning dengan merendam gel dalam larutan yang terdiri atas 0,2 g AgNO3 ; 80 μl NaOH 10 N ; 0,8 ml NH4OH ; 200 ml akuades
selama selama 15 menit. Gel kemudian dicuci kembali dengan aquades selama 20 menit sambil digoyang untuk menghilangkan perak yang tidak berikatan dengan DNA. Fragmen DNA yang berikatan dengan perak dapat dideteksi dengan merendam gel dalam larutan NaOH 0,03 g/ml dan formalin yang dipanaskan pada suhu 45 oC sampai fragmen pita DNA tampak. Setelah fragmen DNA tampak, reaksi kemudian dihentikan dengan menggunakan asam asetat glasial (200 μl / 200 ml aquades).
- Penentuan posisi pita DNA
Penentuan posisi pita DNA pada gel poliakrilamida dilakukan secara manual. Pita DNA yang muncul pada gel poliakrilamida diasumsikan sebagai alel mikrosatelit. Ukuran dan jumlah dari alel yang muncul pada gel ditentukan berdasarkan asumsi bahwa semua pita DNA dengan laju migrasi yang sama
15
adalah homolog (Leung et al., 1993), sedangkan alel dengan migrasi paling cepat ditetapkan sebagai alel A, berikutnya adalah alel B dan seterusnya.
Analisis Data
1. Frekuensi Alel
Frekuensi gen penentu sifat Polled diperoleh dari analisis penciri PCR-RLFP dihitung menggunakan rumus (Nei, 1987) :
Keterangan :
xi = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii (homozigot)
nij = jumlah individu bergenotipe ij (heterozigot)
N = jumlah total sampel
2. Nilai Heterozigositas Pengamatan
Keragaman genetik (genetic variability) dilakukan melalui estimasi nilai heterozigositas pengamatan (Ho), heterozigositas harapan (He) dan standar eror
heterozigositas harapan (Weir, 1996) :
Keterangan :
Ho = nilai heterozigositas pengamatan
N1ij = jumlah individu heterozigositas pada lokus ke-1
16
3. Nilai Heterozigositas Harapan
Keterangan :
Ho = Nilai heterozigositas harapan P1i = frekuensi alel ke-I pada lokus 1 n = jumlah alel pada lokus ke-1
17
HASIL DAN PEMBAHASAN
Amplifikasi DNA Mikrosatelit
Hasil amplifikasi PCR terhadap lokus ILSTS017 dan ILSTS089 pada 100 sampel DNA genom sapi Bali yang terdiri dari 11 sampel darah sapi Bali Polled
dan 89 sampel darah sapi Bali bertanduk menghasilkan pita/alel
sebagaimanaditampilkan pada Gambar 1 dan Gambar 2.
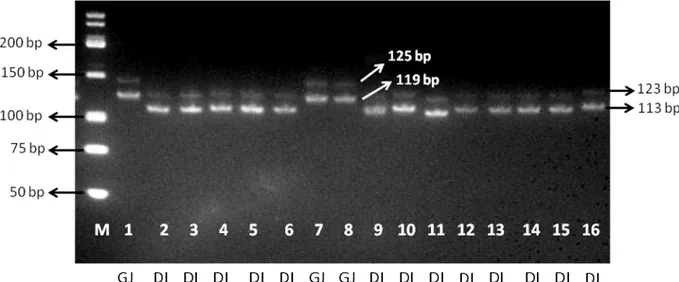
Gambar 1. Visualisasi hasil amplifikasi mikrosatelit lokus ILSTS017 pada mesin PCR (M) Marker; 1-11 Sapi Bali Bertanduk; 12-16 Sapi Polled. Berdasarkan hasil penelitian yang telah dilakukan, Gambar 1 memperlihatkan lokus ILSTS017 dapat mengamplifikasi sampel DNA sapi Bali dengan baik. Dalam proses amplifikasi, suhu annealing sangat menentukan keberhasilan. Beberapa hal yang umum dilakukan untuk optimasi PCR diantaranya adalah suhu annealing (penempelan primer), konsentrasi Mg2+, konsentrasi primer dan konsentrasi DNA target (Viljoen et al., 2005). Suhu
18
DNA cetakan selama proses PCR. Suhu annealing yang digunakan pada
penelitian ini untuk kedua lokus tersebut adalah 60ºC.
Hasil visualisasi menunjukkan bahwa panjang fragmen dapat menentukan
genotip masing-masing ternak. Pada lokus ILSTS017 terdapat 4 jenis alel yang ditemukan, DNA dengan panjang fragmen 113 pb di identifikasi sebagai alel D, panjang fragmen 123 pb sebagai alel I, panjang fragmen 119 pb sebagai alel G dan panjang fragmen 125 pb sebagai alel J. Dengan demikian ternak dengan panjang kedua fragmen 113 pb dan 123 pb bergenotip DI dan ternak dengan panjang fragmen 119 pb dan 125 pb bergenotip GJ. Hal ini sesuai dengan penelitian yang dilakukan Utomo et al. (2011) bahwa alel yang teramplifikasi melalui proses PCR pada lokus ILSTS017 menghasilkan total alel sebanyak 4 alel dengan macam alel D, I, G dan J dengan kisaran panjang alel dari 113-125 pb.
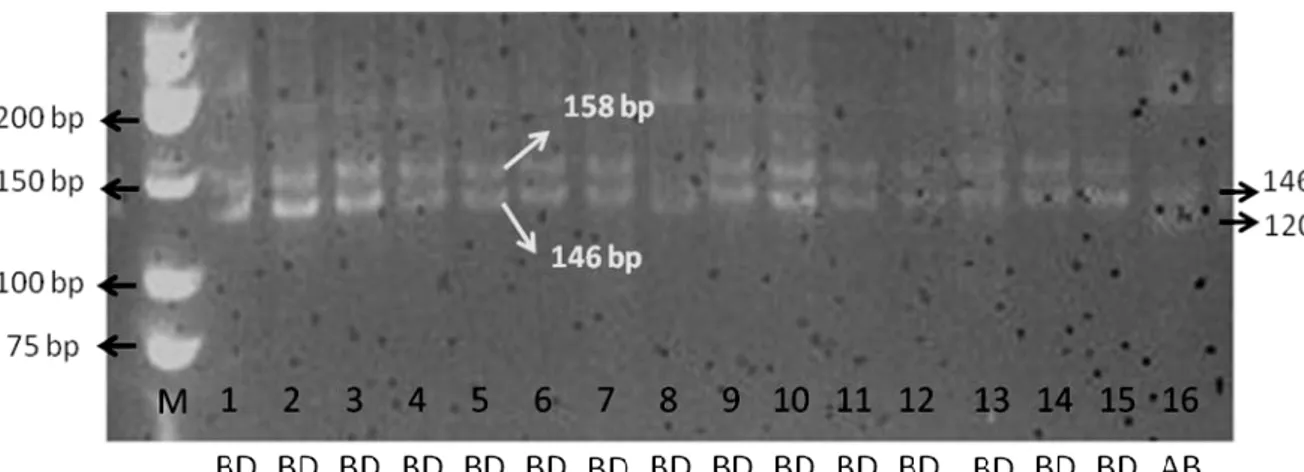
Gambar 3. Visualisasi hasil amplifikasi mikrosatelit lokus ILSTS089 pada mesin
PCR (M) Marker ; 1-8 Sapi Bali Polled ; 9-16 Sapi Bali Bertanduk. Adapun pada lokus ILSTS089 ditemukan 3 jenis alel, dimana panjang
fragmen 120 pb di identifikasi sebagai alel A, panjang fragmen 146 pb sebagai alel B dan panjang fragmen 158 pb sebagai alel D. Ternak dengan panjang kedua fragmen 120 pb dan 146 pb bergenotip AB dan ternak dengan panjang kedua
19
fragmen 146 pb dan 158 pb bergenotip BD. Lokus ILSTS089 menghasilkan total alel sebanyak 3 alel dengan macam alel A, B dan D dengan kisaran panjang 120-158 pb. Sedangkan pada penelitian Kemp et al. (1995) yang menghasilkan panjang fragmen 128 pb.
Hasil amplifikasi tersebut menunjukkan bahwa amplifikasi DNA mikrosatelit bersifat polimorfik pada semua lokus. Nei dan Kumar (2000) menyatakan bahwa variasi genetik terjadi jika terdapat dua alel atau lebih dalam satu populasi dan biasanya lebih dari 1%.
Frekuensi Genotipe dan Alel
Keragaman genetik dari populasi dapat di evaluasi diantaranya melalui jumlah alel per lokus, rata-rata jumlah alel untuk semua lokus dan heterozigositas (Sun et al., 2008). Nei dan Kumar (2000) menyatakan bahwa variasi genetik terjadi jika terdapat dua alel atau lebih dalam satu populasi dan biasanya lebih dari 1%. Hasil analisis frekuensi genotipe dan alel pada lokus ILSTS017 dan ILSTS089 pada sapi Bali dapat dilihat pada Tabel 2 dan Tabel 3.
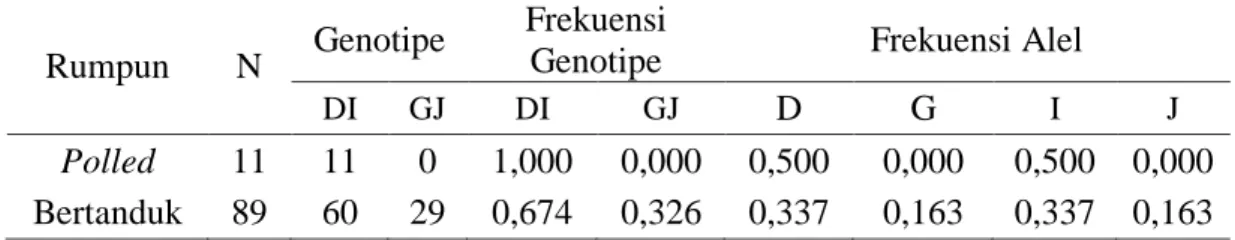
Tabel 2. Frekuensi Genotip dan Alel Lokus ILSTS017
Hasil identifikasi genotipe sapi Bali pada lokus ILSTS017 pada Tabel 2 menunjukkan bahwa sapi Bali Polled mempunyai 1 jenis genotipe yaitu genotipe DI sebanyak 11 ekor, dan sapi Bali bertanduk mempunyai 2 jenis genotipe yaitu genotipe DI sebanyak 60 ekor dan genotipe GJ sebanyak 29 ekor, sedangkan
Rumpun N Genotipe
Frekuensi
Genotipe Frekuensi Alel
DI GJ DI GJ D G I J
Polled 11 11 0 1,000 0,000 0,500 0,000 0,500 0,000
20
genotipe DD, II, GG, dan JJ tidak ditemukan pada sapi Bali Polled maupun yang bertanduk.
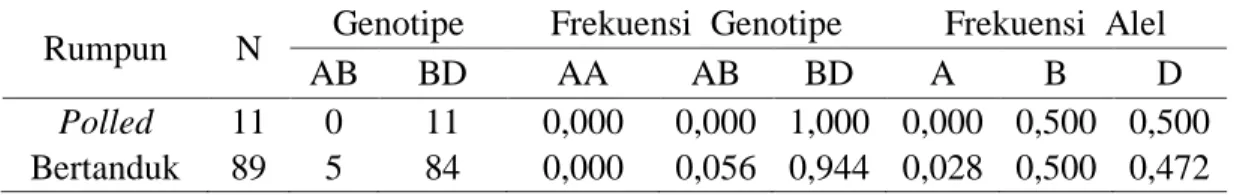
Tabel 3. Frekuensi Genotipe dan Alel Lokus ILSTS089
Adapun hasil identifikasi pada lokus ILSTS089 pada Tabel 3 menunujukkan bahwa sapi Bali Polled mempunyai 1 jenis genotipe yaitu genotipe BD sebanyak 11 ekor, dan sapi Bali bertanduk mempunyai 2 jenis genotipe yaitu genotipe AB sebanyak 5 ekor dam genotipe BD sebanyak 84 ekor. Dari hasil penelitian ini menunjukkan bahwa setiap populasi dari tiap lokus bersifat polimorfik atau beragam. Sifat polimorfik ini ditentukan dengan mengidentifikasi jumlah alel pada suatu populasi. Hal ini sesuai dengan pendapat Nei dan Kumar (2000) yang menyatakan bahwa gen dikatakan polimorfik apabila salah satu alelnya kurang dari 99%. Keragaman dapat ditunjukkan dengan adanya dua alel atau lebih dalam populasi. keragaman genetik terdapat di dalam suatu individu bilamana ada dua alel untuk gen yang sama merupakan perbedaan konfigurasi DNA yang menduduki lokus yang sama pada suatu kromosom (Indrawan et al., 2007).
Nilai Hetorozigositas
Pendugaan nilai heterozigositas sangat penting untuk diketahui sebagai gambaran variabilitas genetik (Marson et al., 2008). Nilai heterozigositas pengamatan (Ho) dan heterozigositas harapan (He) pada lokus ILSTS017 dan
ILSTS089 dapat dilihat pada Tabel 4 dan Tabel 5.
Rumpun N Genotipe Frekuensi Genotipe Frekuensi Alel
AB BD AA AB BD A B D
Polled 11 0 11 0,000 0,000 1,000 0,000 0,500 0,500
21
Tabel 4. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas Harapan (He)
Lokus ILSTS017
Rumpun N H pengamatan H harapan
Polled 11 1,000 0,750
Bertanduk 89 1,000 0,860
Dari Tabel 4 dapat dilihat bahwa pada lokus ILSTS017 pada sapi Bali Bertanduk dan Sapi Bali Polled masing-masing memiliki nilai heterozigositas pengamatan (He) yaitu 1,000. Sedangkan nilai heterozigositas harapan (Ho) pada
sapi Bali Polled yaitu 0,750 dan pada sapi Bali bertanduk yaitu 0,860.
Tabel 5. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas Harapan (He)
Lokus ILSTS089
Rumpun N H pengamatan H harapan
Polled 11 1,000 0,500
Bertanduk 89 1,000 0,527
Adapun pada lokus ILSTS089 dapat dilihat pada Tabel 5 bahwa, nilai heterozigositas pengamatan (He) pada sapi Bali bertanduk dan sapi Bali polled
yaitu 1,000. Sedangkan nilai heterozigositas harapan (Ho) pada sapi Bali Polled
yaitu 0,500 dan pada sapi Bali bertanduk yaitu 0,527.
Perhitungan nilai heterozigositas berdasarkan kaidah Nei (1987) bahwa nilai heterozigositas berkisar antara 0-1, apabila nilai heterozigositas sama dengan 0 (nol) maka diantara populasi yang diukur memiliki hubungan genetik sangat dekat dan apabila nilai heterozigositas sama dengan 1 (satu) maka diantara populasi yang diukur tidak terdapat hubungan genetik atau pertalian genetik sama sekali.
22
KESIMPULAN DAN SARAN
Kesimpulan
Berdasarkan hasil penelitian yang telah dilakukan maka dapat disimpulkan bahwa:
1. Hasil amplifikasi mikrosatelit terhadap lokus ILSTS017 dan ILSTS089 menghasilkan pita/alel yang beragam (polimorfik)
2. Pada lokus ILSTS017 terdapat 4 jenis alel yang ditemukan yaitu alel D, I, G, dan J bergenotipe DI dan GJ, sedangkan pada lokus ILSTS089 terdapat 3 jenis alel yang ditemukan yaitu alel A, B, dan D bergonotipe AB dan BD, 3. Pada Lokus ILSTS017 dan ILSTS089, alel yang ditemukan tidak dapat
digunakan sebagai gen penciri sifat Polled pada sapi Bali.
Saran
Berdasarkan kesimpulan penelitian tersebut maka disarankan untuk melakukan penlitian identifikasi kemurnian sapi Bali Polled menggunakan metode yang lain.
23
DAFTAR PUSTAKA
Abdullah, M.A.N. 2008. Karakterisasi genetik sapi Aceh menggunakan analisis keragaman fenotipik, daerah D-Loop DNA mitokondria dan DNA mikrosatelit [disertasi]. Sekolah Pascasarjana, Institut Pertanian Bogor, Bogor.
Batan, I.W. 2006. Sapi Bali dan Penyakitnya. Fakultas Kedokteran Hewan Universitas Udayana. Denpasar.
Bennet, P. 2000. Microsatellites. J. Clin. Pathol. Mol. Pathol. 53: 177-183.
Bloot, S. C., J. L. Williams, and C. S. Haley. 1998. Genetics relationship among European cattle breeds. Anim Genet. 29: 273-282.
Brockmann, G. A., Martin, J., Teuscher, F., and Schwerin, M. 2000. Marker controlled inheritance of the polled locus in Simmental cattle. Arch. Tierz.,
43(3), 207–212.
Bugiwati S.R.A. 2007. Pertumbuhan dimensi tubuh pedet jantan sapi Bali di Kabupaten Bone dan Barru Sulawesi Selatan. J Sains Teknol 7(2):103-108. Cargill, E.J., Nissing, N.J. and Grosz, M.D., 2008. Single nucleotide
polymorphisms concordant with the horned / polled trait in Holsteins. BMC
Research Notes, 9(1), pp.1–9.
Chaudhari, MV., Parmar S.N.S, Joshi C.G, Bhong C.D, Fatima S, Thakur MS, and Thakur SS. 2009. Moleculer characterization of Kenkatha and Gaolao (Bos indicus) cattle breeds using microsatellite markers. Anim Biodiv and
Conserv 32(2): 71-78.
Comings, D.E. 1998. Polygenic inheritance and micro minisatellites. Molecular Psychiatry 3:21-31.
Dianti. 2014. Pengertian Keragaman Genetik. http://www. Sridianti
.com/pengertian-keragaman-genetik.html.Diakses pada tanggal 7 Oktober 2017.
Fitzsimmons, C.J., S.M. Schmutz, R.D. Bergen and J.J. McKinnon. 1998. A potential association between the BM1500 microsatellite and fat deposition in beef cattle. Mamm. Genome 9:432-434.
Gunawan, D., Pamungkas dan L. Affandhy. 2004. Sapi Bali, Potensi, Produktivitas dan Nilai Ekonomi. 6 Ed. Kanisius. Yogyakarta.
24
Handiwirawan, E. dan Subandriyo. 2004. Potensi dan keragaman sumberdaya genetik sapi bali. Lokakarya Nasional Sapi Potong. Pusat Penelitian dan Pengembangan Peternakan. Hlm. 50-60.
Hardjosubroto, W. dan J.M. Astuti. 1993. Buku Pintar Peternakan. Jakarta: Pt Gramedia Widiasarana Indonesia.
Hendrick, P. W. 2000. Genetics of Population. 2nd Ed. Jones and Barlett Publishers. Sudbury. Massachussetts.
Indrawan, M., R.B. Primack dan J. Supriatna. 2007. Biologi Konservasi. Yayasan Obor Indonesia. Jakarta.
João F.C. de Oliveira, J.P. Neves, E.A. Almeida, C.S. Steigleder, J.C.F. Moraes, P.B.D. Gonçalves and T.A. Weimer. 2005. Association between reproductive traits and four microsatellites in Brangus-Ibagé cattle. Genetics and Molecular Biology, 28: 54-59.
Kantanen, J., I. Olsaker, L. E. Holm, S. Lien, J. Vilkki, K. Brusgaard, E. Eythorsdottir, B. Danell and S. Adalsteinsson. 2000. Genetic diversity and population structure of 20 North European cattlebreeds. Journal of Heredity 91: 446–57.
Karthickeyan SMK, Kumarasamy P, Sivaselvam SN, Selvam R, and Thangareju P. 2008. Analysis of microsatellite markers in Ongole breed of cattle. Indian
J Biotech 7:113-116.
Kemp, S.J., O. Hishida, J. Wambugu, A. Rink, A.J. Teale, M.L. Longeri, R. Z. MA, Y. DA, H.A. Lewin, W. Barendse and A.J. Teale. 1995. A panel of polymorphic bovine, ovine and caprine microsatellite markers. Anim. Genet. 26: 299-306.
Lauwerier, R.C.G.M., 2015. Polled cattle in the Roman Netherlands. Livestock
Science, 179, pp.71–79.
Lumban Gaol, A. D., Suatha, I. K., dan Wandina, I. N. 2013. Struktur Genetika Populasi Kambing Menggunakan Marka Molekul Mikrosatelit D18S536. Jurnal Indonesia Medicus Veterinus 2(1): 32-42.
Marson EP, Ferraz JBS, Meirelles FV, and Baliero JCC. 2008. Effect of polymorphisme of LHR and FSHR genes on sexual precocity in a Bos taurus x Bos Indicus beef composite population. Gen Mol Res 7:243-251.
Martodjo H. 2003. A Simple Selection Program for Smallholder Bali Cattle Farmers. In : Strategies to Improve Bali Cattle in Eastren Indonesia. K. Entwistle and D.R . Lindsay (Eds). ACIAR Proc. No. 110. Canberra.
25
Maskur, M. dan B. T. 2007. Identifikasi Genetik Menggunakan Marker Mikrosatelit dan Hubungannya dengan Sifat Kuantitatif pada Sapi. Media
Peternakan, 30(3), 147–155.
Masudana, I W. 1990. Perkembangan Sapi Bali di Bali dalam Sepuluh Tahun Terakhir (1980- 1990). Proceeding Seminar Nasional Sapi Bali. Denpasar, 20-22 September 1990. Denpasar: Fakultas Peternakan Universitas Udayana. Hlm A-11-A-30.
Metta M, Kanginakudru S, Gudisewa S, and Nagaraju J. 2004. Genetic characterization of the Indian cattle breeds, Ongole and Deoni (Bos indicus), using microsatellite markers – a preliminary study. BMC Genetics 5(16):1-5. Mohamad, K., M. Olsson, H.T.A. Van Tol, S. Mikko, B.H. Vlamings, G.
Andersson, H.R. Martinez, B. Purwantara, R.W, and Paling, B. Colenbranderand J. A. Lenstra. 2009. On the origin of Indonesia cattle. Plos One 4(5): e5490.
Ismail. M, dan Nuraini, R.P., 2014. Perlemakan pada Sapi Bali dan Sapi Madura Meningkatkan Bobot Komponen Karkas dan Menurunkan Persentase Komponen Nonkarkas. Jurnal Veteriner, 15(3), pp.417–424.
Murtidjo, B. A. 1990. Beternak Sapi Potong. Kanisius Yogyakarta.
Namikawa T, Otsuka J, and Martojo H. 1980. Coat colour variations of Indonesian cattle. The origin and phylogeny of Indonesian native livestock
(Part III): Morphological and genetically investigations on the
interrelationship between domestic animals and their wild forms in Indonesia.
The Research Group of Overseas Scientific Survey 31-34.
Nei, M. 1987. Molecular Evolution and Genetics. Columbia University Press, New York.
Nei, M. and Kumar S. 2000. Molecular Evolutionery And Phylogenetics. Oxford University Press, New York.
Newton, C.R., and Graham. 1994. Polymerase Chain Reaction. Bios Scientific Publishers.
Oka, I.G.L. and D. Darmadja. 1996. History and development of Bali Cattle. Proceedings seminar on Bali cattle, a special spesies for the dry tropics, held by Indonesia Australia Eastern University Project (IAEUP), 21 September 1996. Udayana University Lodge, Bukit Jimbaran, Bali.
Oliveira, J.F.C., J.P. Neves, J.C.F. Moraes, P.B.D. Gonçalves, J.M. Barr, A.G.V. Hernandez and G. Bonotto. 2002. Follicular population and steroid levels in Brangus Ibagé with distinct levels of fertility. Anim. Reprod. Sci. 73:1-10.
26
Pane, I. 1986. Pemuliabiakan Ternak Sapi. PT. Gramedia, Jakarta.
Payne WJA, and Hodges J. 1997. Tropical Cattle: Origin, Breeds and Breeding
Policies. Blackwell Science.
Payne, W.J.A. and D.H.L. Rollinson. 1973. Bali Cattle. World Anim. Rev. 7: 13-21.
Purwantara, B., R.R. Noor, G. Anderson, and H. Rodriguez-Martinez. 2012. Banteng and Bali cattle in Indonesia: status and forecast. Reprod Dom Anim, 47: 2-6.
Rincon AMS, Montiel HP, and Bracamonte GMP. 2007. Assessment of genetic structure in Mexican charolais herds using microsatellite markers. Mol Biol
and Gen 10(4):1-7.
Sarbaini. 2004. Kajian keragaman karakteristik eksternal dan DNA mikrosatelit sapi pesisir Sumatera Barat [disertasi]. Bogor: Sekolah Pascasarjana, Institut Pertanian Bogor.
Sastradipradja, D. 1990. Potensi internal sapi Bali sebagai salah satu sumber plasma nutfah untuk menunjang pembangunan peternakan sapi potong dan ternak kerja secara nasional. Pros. Seminar Nasional Sapi Bali. Denpasar, 20-22 September. Denpasar: Fakultas Peternakan Universitas Udayana. Hlm A-47–A54.
Satriani N, Farajallah A, dan Muladno. 2002. Keragaman genetik Sapi Peranakan Ongole (PO) berdasarkan uji DNA mikrosatelit. Med Pet (25)3:84-91.
Sun W, Chen H, Lei C, Lei X, and Zhang Y. 2008. Genetic variation in eight Chinese cattle breeds based on the analysis of microsatellite markers. Genet
Sel Evol 40:681-692.
Tegelstrom, H. 1992. Mithocondrial DNA in natural population: An improved routine for screening of genetic bariation breed on sensitive silver staining. Electrophoresis. 7:226-22.
Teneva A. 2009. Molecular markers in animal genome analysis. Biotech Anim
Husb 25(5-6):1267-1284.
Utomo, B.N., R.R. Noor, C. Sumantri, I. Supriatna, dan E.D. Gurnardi. 2010. Keragaman morfometrik dan fenotipik sapi Katingan di Kalimantan Tengah. JITV. 15.
Viljoen, G. J., L. H. Nel and J. R. Crowther. 2005. Molecular Diagnostic PCR
Handbook. Springer, Dordrecht, Netherland.
Weir BS. 1996. Genetic Data Analysis : Mthod for Discrete Population Genetic Data. Second ed. Sunderland, MA USA. Sinauer Associates.
27
Winaya A, Muladno, dan Tappa B. 2007. Panel 16 lokus mikrosatelit untuk deteksi polimorfisme dan hubungan filogenetik pada genom sapi. Med Pet 24(2): 81-88.
Zulkharnaim, Jakaria, dan D.Ronny.R.N., 2010. Identifikasi Keragaman Genetik Gen Reseptor Hormon Pertumbuhan (GHR). Media Peternakan, 33(2), pp.81–87.
1
28
29
RIWAYAT HIDUP
DINDA FEBRIANTI ADAM, lahir di Jakarta pada tanggal
03 Februari 1995, sebagai anak pertama dari lima bersaudara dari pasangan bapak H. Heri Husary Adam dan ibu Hj. Fauziah. Jenjang pendidikan formal penulis dimulai dari SD Negeri 11 Pangkajene Sidrap ,lulus tahun 2007. Setelah lulus di SD, kemudian malanjutkan di SMP Ramatul Asri Enrekang, lulus tahun 2010, dan SMA Ummul Mukminin Makassar, lulus pada tahun 2013. Setelah menyelesaikan SMA, penulis diterima di Perguruan Tinggi Negeri (PTN) melalui jalur Seleksi Bersama Masuk Perguruan Tinggi Negeri (SBMPTN) pada Program Studi Peternakan, Fakultas Peternakan, Universitas Hasanuddin, Makasssar. Selama kuliah penulis pernah menjadi Asisten di Laboratorium Ternak Potong. Penulis juga merupakan anggota Himpunan Mahasiswa Produksi Ternak Universitas Hasanuddin (HIMAPROTEK-UH),