IDENTIFIKASI KERAGAMAN GEN UTMP
EXON
4 PADA SAPI
BALI
NENIK TRI WAHYUNI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN
FAKULTAS PETERNAKAN
INSTITUT PERTANIAN BOGOR
BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN SUMBER
INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Identifikasi Keragaman Gen UTMP Exon 4 pada Sapi Bali adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Mei 2014
Nenik Tri Wahyuni
ABSTRAK
NENIK TRI WAHYUNI. Identifikasi Keragaman Gen UTMP Exon 4 pada Sapi Bali. Dibimbing oleh JAKARIA dan ASEP GUNAWAN.
Sapi bali sebagai sumber daya genetik ternak asli Indonesia yang memiliki beberapa keunggulan, salah satunya memiliki kemampuan reproduksi yang tinggi. Gen Uterine Milk Protein (UTMP) adalah gen yang diduga kuat berpengaruh terhadap masa hidup produktif yang berkaitan dengan sifat reproduksi yang berperan penting dalam mendukung konsepsi selama kebuntingan. Tujuan penelitian ini adalah mengidentifikasi ada tidaknya polimorfisme gen UTMP sebagai pengontrol sifat reproduksi pada sapi bali dengan metode Polymerase Chain Reaction-Restriksi Fragment Length Polymorphism (PCR-RFLP). Sebanyak 123 sampel darah yang berasal dari BPTU sapi bali dan BIBD dideteksi keragaman gen UTMP menggunakan enzim restriksi BsrI yang memotong sekuens basa C|CAGT. Amplifikasi gen UTMP menghasilkan fragmen dengan panjang 406 bp pada posisi di exon 4 dengan suhu annealing 60 ⁰C. Hasil analisis PCR-RFLP menunjukkan bahwa sapi bali yang dianalisis memiliki alel monomorfik AA sampel BBTU dan BIBD sebesar 100%. Hasil sekuen fragmen gen UTMP exon 4 menunjukkan tidak ditemukan mutasi pada gen tersebut pada sapi Bali tetapi ditemukan insersi motif DNA minisatelit sepanjang 77 basa.
Kata kunci : gen Uterine Milk Protein (UTMP), PCR-RFLP, sapi bali
ABSTRACT
NENIK TRI WAHYUNI. Identification of UTMP Gene Polymorphisms Exon 4 In Bali Cattle . Supervised by JAKARIA and ASEP GUNAWAN.
The bali cattle, one of Indonesian specific animal genetic resource, has many advantage, one of that advantage is high in reproductivity. Uterine Milk Protein (UTMP) gene is expected having an effect on lifes’ productivity related to the characteristic of reproduction and has main function in supporting conception during pregnancy. The aim of this research was to identify the existence of UTMP polymorphisms gene as controller of reproductions characteristic in Bali cattle using the PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Length Polymorphism). One hundred twenty three blood samples of bali cattle derived from BPTU and BIBD was detected its presence of genetic diversity at UTMP gene using BsrI which restric C|CAGT basic sequences. UTMP gene amplification was performed with temperature of annealing 60 ⁰C. The results of PCR-RFLP analysis indicated that bali cattle have been analysed monomorphic A allele at BPTU and BIBD was 100%. Result of sequence fragment UTMP gene exon 4 was indicate insersi mutation 77 basic until long change sequence it bands 406 base pair.
IDENTIFIKASI KERAGAMAN GEN UTMP
EXON
4 PADA SAPI
BALI
NENIK TRI WAHYUNI
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Peternakan
pada
Departemen Ilmu Produksi dan Teknologi Peternakan
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN
FAKULTAS PETERNAKAN
INSTITUT PERTANIAN BOGOR
BOGOR
Judul Skripsi : Identifikasi Keragaman Gen UTMP Exon 4 Pada Sapi Bali Nama : Nenik Tri Wahyuni
NIM : D14100116
Disetujui oleh
Pembimbing I
Dr Jakaria, SPt MSi Dr agr Asep Gunawan, SPt MSc
Pembimbing II
Diketahui oleh
Ketua Departemen Prof Dr Ir Muladno, MSA
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan November 2013 hingga Januari 2014 ini ialah gen reproduksi pada sapi Bali, dengan judul Identifikasi Keragaman Gen UTMP Exon 4 pada Sapi Bali.
Terima kasih penulis ucapkan kepada Bapak Dr Jakaria, SPt MSi selaku pembimbing akademik dan pembimbing skripsi, Bapak Dr agr Asep Gunawan, SPt MSc selaku dosen pembimbing skripsi atas waktu, tenaga, saran, dan kesabaran yang telah diberikan. Terimakasih juga penulis ucapkan kepada Bapak Edit Lesa Aditya SPt MSc selaku dosen penguji sidang yang telah memberikan masukan dan saran dalam penulisan skripsi ini. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya. Tak lupa penulis sampaikan terimakasih kepada Ricky SF, Devi S, Luthfia I, Sherly J, Irine FZ, Kiky U, Dhini, Kak Alit, Kak Ferdi, Kak Erik, Kak Irine, Kak Furqon, Ishfi dan Ria PR. Semoga karya ilmiah ini bermanfaat.
Bogor, Mei 2014
DAFTAR ISI
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Ruang Lingkup Penelitian 2
METODE 2
Lokasi dan Waktu penelitian 2
Bahan 2
Alat 3
Prosedur 3
Ekstraksi DNA 3
Amplifikasi DNA 3
Genotyping Gen UTMP 4
Sekuens Fragmen Gen UTMP Exon 4 5
Analisis Data 5
HASIL DAN PEMBAHASAN 5
Amplifikasi Gen UTMP 5
Penentuan Genotipe Gen UTMP 6
Frekuensi Alel Gen UTMP 7
Analisis sekuens gen UTMP 8
SIMPULAN DAN SARAN 10
Simpulan 10
Saran 10
DAFTAR PUSTAKA 11
LAMPIRAN 13
DAFTAR GAMBAR
1 Posisi Penempelan primer pada fragmen gen UTMP dan situs pemotongan
enzim BsrI 4
2 Hasil amplifikasi gen UTMP pada gel agarose1.5% 6
3 Penentuan genotip gen UTMP|BsrI 7
4 Pola Insersi pada gen UTMP sepanjang 77 basa 8
5 Rekonstruksi gambar gen UTMP 9
DAFTAR LAMPIRAN
1 Sekuens gen uterine milk protein (UTMP) yang diakses di GanBank dengan kode akses L22095 132 Hasil BLAST nukleotida sekuens gen UTMP sapi Bali 14
1
PENDAHULUAN
Latar Belakang
Sapi bali merupakan sapi hasil dari domestikasi banteng (Bos javanicus) (Nijman et al. 2003) dan merupakan salah satu sumberdaya genetik ternak asli Indonesia yang perlu dipertahankan dan dilestarikan keberadaannnya. Populasi sapi bali tahun 2013 diperkirakan sekitar 4.483.890 (Ditjennak 2013) dan berkontribusi sekitar 27% terhadap total sapi Indonesia. Daerah sumber bibit sapi bali yang difokuskan dalam penelitian ini adalah BPTU dan BIBD sapi bali di pulau Bali. Di BPTU sapi bali , berbagai teknik dan kiprah serta pengembangan plasma nutfah kebanggaan Indonesia terus berjalan hingga saat ini. BPTU dan BIBD merupakan daerah penghasil bibit-bibit unggul dari sapi bali yang dianggap mempunyai keunggulan sebagai penghasil bibit. Pada sapi Bali telah dilakukan program nasional pemurnian dan peningkatan mutu genetik ternak sejak tahun 1976 di pulau Bali sesuai dengan Surat Keputusan (SK) Menteri Pertanian nomor 776/Kpts/Um/12/1976.
Sapi bali mempunyai keunggulan yang tidak dimiliki sapi bangsa lain diantaranya memiliki kemampuan beradaptasi dengan lingkungan marginal seperti kualitas pakan yang rendah, iklim tropis, ketersediaan air yang kurang, manajemen yang kurang baik dan ketahanan terhadap parasit (Zulkharniem et al.
2010). Selain itu sapi bali mempunyai kemampuan fertilitas berkisar antara 83%-86 % (Siswanto et al. 2013) dan persentase karkas yang tinggi serta kadar lemak yang rendah (Bugiwati 2007). Siswanto et al. (2013) melaporkan bahwa penampilan reproduksi sapi bali yang dipelihara secara intensif adalah umur sapi bali mengalami birahi pertama 718.57± 12.65 hari, umur pertama melahirkan 1104.51± 23.82 dan calving interval 350.46±27.98 hari. Namun disamping kelebihannya tersebut, terdapat beberapa kelemahan pada sapi Bali diantaranya memiliki produksi susu yang relatif rendah dan tingkat kematian anak yang cukup tinggi mencapai 30% (Tolihere 2002). Seleksi sapi bali dengan kemampuan hidup yang tinggi untuk kelangsungan hidup anak sangat penting untuk dilakukan melalui seleksi yang akurat.
Aplikasi penanda molekuler memungkinkan untuk digunakan sebagai alat seleksi yang akurat melalui pendekatan kandidat gen karena dimungkinkan dapat diprediksi sejak awal dan diturunkan dari generasi ke generasi. Salah satu gen yang diduga kuat berpengaruh terhadap masa hidup produktif dan produksi susu yaitu gen Uterine Milk Protein (UTMP). UTMP atau lebih dikenal dengan protein susu rahim adalah protein utama yang disekresikan oleh endometrium dibawah kontrol dari progestron dan ekspresinya dominan ditemukan pada endometrium yang mengindikasikan berperan penting dalam mendukung konsepsi selama masa kebuntingan (Ing et al. 1989).
Pada ternak lain seperti domba dan babi, UTMP dikenal juga sebagai serin
2
kromosom 21 dalam wilayah genomic 321.6 kb yang terdiri dari 5 exon (Ulbrich
et al. 2009). Lebih lanjut dilaporkan identifikasi keragaman gen UTMP ditemukan berhubungan nyata terhadap masa hidup produktif pada sapi holstein (Khatib et al. 2007). Berdasarkan informasi tersebut, gen UTMP di yakini sebagai salah satu kandidat gen yang dapat digunakan untuk memperbaiki masa hidup produktif dan produksi susu pada sapi bali.
Tujuan Penelitian
Tujuan dari penelitian ini adalah mengidentifikasi keragaman gen UTMP pada posisi exon 4 sapi bali di Balai Pembibitan Ternak Unggul dan Balai Inseminasi Buatan Daerah pulau Bali.
Ruang Lingkup Penelitian
Penelitian ini meliputi analisis pada 100 ekor sapi bali yang terdapat di Balai pembibitan Ternak Unggul (BPTU) dan 23 ekor sapi Balai Inseminasi Buatan Daerah sapi bali, pulau Bali. Keragaman gen UTMP dianalisis menggunakan teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Ada tidaknya mutasi di fragmen gen UTMP exon 4 dianalisis melalui teknik sequencer. Analisis data dilakukan melalui pendekatan frekuensi alel dan analisis sekuens menggunakan Basic Local Alignment Search Tools (BLAST) nukleotida ( Altchul et al. 1997).
METODE
Lokasi dan Waktu Penelitian
Penelitian ini dilakukan di Laboratorium Pemuliaan dan Genetika Ternak Departemen Ilmu Produksi dan Teknologi Peternakan Institut Pertanian Bogor. Penelitian dilakukan pada bulan November 2013-Januari 2014.
Bahan
Sampel DNA yang dianalisis berasal dari sampel darah 100 ekor sapi bali di BPTU dan sampel semen 23 ekor sapi bali di BIBD pulau Bali yang merupakan koleksi Laboratorium Genetika Molekuler Ternak. Bahan yang digunakan untuk ekstraksi DNA adalah adalah DW (Destilation water), NaCl , 1x STE, Proteinase -K, SDS 10% (Sodium Dodesil Sulfat) , larutan fenol, CIAA (Kloroform Iso Amil Alkohol), etanol absolut, dan bufer TE 80% (tris EDTA). Bahan yang digunakan untuk amplifikasi, elektroforesis dan analisis RFLP adalah sampel DNA hasil ekstraksi, destilation water (DW), 10 x buffer, MgCl2, pasangan primer forward
3
Nucleotide Triphospat (dNTPs), produk PCR, agarose, 0.5 x Tris-Borat EDTA (TBE), Ethidium Bromide (EtBr), loading dye (0.01% Xylene Cyanol, 0.01% Bromthymol blue dan 50% gliserol), marker 100 bp, enzim restriksi BsrI, dan bufer B.
Bahan yang digunakan untuk analisis sekuensing adalah produk PCR dan
primer forward fragmen gen UTMP. Primer yang digunakan untuk
mengamplifikasi gen Uterine Milk protein (UTMP|BsrI) berdasarkan Khatib et al. (2007) adalah forward: 5´- GGCCCTACATCAAGCTGAGA -3´ dan reverse: 5´ -CTACTCAACTTGGGGGTTGA -3´ dengan panjang produk 327 bp.
Alat
Alat yang digunakan antara lain 1 set pipet mikro dan tipnya, tabung
eppendorf, vortexmixer, refrigerator, alat sentrifugasi, freezer, mesin PCR, 1 set alat pencetak gel, power supply 100 volt, microwave, stearer, gelas ukur, dan tabug erlenmeyer.
Prosedur Ekstraksi DNA
Eksktraksi DNA dilakukan berdasarkan metode Sambrook et al. (1989). Ekstraksi dilakukan pada 2 jenis sampel berbeda yaitu sampel darah dan sampel semen. Yang membedakan dari kedua sampel tersebut dalam ekstraksi DNA yaitu volume awal yang digunakan. Sampel darah segar yang digunakan dalam ekstraksi sebanyak 200 μL sedangkan untuk sampel semen yang digunakan sebanyak 400 μL. Sampel dimasukkan dalam tabung 1.5 mL, selanjutnya ditambahkan 1 000 μL larutan NaCl 0.2%. Sampel di sentrifugasi pada kecepatan 8 000 rpm selama 5 menit, lalu bagian supernatannya dibuang. Kemudian
ditambahkan 350 μL larutan 1xSTE (Sodium tris EDTA), 40 μL SDS 10%
(sodium dodesil sulfat), dan 20 μL proteinase K. Sampel dikocok pelan dalam inkubator pada suhu 55 oC selama 2 jam. Molekul DNA selanjutnya dimurnikan dengan metode fenol-chloroformyaitu dengan ditambahkan 400 μL larutan fenol,
400 μLchloroform iso amil alcohol (CIAA), dan 40 μL 5M NaCl, lalu dikocok pelan pada suhu ruang selama 1 jam. Kemudian disentrifugasi selama 5 menit dengan kecepatan 12 000 rpm.
Bagian DNA yang bening dipindahkan pipet ke tabung 1.5 mL yang baru.
Selanjutnya ditambahkan 800 μL EtOH absolut dan 40 μL 5M NaCl. Molekul DNA kemudian disimpan selama semalam (over night) pada suhu -20 oC. Molekul DNA kemudian dipisahkan dari etanol absolut dengan cara disentrifugasi selama 5 menit dengan kecepatan 12 000 rpm. Bagian supernatan dibuang dan didiamkan dalam keadaan terbuka hingga alkohol tersebut kering. Kemudian endapan yang diperoleh disuspensikan dalam 100 μL TE (tris EDTA).
Amplifikasi DNA
4
μL sampel DNA yang sudah di ekstraksi, dan master mix yang terdiri dari 10.75
μL DW, 1.5 μL buffer, 1 μL MgCl, 0.3 μL dNTPs , 0.2 μL primer forward dan 0.2
μL primer reverse, dan 0.05 taq polymerase.
Campuran tersebut kemudian dimasukkan ke dalam mesin PCR dengan kondisi suhu predenaturasi 95 oC selama 5 menit, 35 siklus (denaturasi 95 oC selama 10 detik, annealing 60 ⁰C selama 20 detik, dan ekstensi 72 oC selama 30 detik) dan post ekstensi 72 oC selama 5 menit, elongasi akhir pada suhu 72 ⁰C selama 5 menit dalam 1 siklus. Produk PCR dielektroforesis menggunakan
agarose 1.5%. Posisi penempelan primer dan situs pemotong enzim BsrI pada sekuens gen UTMP exon 4 dapat dilihat pada Gambar1.
Genotyping Gen UTMP
Genotyping dengan metode Restriction Fragment Length Polymorphism
(RFLP) dilakukan dengan menggunakan produk PCR sebanyak 5μL yang dipindahkan ke dalam tabung 0.5 mL dan ditambahkan 1 μL DW, 0.3 μL enzim restriksi BsrI dan 0.7 bufer BsrI. Kemudian campuran tersebut diinkubasi pada suhu 65 ⁰C selama 16 jam. Sampel DNA yang telah dipotong dengan enzim restriksi kemudian dielektroforesis pada gel agarose 2% dengan tegangan 100 volt selama 30 menit.
Elektroforesis produk PCR-RFLP diawali dengan membuat gel agarose
2% yang dibuat dengan cara melarutkan 0.6 g agarose dalam larutan 0.5 x TBE sebanyak 30 mL kemudian dipanaskan dalam microwave selama 5 menit, dikocok menggunakan magnetic stirrer dan ditambahkan 2.5 μL EtBr. Agarose yang sudah dipanaskan dituang dalam pencetak gel , sisir ditempatkan pada tepian gel hingga mengeras. Setelah gel mengeras kemudian sisir dicabut hingga terbentuk sumur-sumur gel. Selanjutnya gel ditempatkan dalam gel tray elektroforesis yang berisi larutan buffer.
Produk PCR-RFLP sebanyak 5 μL dicampur dengan 1 μL loading dye
kemudian dimasukkan dalam sumur-sumur gel. Sumur bagian sebelah kiri digunakan untuk menempatkan marker DNA 100 bp sebanyak 2 μL. Gel kemudian dialiri listrik 100 volt selama 30-45 menit. Molekul DNA yang bermuatan negatif pada pH netral akan bergerak ke arah positif (anode). Setelah di elektroforesis gel agarose dilihat panjang pita DNAnya dengan menggunakan
5
sinar ultraviolet dengan mesin UV Transiluminator dan dibandingkan dengan
marker untuk mengetahui panjang fragmennya.
Sekuens Fragmen GenUTMP Exon 4
Sekuens fragmen gen UTMP exon 4 dilakukan pada individu sapi bali bergenotipe AA dengan jumlah 1 sampel. Primer yang digunakan untuk sekuens fragmen gen UTMP hanya primer forward. Sekuensing gen UTMP dilakukan dengan menggunakan mesin sekuenser (ABI Prims 3100-Avant Genetic Analyzer) di perusahaan sekuensing 1st Base, Selangor, Malaysia.
Analisis Data Frekuensi Alel Gen UTMP
Keragaman alel yang terdapat pada sapi bali di BPTU dan BIBD yang telah dianalisis kemudian dihitung dengan pendekatan frekuensi alel. Nilai Frekuensi alel dan genotip (Xi) UTMP diperoleh menggunakan rumus sebagai berikut (Nei dan Kumar 2000):
Xi =2nii + ∑nij 2N
Keterangan :
Xi = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii
nij = jumlah individu bergenotipe ij
N = jumlah individu sampel
Homologi Gen UTMP Exon 4
Hasil sekuens dianalisis dengan program BioEdit (Hall 1999). Selanjutnya untuk mengetahui kesamaan dengan gen UTMP yang terdapat di GenBank dianalisis menggunakan metode BLAST (www.ncbi.nhl.nih.gov./BLAST). Untuk merekonstruksi atau melihat ada tidaknya mutasi pada sekuens fragmen gen UTMP pada exon 4 dilakukan analisis menggunakan program Molecular Evolutionary Genetic Analysis (MEGA5) dengan metode ClustalW (Tamura et al.
2011).
HASIL DAN PEMBAHASAN
Amplifikasi Gen UTMP
Amplifikasi fragmen gen UTMP pada sapi bali dengan menggunakan metode Polymerase Chain Reaction (PCR) pada mesin thermocycler dengan suhu
6
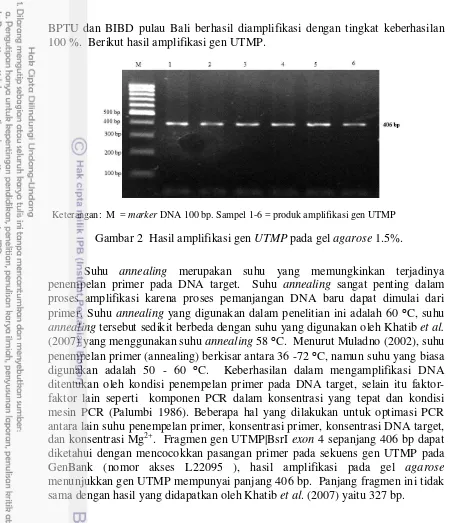
BPTU dan BIBD pulau Bali berhasil diamplifikasi dengan tingkat keberhasilan 100 %. Berikut hasil amplifikasi gen UTMP.
Suhu annealing merupakan suhu yang memungkinkan terjadinya penempelan primer pada DNA target. Suhu annealing sangat penting dalam proses amplifikasi karena proses pemanjangan DNA baru dapat dimulai dari primer. Suhu annealing yang digunakan dalam penelitian ini adalah 60 ⁰C, suhu
annealing tersebut sedikit berbeda dengan suhu yang digunakan oleh Khatib et al.
(2007) yang menggunakan suhu annealing 58 ⁰C. Menurut Muladno (2002), suhu penempelan primer (annealing) berkisar antara 36 -72 ⁰C, namun suhu yang biasa digunakan adalah 50 - 60 ⁰C. Keberhasilan dalam mengamplifikasi DNA ditentukan oleh kondisi penempelan primer pada DNA target, selain itu faktor-faktor lain seperti komponen PCR dalam konsentrasi yang tepat dan kondisi mesin PCR (Palumbi 1986). Beberapa hal yang dilakukan untuk optimasi PCR antara lain suhu penempelan primer, konsentrasi primer, konsentrasi DNA target, dan konsentrasi Mg2+. Fragmen gen UTMP|BsrI exon 4 sepanjang 406 bp dapat diketahui dengan mencocokkan pasangan primer pada sekuens gen UTMP pada GenBank (nomor akses L22095 ), hasil amplifikasi pada gel agarose
menunjukkan gen UTMP mempunyai panjang 406 bp. Panjang fragmen ini tidak sama dengan hasil yang didapatkan oleh Khatib et al. (2007) yaitu 327 bp.
Penentuan Genotipe Gen UTMP
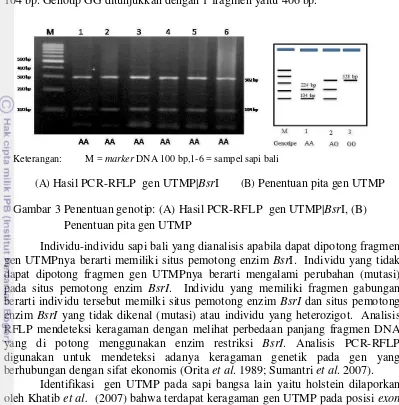
Penentuan genotip dari gen UTMP pada sapi bali dilakukan dengan menggunakan metode PCR-RFLP menggunakan enzim restriksi BsrI yang akan mengenali situs pemotongan C|CAGT yang terletak di daerah exon 4. Hasil produk PCR fragmen gen UTMP|BsrI (406 bp) yang dipotong dengan enzim BsrI
menghasilkan 2 macam fragmen yaitu pada 302 bp dan 104 bp dan diperoleh 1 macam genotip yaitu AA (Gambar 3A) fenomena ini berbeda dengan hasil yang diperoleh Khatib et al. (2007) (Gambar 3B). Pada lokus UTMP|BsrI, ternak sapi dikatakan memiliki genotipe AA jika terdapat 2 fragmen DNA yaitu 302 bp dan
Keterangan: M = marker DNA 100 bp. Sampel 1-6 = produk amplifikasi gen UTMP
7
104 bp. Genotipe AG ditunjukkan dengan 3 fragmen yaitu 406 bp, 302 bp, dan 104 bp. Genotip GG ditunjukkan dengan 1 fragmen yaitu 406 bp.
Individu-individu sapi bali yang dianalisis apabila dapat dipotong fragmen gen UTMPnya berarti memiliki situs pemotong enzim BsrI. Individu yang tidak dapat dipotong fragmen gen UTMPnya berarti mengalami perubahan (mutasi) pada situs pemotong enzim BsrI. Individu yang memiliki fragmen gabungan berarti individu tersebut memilki situs pemotong enzim BsrI dan situs pemotong enzim BsrI yang tidak dikenal (mutasi) atau individu yang heterozigot. Analisis RFLP mendeteksi keragaman dengan melihat perbedaan panjang fragmen DNA yang di potong menggunakan enzim restriksi BsrI. Analisis PCR-RFLP digunakan untuk mendeteksi adanya keragaman genetik pada gen yang berhubungan dengan sifat ekonomis (Orita et al. 1989; Sumantri et al. 2007).
Identifikasi gen UTMP pada sapi bangsa lain yaitu holstein dilaporkan oleh Khatib et al. (2007) bahwa terdapat keragaman gen UTMP pada posisi exon
4 posisi 1 779 (A/G) dan 1 296 (A/G). Hasil penelitian menunjukkan adanya keragaman pada posisi 1 296 yang berhubungan erat dengan kenaikan masa hidup produktif pada sapi holstein. Lebih lanjut dikemukakan genotipe GG dari gen UTMP mempunyai daya tahan hidup embrio yang secara signifikan lebih rendah bila dibandingkan genotipe lainnya. Ing et al. (1989) menjelaskan bahwa sintesis UTMP berada di bawah kendali progesteron dan ekspresinya dominan ditemukan pada endometrium yang mengindikasikan berperan penting dalam mendukung konsepsi selama masa kebuntingan. Ekspresi dominan gen UTMP dalam jaringan reproduksi konsisten dengan peran penting dari gen ini dalam keberhasilan reproduksi dan terbukti berpengaruh nyata dengan usia hidup produktif pada sapi perah (Khatib et al. 2009).
Frekuensi Alel Gen UTMP
Hasil analisis frekuensi alel gen UTMP pada sapi bali baik di BPTU maupun BIBD menunjukkan bahwa ditemukan 1 macam genotipe AA dan alel A
Keterangan: M = marker DNA 100 bp,1-6 = sampel sapi bali
(A) Hasil PCR-RFLP gen UTMP|BsrI (B) Penentuan pita gen UTMP Gambar 3 Penentuan genotip: (A) Hasil PCR-RFLP gen UTMP|BsrI, (B)
8
(1.00) dan tidak ditemukan alel G(0.00). Hasil penelitian menunjukkan bahwa gen UTMP|BsrI pada sapi bali bersifat monomorfik. Berikut tabel perhitungan frekuensi alel sapi bali di BPTU dan BIBD.
Tabel 1 Frekuensi alel gen UTMP pada sapi Bali
Bangsa Jumlah sampel Frekuensi Alel Sumber
A G
Sapi Bali (BPTU) 100 1.00 0.00 Hasil penelitian
Sapi bali (BIBD) 23 1.00 0.00 Hasil penelitian
Sapi Holstein (CDDR) 1363 0.28 0.72 Khatib et al. 2007
Sapi Holstein (UW) 913 0.36 0.64 Khatib et al. 2007
Nei (1987) menyatakan bahwa suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama dengan atau kurang dari 0.99 (99%). Frekuesi alel merupakan frekuensi relatif dari suatu alel dalam populasi (Nei dan Kumar 2000). Selain frekuensi alel, keragaman genetik juga digunakan untuk melihat nilai heterozigositas. Pendugaan nilai heterozigositas penting diketahui untuk mendapatkan informasi mengenai tingkat polimorfisme suatu alel (Falconer dan Macay 1996). Heterozigositas digunakan dalam mengukur keragaman pada suatu populasi. Allendorf dan Luikart (2006) menjelaskan bahwa nilai heteozigositas dipengaruhi oleh frekuensi alel dan jumlah alel serta jumlah sampel yang digunakan.
Analisis sekuens gen UTMP
Hasil dari analisis sekuens gen UTMP yang dipotong menggunakan enzim
BsrI terhadap sekuens gen UTMP atau SERPINA14 Bos Taurus memiliki kesamaan pada GenBank (kode akses GenBank L22095) dengan tingkat kesamaan 98%. Gen UTMP yang diamati terdapat fragmen lain yang menyebabkan insersi nukleotida pola minisatelit dengan motif 38 ulangan pada sekuens gen UTMP pada exon 4 sapi bali di BPTU dan BIBD pulau Bali. Berikut sekuen gen UTMP yang mengalami insersi pola minisatelit yang dijelaskan pada gambar 4.
9
Insersi menyebabkan perubahan panjang sekuens gen UTMP menghasilkan pita yang panjangnya 406 bp. Insersi ini disebabkan karena adanya penambahan basa yang berulang-ulang pada sekuen DNA gen UTMP yang diteliti. Pada gen UTMP yang diteliti sapi bali BPTU dan BIBD tidak ditemukan adanya mutasi melainkan terdapat insersi basa nitrogen. Insersi yang terjadi pada gen UTMP lokasi exon 4 ini akan berpengaruh terhadap ekspresi dan fungsi gen untuk itu perlu dilakukannya identifikasi fenotipik terkait insersi ini terhadap sapi bali. Khatib et al. (2007) melaporkan bahwa single nukleotida polimorfisme
(SNP) gen UTMP A>G yang terdapat pada posisi 1 296 menunjukkan hubungan yang signifikan dengan masa usia produktif (productive life). SNP ini bisa dimanfaatkan untuk seleksi dengan bantuan penanda genetik untuk peningkatan PL (usia hidup produktif) dalam inti pemuliaan. Berikut rekonstruksi gen UTMP yang dijelaskan pada gambar 5.
Berdasarkan hasil sekuensing panjang fragmen gen UTMP yaitu 406 bp. Hal ini karena populasi sapi Bali yang diamati mengalami insersi sepanjang 77 basa pola minisatelit dengan motif pengulangan 38 basa yaitu 5’-CCCAATGAAGGCAAAGGAGGTCCCGGCGGTCGTGAAAGT-3’ sebanyak 4 kali ulangan yang tidak sempurna. Pengertian lokus minisatelit atau Variable Number Tandem Repeat (VNTR) adalah untaian DNA yang berisi sekuens berulang yang panjangnya sekitar 20 hingga 100 pasang basa. Penyebab dari variasi ini adalah unit pengulangan yang berbeda pada alel suatu individu. Sekuens yang menyerupai satellite ini terdiri dari pasangan yang berulang dari unit yang pendek yaitu 5-50 ulangan. Fenomena ini ditemukan secara kebetulan sebagai fragmen yang ukurannya sangat bervariasi. Polimorfik VNTR banyak digunakan sebagai penanda genetik dalam genetika populasi, pemetaan gen, dan kedokteran forensik (Jeffreys et al. 1985). Posisi penempelan primer dan insersi pola minisatelit pada exon 4 pada sekuen gen UTMP disajikan pada Gambar 4.
10
SIMPULAN DAN SARAN
Simpulan
Gen UTMP|BsrI exon 4 pada sapi bali di BPTU dan BIBD diperoleh 1 genotipe AA dan alel A. Sapi bali yang terdapat di BPTU dan BIBD pada gen UTMP bersifat monomorfik. Sekuens gen UTMP sapi bali yang diamati tidak ditemukan adanya mutasi tetapi ditemukan adanya insersi dengan pola minisatelit.
Saran
11
DAFTAR PUSTAKA
Altschul SF, Gish W, Miller W, Mayers EW, Lipman DJ. 1990. Basic local alignment search tool. J. Mol. Biol. 215:403-410.
Allendorf FW, Luikart G. 2007. Conservation and the Genetics of Populations. USA (US): Blackwell Publishing.
Bugiwati SRA. 2007. Pertumbuhan dimensi tubuh pedet jantan sapi bali di Kabupaten Bone dan Barru Sulawesi Selatan. J. Sains dan Tekno. 7:103– 108.
[Ditjennak] Direktorat Jenderal Peteurnakan. 2013. Statistik Peternakan 2013. [terhubung berkala]. http://ditjennak.deptan.go.id. [28 oktober 2013].
Direktorat Bina Pembibitan. 2000. Kebijakan Pembibitan dalam Swasembada Daging 2005. Panduan Seminar Nasional. Departemen Pertanian, Direktorat Jendral Peternakan.
Falconer DS, Mackay TFC. 1996. Introduction to Quantitative Genetics. Ed ke-4. Longman(UK): Essex.
Ing NH, Francis H, Donnels J, Amann J, Roberts M. 1989. Progesteronen induction of the uterine milk protein : Major secretory proteins of Sheep Endometrium. Biology of Reproduction. 41, 643-654.
Jeffreys, AJ, Wilson E, Thein SL. 1985. ‘Hypervariable minisatellite’ regions in human DNA. Nature. Vol 314: 67-73.
Khatib H, Schutzkuz V, Chang YM, Rosa GJM. 2007. Pattern of expression of uterine milk protein gene and its association with productive life in dairy cattle. J. Dairy Sci.90: 2427-2433. doi:10.3168/jds.2006-722
Khatib H, Huang W, Wang X, Train AH, Bindrim AB, Schutzkus V, Monson RL. 2009. Single gene and gene interaction effects on fertilization and embryonic survival rates in cattle. J. Dairy Sci. 92: 2238-2247.
doi:10.3168/jds.2008-1767.
Moioli B, Napolitano F, Catillo G. 2004. Genetic diversity between Piedmontese, Maremmana and Podolica Cattle Breeds. Journal of Heredity. Vol. 95, no. 3, p. 250-256
Muladno. 2002. Seputar Teknologi Rekayasa Genetika. Bogor (ID): Pustaka Wirausaha Muda dan USESE Foundation.
Nei M. 1987. Molecular Evolutionery Genetics. New York (US) : Columbia University Pr.
Nei M, Kumar S. 2000. Molecular Evolution and Phylogenetics. New York (US): Oxford University Pr.
Nijman IJ, Otsen M, Verkaar ELC, Ruijter C, Hanekamp E, Ochieng JW,
Shamshad S, RegeJEO, Hanotte O, Barwegen MW et al. 2003.
Hybridization of banteng (Bos javanicus) and zebu (Bos indicus) revealed by mitochondrial DNA, satellite DNA, AFLP and microsatellites. Nature.
90 : 10-16.
Orita M, Iwahana H, Kanazawa H, Hayashi K, Sekiya T. 1989. Detection of polymorphism of human DNA by gel electrophoresis as single-strand conformation polymorphism. Proc Natl Acad Sci. 86:2766-2770.
12
Siswanto M, Patmawati NW, Trinayani NN, Wandia IN, Puja IK. 2013. Penampilan reproduksi sapi bali pada peternakan intensif di Instalasi pembibitan Pulukan. Jurnal Ilmu dan Kesehatan Hewan 1(1): 11-15.
Sumantri C, Anggraeni A, Farajallah A, Perwitasari D. 2007. Keragaman mikrosatelit DNA sapi perah FH di balai pembibitan ternak unggul Baturraden. Jurnal Ilmu Ternak dan Veteriner. 12 (2): 124-133.
Talib C. 2002. Sapi bali di daerah sumber bibit dan peluang pengembangannya.
Wartazoa 12(3): 100-107.
Tekin S, Padua MB, Newton GR, Hansen PJ. 2005. Identification and cloning of Caprine Uterine Serpin. JMol. Reprod. Dev. 70: 262–270. Wiley-Liss, Inc. Tolihere MR. 2002. Increasing the success rate and adoption of artificial
insemination for genetic improvement of Bali cattle. Working Papers: Bali Cattle Workshop. Bali, 4-7 February 2002.
Ulbrich SE, Thomas F, Katy S, Eva E, Nadine W, George JA, Horst R, Myriam R, Eckhard W, Heinrich M et al. 2009. Evidence for estrus-dependent uterine serpine (SERPINA 14) expression during estrus in the bovine endometrial glandular epithelium and lumen. J Biology of Reproduction. 81:795-805. Weir BS. 1996. Genetic Data Analysis : Method for Discrete Population Genetic
Data. Ed ke-2. Sunderland, MA (US): Sinauer Associates Inc.
Williamson G, Payne WJA, 1993. Pengantar Peternakan di Daerah
Tropis.Yogyakarta (ID): Universitas Gajah Mada Pr.
Winter WH. 2003. Cattle production in eastern Indonesia. A Summary of collaborative research. Bogor (ID): Prosiding Seminar Nasional Teknologi Peternakan dan Veteriner. Bogor: 28-29 September 2003.
13
LAMPIRAN
Lampiran 1 Sekuens gen uterine milk protein yang diakses di GenBank dengan kode akses L22095.1
LOCUS BOVSPIS 1463 bp mRNA linear MAM 26-JUN-1997 DEFINITION Bos taurus uterine milk protein precursor, mRNA, complete cds. ACCESSION L22095
VERSION L22095.1 GI:438480
KEYWORDS serine proteinase inhibitor; serpin; uterine milk protein. SOURCE Bos taurus (cattle)
Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Laurasiatheria; Cetartiodactyla; Ruminantia; Pecora; Bovidae; Bovinae; Bos.
REFERENCE 1 (bases 1 to 1463)
AUTHORS Mathialagan,N. and Hansen,T.R.
TITLE Pepsin-inhibitory activity of the uterine serpins
JOURNAL Proc. Natl. Acad. Sci. U.S.A. 93 (24), 13653-13658 (1996)
REFERENCE 2 (bases 1 to 1463)
AUTHORS Mathialagan,N., Hansen,T. and Roberts,M.R. TITLE Direct Submission
JOURNAL Submitted (03-JAN-1994) Animal Sciences, University of Missouri, 158 ASRC, East Campus Drive, Columbia, MO 65211, USA
14
541 aagaagacca tcagccacca tgtggctgaa aaaacacata cgaaaatcac aaacttaatc 601 accgacctga accctgagac catcctgtgt cttgttaacc acattttctt caaaggcatc 661 ttgaaaagag cttttcagcc caaactcacc cagaaggagg tcttctttgt gaatgaccaa 721 accaaagtgc aggtggacat gatgagaaag acagaacgga tgctttacag ccggtcagag 781 gagctacatg ctacgatggt taagatgcct tgcaaaggaa atgtgtccct aactctcatg 841 cttccagatg ccggacaatt tgacactgat cttaaaaaga tgactgctaa gcgagctaaa 901 cttcagaaaa tcagtgactt cagactggtg cgcttaattt tgcccaagtt gaagatctcc 961 ttcaagataa actttaagca tctgcttccc aagattgacc ccaaacatat actgactgcc 1021 acagcaatct cacaggccat cacatcgaag gctcccctgc ctaatttgga ggccctacat 1081 caagctgaga tagagctgag cgagcacgcc ttaaccgtgg acacagccat tcacacagat 1141 aatctgttga aagtcccagt gaaggcaaag gaggtcccgg cggtcgtgaa agtcccaatg 1201 aaggcaaagg aggtcccggc ggtcgtgaaa gtcccaatga acacaaagga ggtcccagtg 1261 gtcgtgaaag tcccaatgaa cacaaaggag gtcccagtgg tcgtgaaggt caacagaccc 1321 ttcttgctgt ttgtggagga tgagaagact caaagagacc tctttgtggg caaagtcctc 1381 aacccccaag ttgagtagag ccagggccac actgtgcagc acaggaactt agcaggccat 1441 gaataaaaag agtacaattc acc
//
Lampiran 2 Hasil BLAST nukleotida sekuens gen UTMP sapi bali dengan sekuens GenBank kode akses L22095.1
15
RIWAYAT HIDUP
Penulis dilahirkan di daerah Jombang pada tanggal 11 Desember 1990. Penulis merupakan anak ketiga dari 5 bersaudara, dari pasangan Bapak Agus Sucipto dan Ibu Sri Endang Susiani.