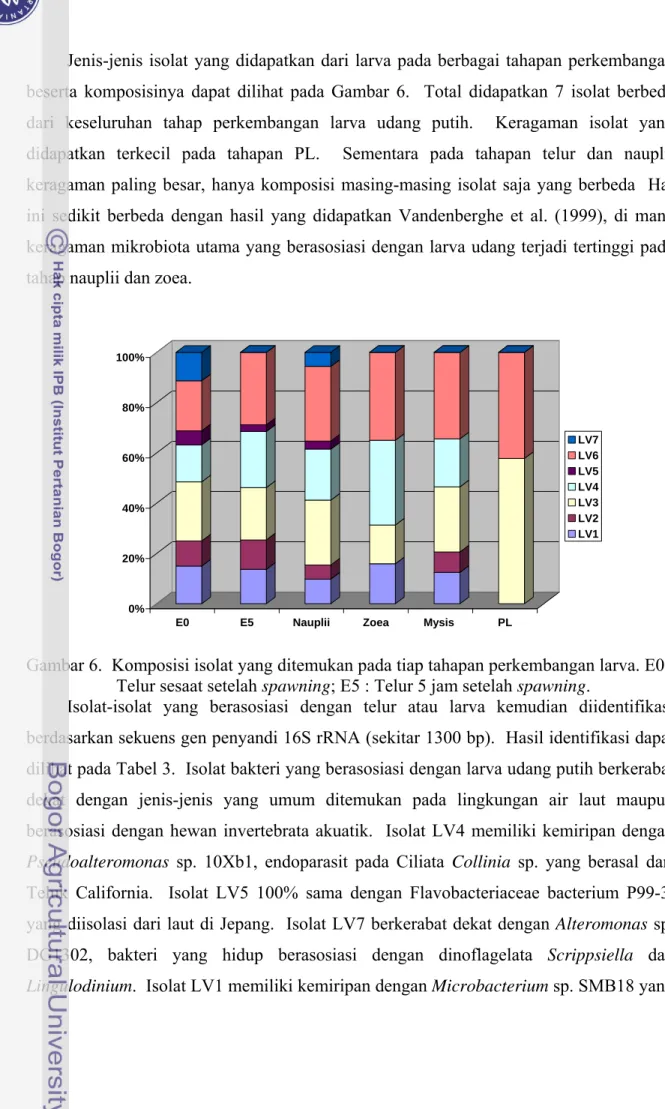
ANALISIS KOMUNITAS BAKTERI
SELAMA TAHAPAN PERKEMBANGAN LARVA
UDANG PUTIH (Litopenaeus vannamei)
ARTINI PANGASTUTI
SEKOLAH PASCA SARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2008
PERNYATAAN MENGENAI DISERTASI DAN SUMBER INFORMASI
Dengan ini saya menyatakan bahwa disertasi berjudul ’Analisis Komunitas
Bakteri Selama Tahapan Perkembangan Larva Udang Putih (Litopenaeus
vannamei)’ adalah karya saya sendiri dan belum diajukan dalam bentuk apapun kepada
perguruan tinggi dimanapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka dalam disertasi ini.
Bogor, November 2008
Artini Pangastuti G361050041
RINGKASAN
Udang putih (Litopenaeus vannamei) merupakan salah satu komoditas ekspor andalan Indonesia. Dalam budidaya udang, komunitas bakteri yang berasosiasi dengan udang telah diketahui memiliki peran penting bagi kelangsungan hidup inangnya. Selama ini, penelitian mengenai komunitas bakteri yang berasosiasi dengan udang masih menggunakan metode kultur. Dalam penelitian ini, metode berbasis molekular, yaitu
Terminal Restriction Fragment Length Polymorphism (T-RFLP) dan Amplified Ribosomal DNA Restriction Analysis (ARDRA) digunakan untuk memonitor keragaman
bakteri yang berasosiasi dengan tahap-tahap perkembangan larva, struktur komunitasnya selama perkembangan larva, serta pembentukan komunitas bakteri ini.
. Sampel untuk analisis diperoleh dari satu siklus produksi di hatchery berupa telur, nauplii, zoea, mysis, dan post larva. T-RFLP dan ARDRA menggunakan marker yang sama, yaitu gen 16S rRNA, tetapi untuk T-RFLP amplifikasi dilakukan dengan primer
forward yang dilabel pada ujung 5’ sedangkan untuk ARDRA menggunakan primer yang
tidak dilabel. Baik T-RFLP maupun ARDRA menunjukkan bahwa tiap tahapan perkembangan larva memiliki pola sidik ragam komunitas bakteri yang unik. Komunitas bakteri berubah secara dinamis mengikuti perkembangan larva, kecuali pada tahapan yang lebih akhir, di mana sistem imunitas dan pencernaan larva sudah berkembang lebih sempurna. Proses molting menyebabkan komunitas bakteri selalu berubah karena lapisan kitin saluran pencernaan ikut mengelupas dan lapisan yang baru bebas dari bakteri. Berdasarkan analisis keragaman, komunitas bakteri pada mysis atau post larva memiliki tingkat keragaman dan distribusi jenis yang paling tinggi dan merata, sementara komunitas bakteri pada zoea adalah yang terendah keragamannya dan tidak merata. Filotipe yang dominan pada masing-masing tahapan berbeda. Demikian juga terdapat perbedaan antara komunitas bakteri yang berasosiasi dengan larva dan komunitas bakteri di air pemeliharaannya. Jenis yang dominan pada air pemeliharaannya bukan merupakan jenis yang dominan berasosiasi dengan larva. Akan tetapi, beberapa filotipe dapat ditemukan pada keduanya, dan secara bersama-sama filotipe-filotipe ini dalam komunitas memiliki kemelimpahan relatif yang besar dan dominan. Selain air, kemungkinan pakan juga mempengaruhi komposisi komunitas bakteri yang berasosiasi dengan larva.
T-RFLP dan ARDRA dapat menggambarkan keragaman dalam komunitas bakteri secara lebih baik dibandingkan teknik kultur, tetapi ARDRA menghasilkan pengukuran tingkat keragaman yang lebih tinggi dibandingkan analisis T-RFLP. Dengan teknik kultur didapatkan 7 isolat yang termasuk dalam 3 kelompok, yaitu Proteobacteria, Bacteroidetes, dan Firmicutes. Tidak satupun dari isolat-isolat ini yang muncul dalam hasil T-RFLP kecuali ada beberapa genus yang sama dalam jumlah minoritas. Kemungkinan sebagian besar bakteri yang berasosiasi dengan larva udang putih tidak dapat dikultur. Identifikasi TRF (Terminal Restriction Fragment) yang dominan untuk tiap tahapan adalah: Proteobacteria untuk telur, Bacteroidetes untuk nauplii, Cyanobacteria untuk zoea, dan Proteobacteria untuk mysis dan post larvae. Tipe ARDRA yang dominan diidentifikasi berdasarkan sekuens gen 16S rRNA klon. Proteobacteria merupakan kelompok yang dominan dalam komunitas bakteri hampir di seluruh tahapan, tetapi terdapat perbedaan dengan hasil T-RFLP. Sementara itu tipe yang dominan pada tahap zoea memiliki kekerabatan paling dekat dengan suatu klon Chloroflexi. Semua sekuens hanya memiliki tingkat kesamaan ≤96% dengan kerabat terdekatnya di database, menunjukkan bahwa sebagian besar bakteri yang berasosiasi dengan larva udang putih adalah spesies yang baru. Kerabat terdekat dari sekuens-sekuens tersebut sebagian besar merupakan jenis bakteri yang tidak dapat dikultur, menguatkan dugaan bahwa bakteri-bakteri dalam komunitas itu mayoritas tidak dapat dikultur. Dengan demikian, teknik yang berbasis kultur tidak dapat diandalkan untuk mengungkap keragaman pada komunitas bakteri ini. Teknik berbasis molekular lebih baik untuk diterapkan dalam monitoring komunitas bakteri sepanjang proses produksi benur di hatchery.
Analisis T-RFLP dapat digunakan untuk memonitor perubahan sidik ragam komunitas bakteri akibat suatu perlakuan. Dengan teknik ini dapat ditunjukkan bahwa perlakuan perendaman dengan Povidone iodine pada telur dan nauplii dapat menurunkan kandungan total bakteri. Akan tetapi efeknya hanya bersifat sementara, jumlah bakteri kembali pulih pada tahapan lebih lanjut. Povidone iodine memiliki efek yang berbeda pada berbagai kelompok bakteri yang berasosiasi dengan larva udang putih. Oleh karena itu, perlakuan ini juga dapat mengubah profil komunitas bakteri yang berasosiasi dengan larva udang putih serta menurunkan tingkat keragamannya. Kemungkinan perlakuan ini
dapat berbalik menimbulkan efek negatif jika menyebabkan bakteri yang menguntungkan tertekan pertumbuhannya dan bakteri patogen menjadi lebih dominan. Untuk itu penggunaan disinfektan untuk mengendalikan jumlah kandungan bakteri perlu dipertimbangkan kembali dari segi keuntungan maupun kerugiannya dalam rangka perbaikan produksi akuakultur.
© Hak Cipta milik IPB, tahun 2008 Hak Cipta dilindungi Undang-Undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber.
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah
b. Pengutipan tidak merugikan kepentingan yang wajar IPB.
2. Dilarang mengumumkan atau memperbanyak sebagian atau seluruh karya tulis dalam bentuk apa pun tanpa izin IPB
ABSTRACT
ARTINI PANGASTUTI. Analyses of bacterial communities during developmental stages of white shrimp (Litopenaeus vannamei) larvae. Supervised by ANTONIUS SUWANTO (Major Advisor), YULIN LESTARI, MAGGY THENAWIDJAYA SUHARTONO (Co advisor)
Terminal Restriction Fragment Length Polymorphism (T-RFLP) and Amplified Ribosomal DNA Restriction Analysis (ARDRA) were used to monitor the population dynamics of bacterial communities associated with different developmental stages of white shrimp (Litopenaeus vannamei) larvae. Samples were collected from egg, nauplii, zoea, mysis, and post larvae stages in one cycle of production at hatchery. Either T-RFLP or ARDRA indicated that every stage of shrimp development had unique fingerprint pattern of microbial community. Diversity analyses showed that bacterial community of mysis or post larvae had the highest diversity and evenness, meanwhile bacterial community of zoea had the lowest diversity. There were differences between bacterial community of larvae and its rearing water for all stages, but some phylotypes were found either in larvae body or rearing water. Identification of most abundant TRF for each stage resulted in Proteobacteria for egg, Bacteroidetes for nauplii, Cyanobacteria for zoea, and Proteobacteria for mysis and post larvae. Bacterial community richness were higher using molecular-based techniques than culture, but ARDRA resulted in more diversity and evenness than T-RFLP analyses for almost all stages of larvae development. Dominant ARDRA types were identified based on the sequence of 16S rRNA genes. Proteobacteria was dominant group in bacterial community for all stages except zoea. All sequences only had ≤96% similarity with closest relatives in database, suggesting that most bacteria associated with white shrimp larvae were novel species and might be unculturable. As shown by T-RFLP analysis, Povidone iodine treatment for eggs and nauplii caused temporal decrease in total bacterial load. The treatment also affected diversity and changed the profile of bacterial communities associated with white shrimp larvae throughout developmental stages.
ANALISIS KOMUNITAS BAKTERI
SELAMA TAHAPAN PERKEMBANGAN LARVA
UDANG PUTIH (Litopenaeus vannamei)
ARTINI PANGASTUTI
Disertasi
Sebagai salah satu syarat memperoleh gelar Doktor pada
Program Studi Biologi
SEKOLAH PASCA SARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2008
PRAKATA
Puji syukur penulis panjatkan kehadirat Allah SWT atas berkah dan karuniaNya sehingga penulis dapat menyelesaikan penulisan disertasi dengan judul : Analisis Komunitas Bakteri Selama Tahapan Perkembangan Larva Udang Putih (Litopenaeus
vannamei).
Penelitian ini tidak akan terlaksana tanpa bantuan dari banyak pihak. Untuk itu penulis mengucapkan terima kasih kepada Prof. Dr. Antonius Suwanto, Dr. Ir. Yulin Lestari, serta Prof. Dr. Maggy T. Suhartono atas bimbingan dan arahan sebelum dan selama pelaksanaan pelitian serta penulisan naskah disertasi ini. Sungguh banyak masukan dan ilmu yang penulis dapatkan dari para pembimbing yang sangat berguna dalam kelanjutan karir penulis setelah ini. Sungguh suatu kehormatan bagi penulis untuk dapat bekerja sama dengan para pembimbing. Terima kasih juga kepada Dr. Aris Tri Wahyudi, Dr. Widanarni, dan Dr. Akhmaloka atas kesediaannya menguji dan memberi masukan bagi disertasi ini.
Penulis juga mengucapkan terima kasih pada PT. Charoen Phokphand Indonesia atas dana dan fasilitas untuk penelitian yang telah diberikan, juga pada Marine Research Center PT. Central Pertiwi Bahari untuk fasilitas pemeliharaan larva udang. Terima kasih kepada seluruh staf peneliti dari R &D PT. CPI dan MRC PT. CPB atas segala bantuannya selama penulis melaksanakan penelitian ini. Untuk analisis bioinformatika, penulis mengucapkan terima kasih kepada Yepy Hardy Rustam dari PT. CPI atas bantuannya.
Ucapan terima kasih juga penulis sampaikan kepada Dirjen DIKTI Depdiknas atas biaya studi S3 di Institut Pertanian Bogor melalui BPPS.
Terakhir, penulis mengucapkan terima kasih yang sebesar-besarnya untuk suami, anak-anak, dan segenap keluarga atas dukungan dan kesempatan yang selalu diberikan pada penulis selama menempuh pendidikan dalam 3 tahun ini.
Bogor, November 2008 ARTINI PANGASTUTI
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 31 Mei 1975 dari ayah Hadi Subroto dan ibu Budiarti Wirasmani. Pendidikan dasar dan menengah penulis tamatkan di Surakarta, yaitu SDN Cemara Dua tahun 1987, SMPN I tahun 1990, dan SMAN I tahun 1993. Tahun 1993 penulis diterima di Institut Pertanian Bogor melalui jalur USMI dan diterima di Jurusan Biologi FMIPA IPB tahun berikutnya. Gelar Sarjana Sains penulis dapatkan pada tahun 1998 dan langsung bekerja di FMIPA Universitas Sebelas Maret Surakarta. Pada tahun yang sama, melalui program DUE dari DIKTI, penulis melanjutkan studi S2 di Institut Pertanian Bogor dengan mengambil spesialisasi mikrobiologi. Gelar Magister Sains diterima pada tahun 2001. Kemudian pada 2005 penulis kembali ke Institut Pertanian Bogor untuk menempuh studi S3 dengan sponsor BPPS.
Setelah diterima pada tahun 1998 di FMIPA UNS, penulis diangkat sebagai Pegawai Negeri Sipil pada tahun 2000 dan menjabat sebagai Asisten Ahli tahun 2002. Berturut-turut pada tahun 2004 dan 2005 penulis mendapatkan dana Penelitian Dosen Muda dari Dikti.
DAFTAR ISI Halaman DAFTAR TABEL... v DAFTAR GAMBAR... vi PENDAHULUAN... 1 TINJAUAN PUSTAKA... 4
Produksi dan Ekspor Udang Indonesia... 4
Perkembangan dan Siklus Hidup Udang Putih (Litopenaeus vannamei)... 5 Komunitas Bakteri pada Hewan Akuatik... 8
Pembentukan dan Dinamika Komunitas Bakteri pada Hewan Akuatik 9 Keragaman Komunitas Bakteri yang Berasosiasi dengan Hewan Akuatik... 11
Pengaruh Pakan pada Komposisi Mikrobiota... 13
Interaksi Mikrobiota dengan Inang... 16
Eksplorasi Komunitas Bakteri Berbasis Molekular... 19
BAHAN DAN METODE... 23
Tempat dan Waktu Penelitian... 23
Pengambilan dan Penyiapan Sampel... 23
Ekstraksi DNA... 24
Amplifikasi Gen 16S rRNA untuk T-RFLP... 24
Digesti Amplikon... 25
Analisis T-RFLP... 25
Penghitungan Jumlah Bakteri dan Isolasi... 25
Amplifikasi gen 16S rRNA untuk ARDRA... 26
Kloning Produk PCR 16S rRNA... 26
ARDRA... 26
Sekuensing Gen Penyandi 16S rRNA... 27
Analisis Keragaman... 27
Diagram Alur Pekerjaan... 28
HASIL DAN PEMBAHASAN... 29
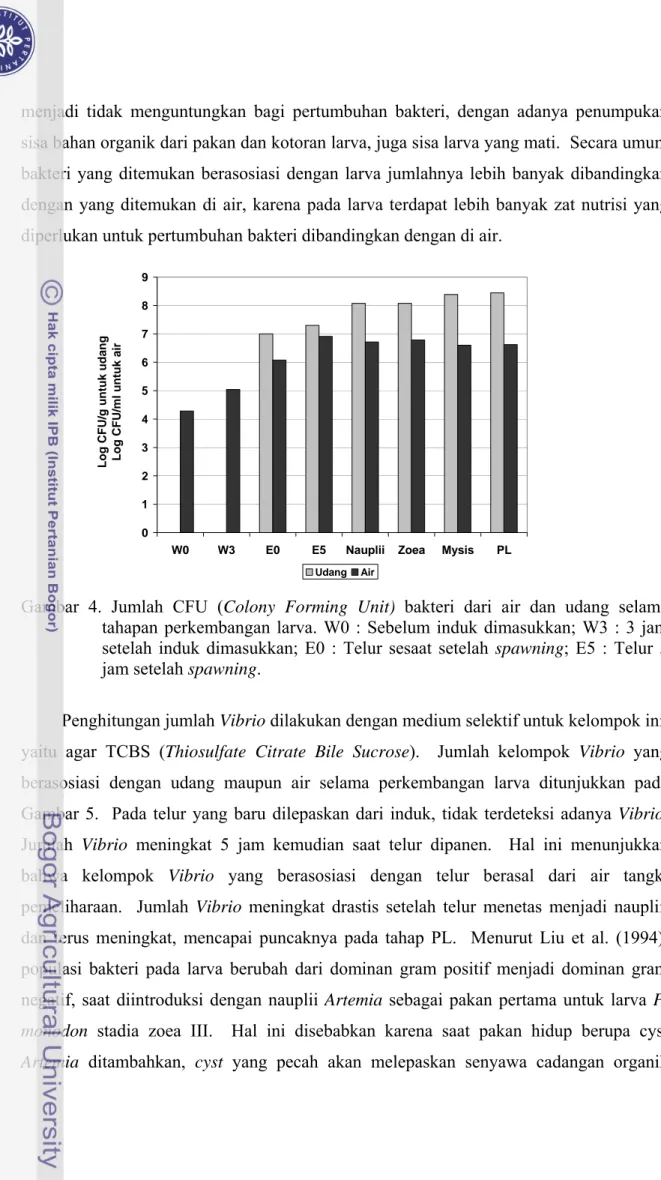
Penghitungan Jumlah dan Isolasi Bakteri yang Dapat Dikultur... 29
Analisis Komunitas Bakteri dengan T-RFLP... 35
Pembentukan Komunitas Bakteri yang Berasosiasi Dengan Larva... 44
Amplified Ribosomal DNA Restriction Analysis (ARDRA)………… 50
Perbandingan Teknik Berbasis Kultur dan Molekular... 56
KESIMPULAN DAN SARAN... 71
Kesimpulan ... 71
Saran ... 71
DAFTAR PUSTAKA... 73
DAFTAR TABEL
Halaman 1. Produksi udang Indonesia menurut spesies (dalam Ton)... 5 2. Tahapan perkembangan larva udang putih... 7 3. Hasil identifikasi isolat yang berasosiasi dengan telur/larva... 32 4. Perbandingan jumlah relatif (%) kelompok bakteri yang ditemukan pada
tahapan perkembangan larva... 38 5. Filotipe yang berasosiasi dengan 3 atau lebih tahapan perkembangan larva
udang putih... 39 6. Filotipe unik yang ditemukan pada tahapan perkembangan larva tertentu... 41 7. Keragaman bakteri pada tiap tahap perkembangan larva... 42 8. Filotipe yang kemungkinan berasal dari induk... 49 9. Keragaman komunitas pada tiap tahapan perkembangan larva dengan
ARDRA... 52 10. Hasil analisis sekuens dari klon yang dominan pada tiap tahapan... 54 11. Perbandingan keragaman yang terukur dengan teknik T-RFLP dan
ARDRA... 59 12. Perbandingan kelompok yang dominan pada tahap-tahap perkembangan
larva dengan menggunakan teknik berbeda... 60 13. Keragaman komunitas bakteri yang berasosiasi dengan tahapan
perkembangan larva udang putih tanpa dan dengan perlakuan iodine…….. 67 14. Filotipe yang sensitif terhadap iodine………. 68 15. Filotipe yang resisten terhadap iodine... 69
DAFTAR GAMBAR
Halaman 1. Siklus hidup Udang Putih (Litopenaeus vannamei)... 6 2. Prosedur dalam analisis T-RFLP... 21 3. Skema pengambilan sampel dalam satu siklus produksi benur di hatchery... 24 4. Jumlah CFU (Colony Forming Unit) bakteri dari air dan udang... 30 5. Jumlah CFU (Colony Forming Unit) untuk kelompok Vibrio dari air dan
udang…...………... 31 6. Komposisi isolat yang ditemukan pada tiap tahapan perkembangan larva.... 32 7. Komposisi TRF Sau3A1 selama tahapan perkembangan larva... 37 8. Perkiraan jumlah bakteri berdasarkan luas Peak Area T-RFLP... 43 9. Perbandingan komposisi TRF Sau3A1 pada udang dengan air
pemeliharaan selama tahapan perkembangan larva... 46 10. Grafik hubungan antara jumlah klon dan jumlah tipe yang ditemukan... 51 11. Hubungan kekerabatan klon yang dominan pada tiap tahapan
perkembangan larva udang putih dengan beberapa bakteri yang memiliki kedekatan kekerabatan berdasar hasil analisis sekuensing... 55 12. Jumlah total bakteri pada udang tanpa dan dengan perlakuan iodine... 62 13. Jumlah total Vibrio pada udang dengan dan tanpa perlakuan iodine... 63 14. Komposisi komunitas bakteri yang berasosiasi dengan udang tanpa dan
dengan perlakuan iodine... 64 16. Komposisi Vibrio yang berasosiasi dengan udang tanpa dan dengan
perlakuan iodine... 65 17. Perkiraan jumlah total bakteri tanpa dan dengan perlakuan iodine
berdasarkan total peak area TRF... 66 18. Perbandingan komposisi TRF Sau3A1 selama tahapan perkembangan
DAFTAR LAMPIRAN
Halaman 1. TRF hasil pemotongan dengan enzim Sau3A1 yang ditemukan pada
tahap-tahap perkembangan larva udang putih... 82 2. Elektroforegram analisis T-RFLP komunitas bakteri pada udang dan air
PENDAHULUAN
Dengan semakin terbatasnya jumlah ikan dan produk perikanan lain yang dapat ditangkap langsung, usaha budidaya menjadi alternatif untuk memenuhi permintaan akan produk perikanan yang semakin meningkat. Dewasa ini sebagian besar usaha budidaya udang terpusat di dua wilayah, yaitu Asia dan Amerika Latin. Indonesia merupakan salah satu negara produsen terbesar, berada pada urutan ke-6 di tahun 2004. Pada awalnya, jenis udang yang dibudidayakan didominasi oleh udang windu (Penaeus
monodon) dan P. margueinsis. Akan tetapi, produksi udang putih (Litopenaeus vannamei) semakin meningkat dan mulai menggantikan jenis yang lain. Menurut FAO
(2006), produksi L. vannamei meningkat sebesar 120% antara tahun 2002-2003.
Permintaan yang meningkat pesat untuk komoditas ini telah memaksa petani untuk mengintensifkan usaha budidaya untuk meningkatkan produksi. Walaupun dapat meningkatkan produksi, usaha budidaya intensif telah menyebabkan kerugian ekonomi yang cukup signifikan bagi para petani akhir-akhir ini karena masalah penyakit. Kondisi budidaya yang intensif, seperti padat tebar tinggi, akumulasi bahan organik sisa pakan yang berlebih serta sisa udang yang mati, serta penggunaan senyawa anti mikroba yang berlebih, menyebabkan timbulnya penyakit yang berakibat kematian massal udang. Di Asia, kematian massal pada udang yang dibudidayakan akibat White Spot Syndrome
Virus (WSSV) dan Yellow Head Virus (YHV) telah menyebabkan kerugian sekitar $1
milyar per tahun sejak tahun 1994 (Lightner et al. 1994). Di Indonesia sendiri, penyakit yang berasosiasi dengan Vibrio spp. dan virus merupakan masalah utama pada budidaya udang.
Komunitas bakteri diduga memiliki pengaruh terhadap pertumbuhan dan kemampuan bertahan hidup hewan akuatik. Adanya perbaikan pertumbuhan ini diduga berkaitan dengan komunitas bakteri yang hidup berasosiasi. Peranan komunitas bakteri ini pada udang belum diketahui, tetapi diduga pengaruhnya cukup signifikan bagi kelangsungan hidup inang. Bakteri dapat membantu nutrisi inang baik secara langsung maupun tidak langsung dengan memproduksi enzim-enzim pencernaan dan senyawa esensial. Keberadaan komunitas bakteri juga dapat mencegah bakteri oportunis untuk berproliferasi dan mengkolonisasi tubuh udang, dan ini merupakan mekanisme
pertahanan tubuh yang penting pada tahap larva di mana sistem imunitas belum berkembang sempurna (Vanbelle et al. 1990).
Sampai saat ini, beberapa penelitian mengenai komunitas bakteri yang berasosiasi dengan udang telah dilakukan untuk berbagai tujuan, sebagian besar adalah untuk mendapatkan galur yang potensial untuk probiotik dan galur yang berasosiasi dengan suatu penyakit (Colorni 1985, Hameed 1993, Straub & Dixon 1993, Vandenberghe et al. 1999, Makridis et al. 2000). Akan tetapi seluruh studi di atas melakukannya dengan metode kultur sehingga hanya jenis-jenis bakteri yang dapat dikulturkan yang terwakili. Padahal, sebagian besar bakteri yang hidup di lingkungan adalah bakteri yang tidak dapat ditumbuhkan pada medium artifisial di laboratorium (Amann et al. 1995). Dari bakteri keseluruhan bakteri yang hidup berasosiasi dengan hewan akuatik yang teramati dengan mikroskop, hanya sebagian kecil yang dapat dikulturkan (Van Elsas & Van Overbeek 1993, Wagner et al. 1994). Oleh karena itu, metode kultur tidak dapat menggambarkan keanekaragaman yang sebenarnya dari komunitas bakteri.
Masalah tersebut dapat diatasi dengan menganalisis DNA atau RNA yang diisolasi langsung dari contoh lingkungan. Beberapa teknik dapat dilakukan, antara lain yang menggunakan gen penyandi 16S rRNA sebagai penanda molekuler seperti ARDRA (Amplified Ribosomal DNA Restriction Analysis), DGGE (Denaturing Gradient Gel
Electrophoresis), dan T-RFLP (Terminal Restriction Fragment Length Polymorphism).
Teknik T-RFLP merupakan salah satu pendekatan molekuler terkini yang dapat menduga adanya perbedaan genetik yang rumit antar galur dan memberikan pengertian yang mendalam tentang struktur dan fungsi suatu komunitas mikroba. Pada teknik ini yang diukur adalah polimorfisme ukuran dari fragmen hasil pemotongan enzim restriksi pada ujung produk PCR (TRF, Terminal Restriction Fragment). T-RFLP dapat digunakan untuk melihat secara kuantitatif perubahan pada komunitas mikroba karena kemelimpahan relatif suatu populasi dalam komunitas dapat dibandingkan antar sampel dengan perlakuan yang berbeda atau diambil pada waktu yang berlainan. Teknik ini telah digunakan untuk analisis mikrobiota pada manusia (Hayashi et al. 2002, Jernberg et al 2005). Dari beberapa studi tersebut, T-RFLP terbukti merupakan alat yang baik untuk memonitor perubahan sidik jari komunitas mikrobiota sesuai waktu atau perlakuan.
Teknik ARDRA merupakan teknik yang lebih diskriminatif daripada T-RFLP karena ARDRA menganalisis seluruh bagian gen 16S rRNA yang teramplifikasi. Akan tetapi teknik ini memiliki kelemahan dalam hal konsumsi waktu dan banyaknya tahapan kerja yang harus dilakukan. Selain itu, untuk mendapatkan hasil yang konsisten diperlukan jumlah pustaka gen 16S rRNA yang cukup besar agar filotipe-filotipe dalam suatu komunitas dapat terwakili. Akibatnya biaya yang diperlukan juga lebih besar.
Penelitian ini bertujuan untuk menganalisis dinamika populasi pada komunitas bakteri yang berasosiasi dengan tahapan-tahapan perkembangan larva udang putih (L.
vannamei). Dengan demikian dapat diperoleh informasi mendalam mengenai keragaman
bakteri yang berasosiasi dengan tahap-tahap perkembangan larva, struktur komunitasnya selama perkembangan larva, dan pembentukan komunitas bakteri ini. Selanjutnya data yang diperoleh dapat menjadi basis bagi penelitian selanjutnya untuk lebih memahami interaksi antara mikroba dan inang serta pengaruh perlakuan tertentu terhadap komunitas bakteri yang berasosiasi dengan larva udang putih.
TINJAUAN PUSTAKA
Produksi dan Ekspor Udang Indonesia
Udang merupakan salah satu komoditas ekspor andalan bagi Indonesia. Usaha budidaya udang berkembang di Indonesia sekitar tahun 1964. Dimulai dari Sulawesi Selatan, budidaya udang terus berkembang, tersebar di Jawa Tengah dan Timur, Pulau Sumatera bagian utara dan selatan, Kalimantan Barat, Selatan, dan Timur serta Nusa Tenggara Barat. Sebelumnya produksi udang Indonesia berasal seluruhnya dari penangkapan langsung di alam. Dengan semakin meningkatnya usaha budidaya udang, pada tahun 2005 jumlah udang yang ditangkap langsung dari alam tinggal sekitar 40% dari total produksi udang Indonesia. Untuk seluruh dunia, pada tahun 1999, budidaya memproduksi sekitar 814250 metrik ton udang, dan ini meliputi kira-kira seperempat dari total produksi udang dunia (Rosenberry 1999). Proporsi ini meningkat pada tahun 2004 menjadi 46% (FAO 2006).
Terdapat tiga tipe usaha budidaya udang di Indonesia, yaitu tambak tradisional, semi intensif, dan intensif. Budidaya semi intensif dan intensif mencakup 25% dari total area budidaya udang, tetapi produksinya mencakup 65% total produksi udang dan 80% ekspor udang Indonesia. Produksi udang meningkat selama periode 2003-2007 sebesar 16,39% dari 192,466 ton pada 2003 menjadi 352,220 ton pada 2007. Tabel 1. menunjukkan produksi udang Indonesia menutut spesies. Dalam kurun waktu 1964-2000, terdapat dua spesies udang yang mendominasi produksi, yaitu P. monodon dan P. marguiensis. Akan tetapi sejak tahun 2005, produksi L. vannamei terus meningkat, walaupun spesies ini bukan berasal dari Indonesia. Produksi L. vannamei di Indonesia meningkat 5 kali lipat pada kurun waktu 2000-2005 (Departemen Kelautan dan Perikanan Indonesia).
Pemerintah telah menetapkan udang pada urutan ke-6 dalam komoditas ekspor non migas. Ekspor udang mengalami peningkatan selama periode 2003-2007 karena tidak adanya kuota. Tahun 2004 Indonesia mengekspor sebesar 122,9 ribu metrik ton, mengalami peningkatan sebesar 37% selama dekade 1994-2004, dengan nilai total US$840 juta. Peningkatan volume ekspor mendorong pada peningkatan nilai produksi udang, yaitu USD850,222 juta pada 2003 menjadi USD 1,048 miliar pada 2007. Nilai ekspor udang pada 2007 mencapai hampir 50% dari nilai ekspor perikanan sebesar USD
2,3 miliar. Laju pertumbuhan produksi rata-rata untuk Indonesia antara tahun 2004-2008 diperkirakan sebesar 4% per tahun. Seluruh data di atas diperoleh dari Departemen Kelautan dan Perikanan.
Pada tahun 2009, Pemerintah Indonesia telah menargetkan produksi sebesar 540.000 ton untuk P. monodon dan L. Vannamei (Departemen Kelautan dan Perikanan Indonesia). Akan tetapi, ada beberapa masalah yang dapat menghambat peningkatan produksi, diantaranya adalah penurunan kualitas benih akibat inbreeding dan kualitas induk yang tidak terstandardisasi dengan baik, degradasi lingkungan, dan pencemaran air. Selain itu, penggunaan pakan yang berlebih serta padat tebar yang tinggi, juga penggunaan antibiotik secara tidak bijaksana menyebabkan penyakit udang semakin menyebar dan menyebabkan penurunan produksi. Residu antibiotik yang digunakan petani untuk mengatasi penyakit juga menyebabkan produk udang Indonesia banyak yang tidak memenuhi standar beberapa negara yang menjadi tujuan ekspor.
Tabel 1. Produksi udang Indonesia menurut spesies (dalam Ton)
Spesies 2001 2002 2003 2004 2005 P. monodon 103.603 112.840 133.636 131.399 134.686 P. marguiensis 25.862 24.708 35.249 33.797 27.088 Metapenaeus sp. 19.093 21.634 22.881 19.929 13.731 L. vannamei - - - 53.217 103.874 Mysidopsis sp. 610 415 700 226 164 Total 149.168 159.597 192.466 238.568 279.543
Sumber : Departemen Kelautan dan Perikanan Indonesia
Perkembangan dan Siklus Hidup Udang Putih (Litopenaeus vannamei)
Udang putih tergolong dalam famili udang penaeid (Penaeidae). Udang penaeid merupakan anggota dari filum terbesar dalam kingdom binatang, yaitu Arthropoda, yang dikarakterisasi oleh jointed appendages dan eksoskleton/kutikula yang mengalami
molting secara periodik (Bailey-Brock & Moss 1992). Siklus hidup udang putih ini dapat
Gambar 1. Siklus hidup udang putih (Litopenaeus vannamei) (dengan seijin Dr. Agung Setiarto, Universitas Diponegoro Semarang)
Siklus hidup udang penaeid terdiri dari beberapa tahapan berbeda yang ditemukan pada habitat yang bervariasi. Larva mendiami permukaan laut yang kaya akan plankton. Juvenil lebih menyukai air payau di daerah pesisir, sementara udang dewasa biasanya ditemukan pada kedalaman laut dan kadar garam yang lebih tinggi. Larva udang putih melalui 3 tahapan perkembangan, yaitu nauplii, zoea, dan mysis, kemudian bermetamorfosis menjadi post larva (PL).
Saat telur menetas menjadi nauplii, larva belum mengambil makanan dari luar, hanya menghabiskan sisa cadangan makanan dari telur (egg yolk). Selanjutnya pada tahap zoea larva memakan fitoplankton dan kemudian dilanjutkan dengan zooplankton. Pada tahap mysis dan selanjutnya, udang dapat memakan berbagai organisme kecil lain seperti Artemia. Untuk berkembang menjadi dewasa, udang putih harus melalui beberapa siklus molting (Treece & Yates 1988). Molting merupakan proses pelepasan eksoskleton yang terjadi secara periodik. Jika terjadi pertumbuhan, udang harus melepaskan penghubung longgar antara epidermis dan eksoskleton kemudian melepaskan diri dari eksoskleton yang kaku tersebut. Selanjutnya udang mengambil air untuk mengembangkan eksoskleton baru yang fleksibel dan secepatnya mengeraskannya dengan mineral dan protein. Pada akhir tiap tahapan molting, air digantikan oleh jaringan dan eksoskleton mengeras. Akibatnya proses molting ini menghasilkan
juvenil dewasa telur naupli zoea mysis post larvae
penambahan ukuran badan yang diskontinyu. Beberapa tahapan perkembangan yang dilalui oleh larva udang putih ditampilkan pada Tabel 2.
Tabel 2. Tahapan perkembangan larva udang putih
Tahapan Waktu dalam tahapan
Ukuran di akhir tahapan
Telur ±14 jam (28oC) diameter ± 220 µm Nauplii (N) NI, NII,
NIII, NIV, NV
36-51 jam (28oC) Panjang: 0.43-0.58 mm, lebar: 0.18-0.22 mm Zoea I (PI) 36-48 jam (28oC) Panjang total: 1.0 mm
Panjang caudal (ekor): 0.3 mm Zoea II (PII) 36-48 jam (28oC) Panjang total: 1.28-2.01 mm
Panjang caudal (ekor): 0.72-0.87 mm Zoea III (PIII) (P3) 36-48 jam (28oC) Panjang total: 2.4- 2.59 mm
Panjang caudal (ekor): 0.93-1.40 mm Mysis I (MI) (M2) 24 jam (28oC) Panjang total: 3.5 mm
Panjang caudal (ekor): 1.2 mm Mysis II (MII) 24 jam (28oC) Panjang total: 3.3-4.2 mm
Panjang caudal (ekor): 1.2-1.4 mm Mysis III (MIII) 24 jam (28oC) Panjang total: 3.9-4.7 mm
Panjang caudal (ekor): 1.3-1.5 mm Postlarva I (PLI) 24 jam (28oC) Panjang total: 4.2-5.0 mm
Panjang caudal (ekor): 1.4-1.6 mm Sumber : Treece 1985
Komunitas Bakteri pada Hewan Akuatik
Komunitas bakteri sering ditemukan berasosiasi dengan organisme lain. Komunitas ini berkembang pada bagian tertentu dari tubuh organisme yang biasanya memiliki karakteristik khusus, misalnya tersedianya substrat bagi pertumbuhan mikroorganisme yang melimpah. Komunitas mikroba yang secara normal mengkolonisasi organisme ini biasa disebut mikrobiota. Mikrobiota hidup berinteraksi dengan inangnya. Jenis-jenis penyusun mikrobiota dapat memiliki efek negatif, positif, ataupun netral bagi inangnya.
Pada hewan akuatik, mikrobiota yang hidup berasosiasi pada tubuhnya selalu berhubungan dengan air dari lingkungan sekitarnya. Hal ini berbeda dengan pada hewan terestrial yang mikrobiotanya relatif terisolasi dari lingkungan sekitar dan kondisi habitatnya lebih stabil. Akibatnya, mikrobiota pada hewan akuatik bersifat tidak stabil dan biasanya hanya menetap sementara. Air yang masuk ke dalam saluran pencernaan terus menerus membilas dan mengintroduksi spesies-spesies bakteri dari luar. Karena itu, komposisi mikrobiota yang hidup berasosiasi dengan hewan akuatik dapat mencerminkan komposisi mikroorganisme di air yang ada di sekitarnya.
Komunitas bakteri diduga memiliki pengaruh terhadap pertumbuhan dan kemampuan bertahan hidup hewan akuatik. Hal ini dibuktikan dengan misalnya pada udang L. vannamei yang ditumbuhkan pada air yang kaya akan material organik (termasuk di dalamnya mikroba). Ternyata pertumbuhannya lebih baik jika dibandingkan dengan yang ditumbuhkan pada air yang mengalami perlakuan untuk menghilangkan kandungan bahan organiknya, yaitu sebesar 416% untuk larva, 48-89% untuk juvenile, dan 33% untuk broodstock (Leber & Pruder 1988, Moss et al. 1992, Moss 1995). Adanya perbaikan pertumbuhan ini diduga berkaitan dengan komunitas bakteri yang ada dalam kolam pemeliharaan, karena udang yang tumbuh pada air yang banyak mengandung bahan organik memiliki jumlah bakteri aerob yang lebih kecil tetapi keragamannya lebih besar dibandingkan dengan udang yang tumbuh dalam air yang tidak mengandung bahan organik (Moss et al. 2000).
Hasil yang sama juga teramati pada bivalvia. Air laut yang disaring dengan filter berukuran 0,2 μm tidak baik untuk pertumbuhan dan kelangsungan hidup larva
Argopecten purpuratus, dan hasil yang terbaik didapatkan jika menggunakan filter
berukuran 5 μm (Riquelme et al. 1997). Bivalvia kemungkinan tidak dapat dengan efisien mempertahankan partikel berukuran kurang dari 1 μm (Bayne 1983). Air yang disaring dengan filter 5 μm diketahui mengandung partikel dan agregat bakteri yang terdiri dari 4-5 sel yang mungkin dapat ditangkap oleh larva dan memacu pertumbuhan. Prieur et al. (1990) melaporkan bahwa Crassostrea gigas tidak tumbuh dalam periode 7 hari kultur tanpa adanya bakteri. Sementara pada Pecten maximus, filtrat yang diperoleh dengan filter berukuran antara 0,22-1,0 μm dapat memacu pertumbuhan larva (Samain et al. 1987).
Pembentukan dan Dinamika Komunitas Bakteri Pada Hewan Akuatik
Berbeda dengan pada hewan yang ada di darat, mikrobiota pada hewan akuatik hidupnya selalu berhubungan dengan air yang ada di lingkungan sekitarnya. Manusia dan hewan terestrial mengalami perkembangan embrionik dalam amnion, sementara larva dari hewan akuatik harus menghadapi kondisi lingkungan luar pada tahap awal ontogenetiknya. Pada larva invertebrata akuatik yang merupakan filter feeder, terjadi aliran air yang terus-menerus memasuki saluran pencernaannya. Hal ini memperbesar kemungkinan jenis bakteri dalam air mempengaruhi komposisi mikrobiota yang mengkolonisasi saluran pencernaan. Sebagai contoh, jenis-jenis bakteri yang sama ditemukan baik pada air laut maupun saluran pencernaan udang Penaeus japonicus (Moriarty 1990).
Menurut Moriarty (1990), sebagian besar mikroba hanya menempati saluran pencernaan sementara saja. Komposisi mikrobiota dalam saluran pencernaan selalu berubah dengan dinamis seiring masuknya bakteri-bakteri asal air dan makanan. Kelompok bakteri yang sama, tipikal sistem akuatik, berada dalam komposisi mikrobiota. Kemungkinan proliferasi terjadi dalam saluran pencernaan dan sedikit perbedaan pada jumlah atau spesies inisial dalam air atau pakan menentukan spesies mana yang akan mendominasi mikrobiota. Pada ikan Rainbow Trout, hasil perhitungan jumlah bakteri sangat bervariasi, menunjukkan bahwa mikrobiota tidak mengkolonisasi saluran pencernaan secara permanen (Spanggaard et al. 2000). Usus ikan tidak memiliki mikrobiota yang stabil walaupun saluran gastrointestinal ikan merupakan ekosistem yang sangat berbeda dari lingkungan air sekitarnya. Hasil yang berbeda didapatkan oleh Sakata et al. (1980), yang tidak mendapatkan kesamaan antara kelompok bakteri yang diisolasi dari air, usus ikan, maupun pakannya.
Pada hewan akuatik, bagian dalam tubuh yang biasanya dihuni oleh bakteri adalah saluran pencernaan dan insang. Menurut Lavilla-Pitogo et al. (1992), kolonisasi bakteri pada udang terjadi khusus pada organ-organ saluran pencernaan dan jarang ditemukan pada eksoskleton. Bakteri Vibrio vulnificus ditemukan pada hemolimfe dan saluran pencernaan udang windu P. monodon (Sung & Song 1996), sementara pada tahapan zoea dan mysis larva L. vannamei, V. harveyi ditemukan pada saluran pencernaan
(Soto-Rodriguez et al. 2003). Pada ikan Rainbow Trout, penggunaan Fluorescens In Situ
Hibridization (FISH) pada cryo-section saluran pencernaan menunjukkan bahwa bakteri
mikrobiota dominan berada dalam lumen, bukan pada dinding saluran pencernaan (Spanggaard et al. 2000). Hal ini menyebabkan mikrobiota tidak mengkolonisasi saluran pencernaan secara permanen karena akan terus ikut terbuang bersama kotoran dan terus digantikan oleh yang baru. Pada ikan Mas, terjadi fluktuasi dari hari ke hari dalam komposisi mikrobiota yang diisolasi dari kotorannya (Sugita et al. 1987).
Besarnya pengaruh lingkungan terhadap komposisi mikrobiota pada larva hewan akuatik juga disebabkan oleh belum sempurnanya perkembangan sistem pencernaan dan sistem imun (Timmermans 1987, Vadstein 1997), sehingga jenis-jenis bakteri yang hidup di air atau dari pakan bebas masuk ke saluran pencernaan dan menetap. Tidak ada penghalang berupa asam lambung maupun garam empedu yang merupakan barrier bagi masuknya bakteri dari luar. Grizes et al. (1997) menunjukkan bahwa tidak ada spesies yang dominan dan persisten mengkolonisasi usus larva Seabream (Dicentrarchus labrax) dan Seabass (Sparus aurata) selama tahap perkembangannya. Pada Rainbow Trout, variasi komposisi mikrobiota yang ditemukan pada larva juga dipengaruhi oleh lokasi dan waktu pengambilan sampel yang berbeda (Spanggaard et al. 2000).
Selain transmisi secara horisontal dari lingkungannya, hewan akuatik mendapatkan mikrobiota yang berasosiasi dengannya secara vertikal antar generasi, yaitu dari induknya. Bakteri pengoksidasi sulfur yang merupakan simbion pada insang
Calyptogena magnifica (Bivalvia: Vesicomyidae) diketahui ditransmisi secara vertikal
melalui telur bivalvia (Hurtado et al. 2003). Suatu kelompok α-Proteobacteria yang berasosiasi dengan 7 genus sponge laut yang diambil dari beberapa lokasi berbeda ternyata tidak ditemukan pada air laut di sekitarnya dan diduga ditransmisi secara vertikal melalui larva (Enticknap et al. 2006).
Keragaman Komunitas Bakteri Yang Berasosiasi Dengan Hewan Akuatik
Komposisi mikrobiota pada hewan akuatik berbeda dengan pada hewan terestrial. Hal ini disebabkan berbedanya kondisi habitat pada saluran pencernaan keduanya. Jika pada manusia dan hewan terestrial yang mendominasi mikrobiota adalah kelompok bakteri gram positif anaerobik obligat atau fakultatif (Gournier-Chateau et al. 1994), pada
hewan akuatik mikrobiota didominasi oleh kelompok bakteri gram negatif anaerob fakultatif. Baik pada Crustacea, Bivalvia, ataupun ikan air laut, genus yang dominan adalah Vibrio dan Pseudomonas (Moriarty 1990, Sakata 1990, Prieur et al. 1990). Sementara itu, pada ikan air tawar, genus yang dominan adalah Aeromonas, Plesiomonas, dan berbagai enterobacteriaceae (Sakata 1990). Pada ikan Bandeng (Chanos chanos), jenis-jenis bakteri yang ditemukan dari saluran pencernaannya adalah Moraxella,
Aeromonas, Citrobacter, Carnobacterium, Streptococcus, Bacillus, Pseudomonas, Pleisomonas, Staphylococcus, Flavobacterium, Vibrio, dan Serratia (Aslamyah 2006).
Selain itu juga ditemukan genus yang belum pernah dilaporkan diisolasi dari saluran pencernaan, yaitu Micrococcus, Proteus, Planococcus, dan Kurthia. Keempat genus tersebut umumnya ditemukan pada tanah atau sedimen perairan.
Keragaman mikrobiota juga dapat dipengaruhi oleh kondisi lingkungan. Dalam studi yang dilakukan di kolam-kolam hypersaline di pantai San Francisco, California, diketahui bahwa jenis-jenis bakteri yang berasosiasi dengan Artemia franciscana berfluktuasi mengikuti kadar salinitas. Pada kadar salinitas yang rendah, jumlah bakteri dan keragamannya lebih tinggi, genus yang ditemukan adalah Acinetobacter, Aeromonas,
Citrobacter, Vibrio, bakteri enterik, serta Pleisomonas dan Pseudomonas. Saat kadar
salinitas tinggi, jumlah bakteri maupun keragamannya turun drastis, genus yang ditemukan adalah yang lebih halofilik, yaitu Vibrio spp. (Straub & Dixon 1993).
Untuk kelompok sponge, suatu kelompok α-Proteobacteria yang berkerabat dekat ditemukan pada 7 genus sponge laut yang diambil dari beberapa lokasi. Isolat-isolat ini menunjukkan kemiripan sekuens >99% pada gen 16S rRNA-nya, dan berkerabat dekat dengan Pseudovibrio denitrificans (Enticknap et al. 2006). Sponge juga diketahui berasosiasi dengan kelompok Cyanobacteria, sebagian besar adalah dari kelompok
Synechococcus dan Prochlorococcus serta Oscillatoriales. Beberapa sponge laut dari
ordo Dictyoceratida diketahui berasosiasi dengan spesies Cyanobacteria Oscillatoria
spongeliae dalam jumlah besar di mesohyl (bagian dalam) dari sponge (Ridley et al.
2005). Dari beberapa spesies sponge yang diteliti, hanya satu spesies yang memiliki bakteri γ-proteobacteria (masuk dalam kelompok Rhodobacter berdasar gen 16S rRNA) dalam jumlah yang signifikan, dan hanya satu spesies yang memiliki Cyanobacteria jenis lain (Synechocystissp. ).
Mikrobiota utama yang berasosiasi dengan larva udang, terutama pada tahap nauplii dan zoea, mempunyai keragaman yang tinggi (Vandenberghe et al. 1999). Kelompok
Vibrio bukan kelompok yang dominan pada nauplii P. indicus dan juga pada tahap awal
perkembangan larva L. vannamei (Vandenberghe et al. 1998; Vandenberghe et al. 1999). Sementara itu, dari kelompok Vibrio yang ditemukan, jenis yang dominan adalah V.
alginolyticus, baik pada larva sehat maupun sakit (Vandenberghe et al. 1999). Hasil ini
berlawanan dengan Hameed (1993) yang menemukan bahwa kelompok yang dominan selama tahap perkembangan larva sejak telur sampai post-larvae pada P. indicus adalah
Vibrio.
Pengaruh Pakan Pada Komposisi Mikrobiota
Anggota penyusun mikrobiota juga dapat dibawa oleh pakan, terutama jenis pakan alami/hidup. Jenis pakan alami dapat mempengaruhi komposisi mikrobiota hewan yang memakannya. Pada larva Seabream dan Seabass dari hatchery di Yunani dan Spanyol, komposisi mikrobiota tampaknya menggambarkan komposisi bakteri dalam pakan hidup yang diberikan (Grisez et al. 1997). Pada kelompok yang diberi pakan berupa rotifera, jenis yang dominan adalah V. aunguilarum, V. tubiashi, dan bakteri non Vibrio. Sementara, untuk kelompok yang diberi pakan Artemia, yang dominan adalah V.
alginolyticus, V. proteolyticus, V. harveyi, dan V. natriegens.
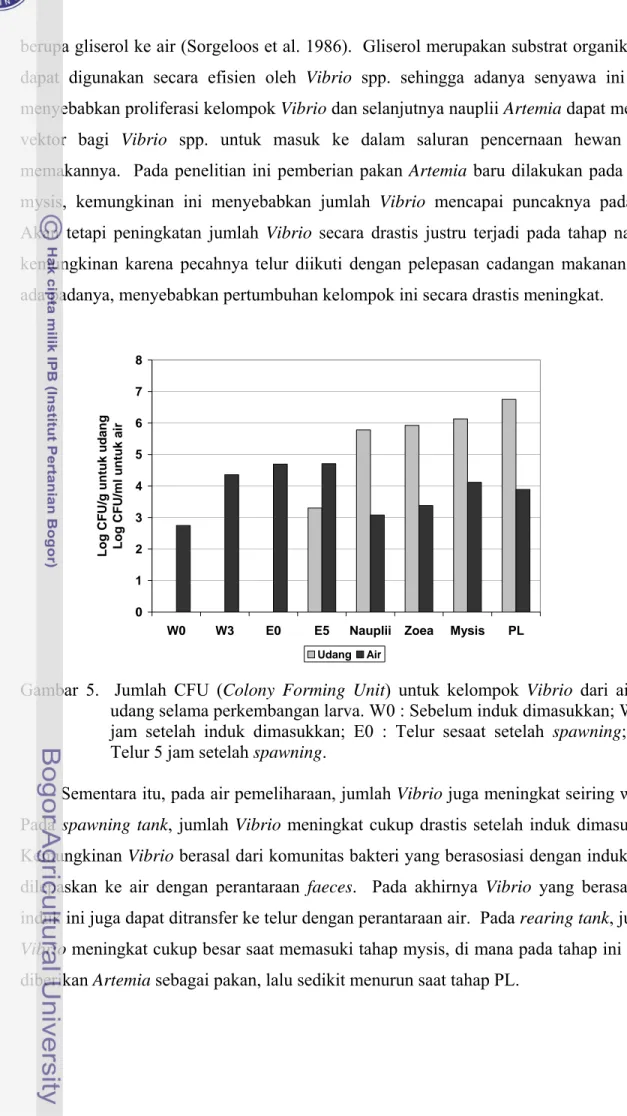
Menurut Liu et al. (1994), populasi bakteri yang berasosiasi dengan larva P.
monodon berubah dari dominan gram positif menjadi dominan gram negatif, saat
diintroduksi dengan nauplii Artemia sebagai pakan pertama untuk larva P. monodon stadia zoea III. V. alginolyticus dan Vibrio spp. yang diisolasi dari tangki penetasan
Artemia ternyata berasosiasi dengan isolat dari tangki pemeliharaan larva tahap mysis,
zoea, maupun post-larva, menunjukkan bahwa Vibrio ini tetap berasosiasi dengan tahap perkembangan berikutnya (Lopez-Torres & Lizarraga-Patida 2001). Akan tetapi, kemungkinan Vibrio spp. tersebut berasal dari tangki pemeliharaan, bukan berasosiasi dengan cyst Artemia. Diketahui bahwa bakteri dominan yang berasosiasi dengan cyst
Artemia adalah kelompok Gram Positif (Lopez-Torres &Lizarraga-Patida 2001). Bakteri
permukaan yang terekspos pada air laut (Duan et al. 1995). Lapisan film tersebut terdiri dari polisakarida, terutama glukosa dan galaktosa (Rodriguez & Bhosle 1991) yang dapat melindungi bakteri terhadap pencucian dan klorinasi dinding tangki pemeliharaan. Bakteri tertentu dapat berkembang selama proses pemeliharaan dalam tangki, walau suplai air, udara, maupun pakan selalu dikontrol.
Pada teknik akuakultur, beberapa metode telah digunakan untuk memperbaiki kualitas mikrobiologis dari pakan hidup, seperti disinfeksi dengan senyawa kimia (GomezGil-RS et al. 1994) dan perendaman dalam air tawar (Rodriguez et al.1991). Secara fisik, radiasi digunakan untuk menurunkan jumlah bakteri yang berasosiasi dengan pakan hidup. Pada Turbot, kemampuan bertahan hidup lebih besar pada kelompok larva yang diberi pakan rotifera (Brachionus plicatilis) yang diiradiasi dengan ultra violet, di mana kandungan bakterinya turun sebesar 88%. Hal ini diduga karena laju kolonisasi bakteri pada saluran pencernaan larva berkurang karena rendahnya jumlah bakteri inisial pada rotifera (Munro et al. 1999).
Selain cara fisik dan kimia, cara lain yang digunakan untuk mengurangi kandungan bakteri pada pakan hidup dapat dilakukan dengan pemberian mikroalga. Pada A.
franciscana, pemberian mikroalga Tetraselmis sp. mampu menurunkan proliferasi bakteri
dan mengintroduksi spesies baru dari kultur alga sehingga menghasilkan mikrobiota yang lebih stabil dengan keragaman tinggi, yang kemudian dapat ditransfer secara langsung kepada mikrobiota larva Halibut (Hipoglossus hipoglossus) yang memakannya (Olsen et al. 2000). A. franciscana berumur 2 hari memiliki jumlah total bakteri 24000 ± 10700 CFU/hewan dengan proporsi Vibrio dan bakteri hemolitik berturut-turut sebesar 58% dan 10% dari total. Mikrobiota didominasi oleh V. alginolyticus (30 dari 40 isolat yang diuji). Setelah 4 jam inkubasi dengan Tetraselmis sp., jumlah bakteri turun rata-rata sebesar 75% dan V. alginolyticus menjadi kurang dominan. Keragaman relatif mikrobiota juga meningkat dari 0,17 menjadi 0,40. Setelah 24 jam inkubasi, jumlah total bakteri semakin turun sementara prevalensi Vibrio menurun dan mikrobiota didominasi oleh genus lain. Keragaman relatif bertambah menjadi 0,82.
Bagaimana mikroalga dapat mempengaruhi mikrobiota pada hewan yang digunakan sebagai pakan hidup sampai saat ini belum jelas. Salah satu hipotesis adalah mikroalga menyebabkan perubahan dalam komposisi dan menurunkan jumlah bakteri pada pakan
hidup dengan cara mengeluarkan isi saluran pencernaan yang merupakan substrat bagi proliferasi bakteri. Umumnya Artemia diberi pakan berupa minyak ikan, suatu substrat yang kaya nutrisi, sehingga memicu proliferasi bakteri tertentu dalam saluran pencernaan. Selain itu, alga diketahui juga memproduksi senyawa antibakteri, seperti dilaporkan untuk Tetraselmis sp.( Austin et al. 1992). Mikroalga mempunyai efek positif bagi pertumbuhan larva ikan selama pemberian pakan pertama dengan meningkatkan kualitas nutrisi pakan dan faktor mikrobialnya. Penambahan mikroalga pada tangki pemeliharaan dapat mengubah komposisi mikrobiota yang berasosiasi dengan larva, tergantung spesies dan tahap perkembangan alga yang digunakan (Salvesen et al. 2000).
Hasil yang berbeda diamati pada Macrobrachium rosenbergii yang diberi pakan hidup berupa nauplii A. salina. Jenis bakteri Aeromonas liquefaciens dan V. anguilarum umum ditemukan pada larva M. rosenbergii tetapi tidak terdapat pada nauplii A. salina (Colorni 1985). Hal ini menunjukkan bahwa larva M. rosenbergii memiliki kemampuan untuk mempertahankan komposisi mikrobiota normal dalam saluran pencernaannya dari pengaruh introduksi bakteri asal makanan.
Jenis pakan buatan juga dapat mempengaruhi komposisi mikrobiota pada hewan akuatik. Pada larva Seabass, pemberian pakan buatan menyebabkan jumlah total bakteri jauh lebih banyak dibandingkan yang diberi pakan hidup. Selain itu, keragaman mikrobiota juga lebih besar pada kelompok yang diberi pakan hidup Artemia (Gatesoupe et al. 1997). Pemberian diet dengan defisiensi besi juga dapat menurunkan jumlah total bakteri dan meningkatkan keragamannya. Hal yang sama juga teramati pada studi yang dilakukan oleh Garatun-Tjeldsto et al. (1989) pada larva ikan Cod di mana pemberian pakan buatan terlalu dini dapat menyebabkan larva gagal bermetamorfosis dan ditemukan sejumlah besar bakteri pada lumen ususnya. Lepasnya komponen larut air dari partikel pakan diduga dapat menyebabkan proliferasi bakteri (Garatun-Tjeldsto 1994). Akan tetapi, pada Japanese Flounder dan Rockfish, jumlah bakteri mencapai maksimum selama periode pemberian pakan hidup, lalu justru menurun setelah diberi pakan buatan (Tanasomwang & Muroga, 1988; 1989).
Jenis pakan juga mempengaruhi komposisi mikrobiota berdasarkan kemampuan hidrolitiknya. Jenis pakan tertentu dapat menseleksi jenis bakteri tertentu yang memiliki kemampuan untuk menggunakannya sebagai substrat. Pada larva Seabass yang diberi
pakan buatan terdapat sejumlah besar Vibrio yang menghasilkan siderofor, protease, fosfolipase, dan amilase, yang hanya diproduksi oleh 25-60% mikrobiota pada kelompok yang diberi pakan hidup. Sebaliknya, lipase adalah produk ekstraselular utama pada kelompok yang diberi pakan hidup (sebesar 80% dari total isolat) tetapi jarang diproduksi pada kelompok yang diberi pakan buatan (<10%) (Gatesoupe et al. 1997).
Interaksi Mikrobiota Dengan Inang
Peran mikrobiota dan interaksinya dengan inang pada hewan akuatik belum jelas. Ada dugaan bahwa bakteri dapat menjadi sumber nutrisi langsung bagi inangnya, dengan kata lain bakteri yang masuk dalan saluran pencernaan akan dicerna. Beberapa ikan memiliki kebiasaan grazing (menelan sedimen) yang kemungkinan dilakukan untuk mendapatkan mikroorganisme yang bisa menjadi sumber nutrisi untuknya. Pertumbuhan
Cherac quadricarinatus air tawar yang dipelihara pada kolam tanah lebih baik
dibandingkan dengan spesies yang dipelihara pada kolam tanah lebih baik dibandingkan dengan yang dipelihara dalam tangki (Xue et al. 1999). Perbedaan ini disebabkan kemampuan ikan yang dipelihara dalam kolam tanah untuk memperoleh tambahan nutrisi yang diperlukan dari bahan detritus di dasar kolam, atau dapat juga detritus yang ditelan ini merupakan sumber inokulum bagi pembentukan mikrobiota saluran pencernaan. Pada beberapa sistem, bakteri heterotrof dan yang berasosiasi dengan detritus merupakan sumber nutrisi yang dominan bagi udang (McIntosh 1999).
Selain sebagai nutrisi langsung, mikrobiota dapat membantu nutrisi inangnya dengan cara membantu pencernaan pakan. Peranan bakteri dalam pencernaan ini juga ditunjukkan oleh terstimulasinya berbagai enzim pencernaan seperti serin protease, kolagenase, amilase, selulase, laminarinase, dan lipase pada udang yang ditumbuhkan pada air yang kaya akan material organik (Moss et al. 2001). Hasil penelitian Aslamyah (2006) menunjukkan kontribusi mikroba yang mampu menghasilkan enzim-enzim ekstraselular pada aktivitas α-amilase, protease, dan lipase saluran pencernaan ikan Bandeng (Chanos chanos) berturut-turut sebesar 12,25; 8,63; dan 5,42 U/g/menit. Hal ini menunjukkan bahwa selain enzim pencernaan yang diproduksi oleh inang, enzim ekstraselular yang diproduksi oleh mikrobiota juga dapat berperan dalam pencernaan di usus inang. Hasil berlawanan dilaporkan oleh Pollack dan Montgomery (1999) yang
menyatakan bahwa bakteri yang dominan pada usus Surgeonfish justru menurunkan aktivitas enzimatik inang dan pada saat yang bersamaan memproduksi enzim lipase yang aktif.
Jenis-jenis penyusun mikrobiota pada hewan akuatik diketahui memang memiliki kemampuan untuk memproduksi enzim-enzim ekstraselular. Vibrio dan Enterobacter pada usus Grey Mullet diketahui memproduksi enzim protease, amilase, kitinase, dan lesitinase yang mungkin memiliki kontribusi pada proses pencernaan (Hamid et al. 1979). Enzim protease bakteri menjadi penting pada ikan yang tidak memiliki lambung seperti Grey Mullet, atau pada larva ikan yang lambungnya belum berkembang. Pada Bandeng (Chanos chanos), dari 18 isolat asal saluran pencernaan yang berhasil diisolasi, 4 isolat bersifat amilolitik aerob, 3 isolat amilolitik anaerob, 5 isolat proteolitik aerob, 2 isolat proteolitik anaerob, 2 isolat lipolitik aerob, serta 2 isolat lipolitik anaerob. Selain itu, inokulasi dengan Carnobacterium sp., probiotik yang mampu memproduksi enzim amilase, diketahui dapat meningkatkan aktivitas amilase dalam saluran pencernaan ikan Bandeng serta meningkatkan penggunaan karbohidrat pakan sebesar 50% dan menurunkan penggunaan protein pakan sebesar 20% oleh juvenile ikan Bandeng (Aslamyah 2006).
Selain kontribusinya pada pencernaan inang, enzim-enzim dan produk ekstraselular lain dari bakteri mikrobiota juga berperan dalam virulensi patogen. Enzim fosfolipase terlibat dalam toksisitas beberapa produk ekstraseluler dari patogen (Chung et al. 1991, Fouz et al. 1993). Pada Aeromonas salmonicida, protease merupakan salah satu faktor virulensi yang penting, demikian juga pada Aeromonas hydrophila dan Aeromonas
sobria (Tajima et al. 1983, Shieh 1987). Sementara itu, V. anguilarum memproduksi zincmetalloprotease dalam virulensinya (Norqvist et al. 1990). A. hydrophila juga
memproduksi suatu metalloprotease yang bersifat letal pada Rainbow Trout (Rodriguez et al. 1992) tetapi diduga peranannya tidak langsung merusak sel inang, tetapi dengan membebaskan besi dari transferrin (Hirst & Ellis 1996). Siderofor merupakan faktor virulensi pada V. anguilarum yang berperan dalam kompetisi untuk mendapatkan besi (Pybus et al. 1994). Siderofor juga dapat memberikan efek probiotik karena suplai besi yang berlebihan dapat menurunkan ketahanan inang terhadap penyakit (Gatesoupe 1997).
Bakteri-bakteri penyusun mikrobiota juga menghasilkan produk lain yang menguntungkan bagi inangnya. Sebagian produk berupa nutrien esensial yang tidak dapat diproduksi sendiri oleh inang atau senyawa yang dapat dimetabolisir oleh inang. Beberapa bakteri diketahui dapat menghasilkan vitamin B12 (Sugita et al. 1991). Produksi poliamin oleh Vibrio spp. dapat mempercepat pematangan saluran pencernaan dan ekspresi enzim-enzim pencernaan (Yamamoto et al. 1979). V. pelagius yang berasal dari larva Turbot (Schophthalmus maximus) diketahui dapat menghasilkan asam lemak eikosapentaenoat (20:5 n-3) sebesar 9% dari total asam lemaknya (Ringo et al. 1992a), demikian juga Vibrio spp. dari air tawar (Ringo et al. 1992b). Calyptogena magnifica (Bivalvia: Vesicomyidae) bahkan tergantung pada bakteri pengoksidasi sulfur simbion pada insangnya untuk kebutuhan nutrisinya (Hurtado et al. 2003).
Inang juga dapat memproduksi metabolit yang menguntungkan bagi bakteri simbionnya. Dengan kondisi aerasi optimum, enzim tyrosinase pada sponge Suberites
domuncula diekspresi. Enzim ini mensintesis senyawa difenol dari senyawa monofenol.
Senyawa difenol ini digunakan oleh bakteri endosimbion untuk pembentukan energi (Muller et al. 2004).
Komunitas bakteri diduga juga memiliki peran dalam pertahanan inang terhadap patogen. Peningkatan respons imun terlihat pada udang yang dipelihara pada air yang kaya akan bahan organik (Primavera et al. 2000). Mekanisme bagaimana mikrobiota dapat membantu pertahanan diri inangnya diduga karena adanya kompetisi antara mikrobiota dengan bakteri patogen dalam saluran pencernaan, baik untuk tempat pelekatan maupun untuk sumber nutrisi. Adanya kompetisi ini juga mencegah tercapainya quorum sensing, yang merupakan mekanisme umum pengaturan ekspresi gen virulensi pada kebanyakan patogen. Selain itu, bakteri-bakteri mikrobiota juga mampu menghasilkan senyawa-senyawa yang bersifat antibakteri untuk berkompetisi dengan bakteri patogen.
Selain membantu ketahanan inang terhadap penyakit, mikrobiota juga dapat menimbulkan penyakit, dan hal ini berkaitan dengan ketidakseimbangan komposisi mikrobiota di mana bakteri patogen oportunis berkembang mendominasi mikrobiota. Pada studi yang dilakukan Sung et al. (2001) di Taiwan, turunnya keragaman spesies bakteri di kolam pemeliharaan larva P. monodon diikuti dengan berkembangnya penyakit
yang menyebabkan kematian massal. Banyak spesies Vibrio ditemukan berasosiasi dengan penyakit yang menyebabkan kematian larva secara massal, seperti di Taiwan (Sung et al. 2001), Filipina (Alapide-Tendencia & Dureza 1997), serta ekuador dan Meksiko (Vandenberghe et al. 1999). Akan tetapi banyak juga spesies Vibrio yang diisolasi dari udang yang sehat, dan hal ini menunjukkan sifat oportunistik dari kelompok bakteri ini (Hameed 1993, Ruangpan & Kitao 1991, Vandenberghe et al. 1998; 1999). Menurut Pizzuto dan Hirst (1995), Galur Vibrio yang virulen jarang ditemukan dan virulensi kemungkinan terjadi akibat adanya transfer faktor-faktor virulensi.
Eksplorasi Komunitas Bakteri Berbasis Molekuler
Studi mengenai keragaman mikrobiota pada hewan akuatik masih menemui kendala. Selama ini yang digunakan adalah teknik kultur, di mana teknik ini sangat tergantung pada medium yang digunakan dan kondisi kultur. Medium buatan dan kondisi kultur menguntungkan bagi sebagian bakteri yang umumnya bersifat heterotrof aerobik, sehingga hanya golongan ini yang terwakili. Hanya sebagian kecil bakteri yang teramati dengan mikroskop yang dapat dikulturkan. Misalnya, pada kulit ikan, hanya kurang dari 0,01% dari total bakteri yang dapat dikulturkan (Bernardsky & Rosenberg 1992). Sementara pada usus ikan Rainbow Trout, persentase bakteri yang dapat dikulturkan lebih besar, rata-rata sekitar 50% dari total bakteri yang ada (Spanggaard et al. 2000).
Masalah ini dapat diatasi dengan teknik berbasis molekuler, yaitu dengan analisis DNA atau RNA yang diambil langsung dari lingkungan. Sampai saat ini beberapa gen telah digunakan sebagai penanda molekuler bagi kelompok bakteri, dan yang paling banyak digunakan adalah gen penyandi 16S rRNA. Gen ini bersifat ubikuitus hanya pada kelompok bakteri sehingga dapat menjadi penanda universal yang spesifik untuk kelompok bakteri. Beberapa teknik berbasis molekular yang sering digunakan untuk analisis komunitas bakteri adalah Denaturing Gradient Gel Electrophoresis (DGGE),
Amplified Ribosomal DNA Restriction Analysis (ARDRA), Fluorescent in-situ Hybridization (FISH), DNA Microarray, dan yang baru-baru ini berkembang adalah Terminal Restriction Fragment Length Polymorphism (T-RFLP) (Marsh et al. 2000).
mengenai komunitas bakteri, seperti biomassa viable, struktur komunitas (profil genotipe populasi bakteri), bahkan status fisiologis dan nutrisi serta aktivitas metabolisme.
T-RFLP merupakan alat yang efektif untuk membedakan komunitas bakteri pada berbagai lingkungan. Metode ini merupakan metode yang cepat untuk menemukan perbedaan utama antar komunitas (Blackwood et al. 2003). T-RFLP merupakan modifikasi dari teknik ARDRA yang telah lebih dulu digunakan secara luas untuk analisis komunitas bakteri. T-RFLP dapat digunakan untuk melihat secara kuantitatif perubahan pada komunitas mikroba karena kemelimpahan relatif suatu populasi dalam komunitas dapat dibandingkan antar sampel dengan perlakuan yang berbeda atau diambil pada waktu yang berlainan. Masing-masing TRF mewakili minimal satu spesies bakteri, terlihat sebagai satu puncak, sementara luas daerah di bawah puncak tersebut menunjukkan kemelimpahan relatifnya. Teknik ini telah digunakan untuk analisis komunitas bakteri maupun fungi pada berbagai lingkungan, seperti komunitas mikrobiota pada manusia (Hayashi et al. 2002, Jernberg et al 2005), cairan tanaman kantung semar (Yogiara et al. 2006), selama fermentasi nata de coco (Seumahu et al. 2007) dan pada rizosfer tanaman kapas transgenik dan non transgenik (Hala et al. 2002). Dari beberapa studi tersebut, T-RFLP terbukti merupakan alat yang baik untuk memonitor perubahan sidik jari komunitas mikroba sesuai waktu atau perlakuan.
Seperti ditunjukkan pada Gambar 2., pada teknik T-RFLP yang diukur adalah polimorfisme ukuran dari panjang potongan enzim restriksi pada ujung produk PCR yang telah dilabel (disebut TRF, Terminal Restriction Fragment) yang kemudian dibaca pada mesin Automated DNA Sequencer. Sistem elektroforesis kapiler pada sequencer menghasilkan data dengan resolusi dan presisi yang tinggi dalam penentuan panjang TRF. Hal ini menyebabkan interpretasi data T-RFLP relatif mudah jika dibandingkan dengan teknik lain seperti DGGE.
Gambar 2. Prosedur dalam analisis T-RFLP
Umumnya, gen penyandi small-subunit (SSU) rRNA yang digunakan sebagai marker filogenetik untuk T-RFLP sehingga hasil analisis T-RFLP dapat digunakan untuk investigasi dalam kisaran luas berbagai kelompok berbeda yang hidup dalam ekosistem di alam, termasuk Bacteria, Eukarya, dan Archaea. Akan tetapi, gen fungsional juga dapat digunakan sebagai marker molekular dalam T-RFLP untuk mentargetkan kelompok yang memiliki fungsi tertentu dari mikroorganisme secara spesifik (Lueders & Friedrich 2003). Marker gen fungsional yang menyandikan enzim tertentu pada jalur metabolisme dapat mengungkapkan fungsi dari mikroorganisme yang terdeteksi dalam lingkungannya. T-RFLP juga dapat memberikan profil komunitas untuk gen fungsional seperti resistensi terhadap logam berat, fiksasi nitrogen, denitrifikasi, dan metanogenesis.
Walaupun memiliki kelebihan dalam segi kepraktisan dan kecepatan, teknik ini juga memiliki keterbatasan. Karena hanya menggunakan ujung 5’ dari gen 16S rRNA, resolusi yang dihasilkan T-RFLP lebih rendah dibandingkan teknik lain yang menggunakan keseluruhan gen. Kesulitan dalam identifikasi anggota komunitas bakteri sering terjadi. Selain itu, adanya bias dalam proses isolasi DNA dan PCR menyebabkan hasil yang didapat tidak konsisten, terutama jika yang digunakan sebagai marker adalah gen fungsional (Lueders & Friedrich 2003).
Digesti dengan enzim restriksi
Produk PCR gen 16S rRNA dengan primer terlabel pada ujung 5’
TRF (Terminal Restriction Fragment)
1 filotipe
Peak heigth/area Jumlah Relatif 1 TRF
5’ 3’
Ukuran TRF (bp) Identitas
Automated DNA sequencer
ARDRA merupakan teknik yang cukup rumit dalam pengerjaan jika dibandingkan dengan TRFLP. Akan tetapi, teknik ini lebih diskriminatif karena teknik ini menggunakan seluruh produk amplifikasi gen 16S rRNA untuk analisis, sehingga resolusi yang dihasilkan juga lebih baik.. ARDRA juga dianggap lebih handal untuk mengetahui jenis apa saja yang berada dalam komunitas bakteri. Hal ini disebabkan gen 16S yang diklon dapat disekuens untuk identifikasinya.
Kelemahan metode ARDRA adalah dalam segi kepraktisan dan biayanya. Waktu yang dibutuhkan relatif lama dengan melalui urutan pekerjaan yang banyak dan berulang. Teknik ini memerlukan proses kloning gen 16S rRNA, yang kemudian diikuti dengan analisis panjang fragmen hasil restriksi dan sekuensing untuk penentuan identitas. Jumlah pustaka gen 16S rRNA yang dibutuhkan juga besar untuk dapat menggambarkan komunitas secara lengkap. Karena itu untuk penerapannya sebagai alat monitoring komunitas bakteri pada suatu lingkungan menjadi tidak praktis.
BAHAN DAN METODE
Tempat dan Waktu Penelitian
Penelitian dilakukan September 2006 sampai Desember 2007. Larva L. vannamei dipelihara di Hatchery Marine Research Center PT. Central Pertiwi Bahari Lampung. Proses kultur bakteri dan ekstraksi DNA dilakukan di Marine Research Center PT. Central Pertiwi Bahari Lampung. Analisis T-RFLP dan ARDRA serta identifikasi isolat dan klon dilakukan di R and D PT. Charoen Phokphand Indonesia, Jakarta.
Pengambilan dan Perlakuan Sampel
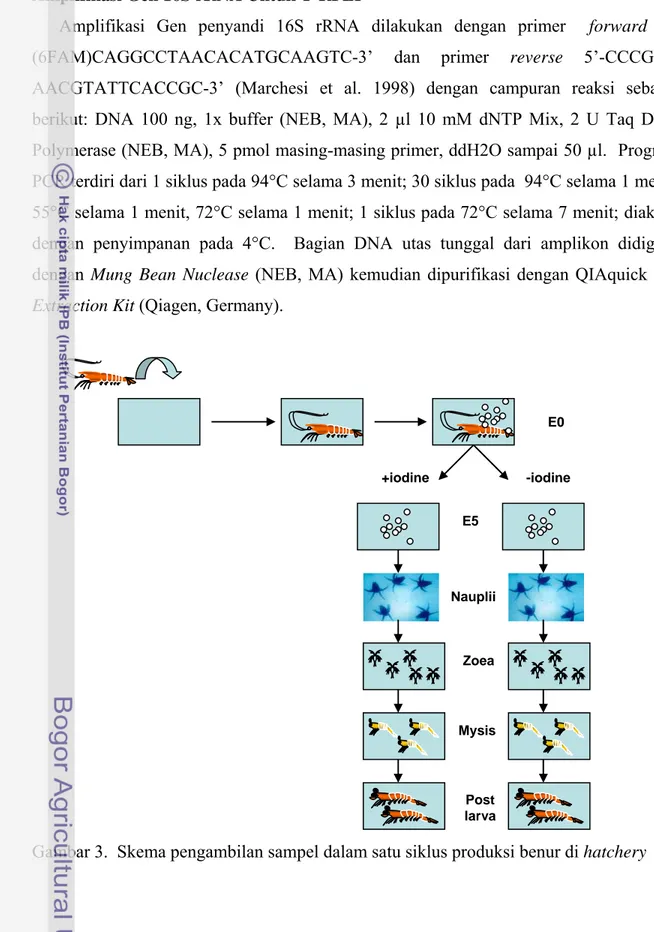
Sampel yang diambil berasal dari siklus produksi yang sama, masing-masing terdiri dari telur, nauplii, zoea, mysis, dan post larva (Gambar 3.). Perlakuan perendaman dengan povidone iodine 20 ppm dilakukan pada sebagian telur dan nauplii yang kemudian dipelihara terpisah dari yang tidak mendapat perlakuan.
Untuk telur, dilakukan 2 kali sampling, yaitu sesaat setelah spawning (E0) dan 5 jam kemudian saat dipanen untuk dipindahkan ke hatching tank (E5). Bersamaan dengan sampling telur atau larva, juga diambil sampel air pemeliharaannya. Selain itu juga dilakukan sampling air pada spawning tank sebelum induk dimasukkan dan 3 jam setelah induk dimasukkan.
Ekstraksi DNA Dari Sampel
Untuk isolasi DNA, telur atau larva diambil sebanyak 0,1 g, dicuci 3 kali dengan 100 ml air laut steril di atas kertas saring steril. Untuk ekstraksi DNA bakteri, telur atau larva diberi perlakuan lysozim 10 mg/ml, kemudian selanjutnya ekstraksi dilakukan dengan
Soil DNA kit (MoBio, CA) mengikuti prosedur yang dianjurkan.
Ekstraksi DNA juga dilakukan pada sampel air tangki pemeliharaan larva. Air sebanyak 1 l disaring dengan membran nilon berpori 0,45 µm. Selanjutnya membran dipotong kecil, kemudian dilakukan proses ekstraksi DNA yang sama dengan pada sampel telur atau larva.
Amplifikasi Gen 16S rRNA Untuk T-RFLP
Amplifikasi Gen penyandi 16S rRNA dilakukan dengan primer forward 5’-(6FAM)CAGGCCTAACACATGCAAGTC-3’ dan primer reverse 5’-CCCGGG AACGTATTCACCGC-3’ (Marchesi et al. 1998) dengan campuran reaksi sebagai berikut: DNA 100 ng, 1x buffer (NEB, MA), 2 µl 10 mM dNTP Mix, 2 U Taq DNA Polymerase (NEB, MA), 5 pmol masing-masing primer, ddH2O sampai 50 µl. Program PCR terdiri dari 1 siklus pada 94°C selama 3 menit; 30 siklus pada 94°C selama 1 menit, 55°C selama 1 menit, 72°C selama 1 menit; 1 siklus pada 72°C selama 7 menit; diakhiri dengan penyimpanan pada 4°C. Bagian DNA utas tunggal dari amplikon didigesti dengan Mung Bean Nuclease (NEB, MA) kemudian dipurifikasi dengan QIAquick Gel
Extraction Kit (Qiagen, Germany).
Gambar 3. Skema pengambilan sampel dalam satu siklus produksi benur di hatchery
E5 Nauplii Zoea Mysis Post larva E0 +iodine -iodine
Digesti Amplikon
Amplikon didigesti dengan enzim restriksi yang memotong dengan frekuensi tinggi, yaitu RsaI dan Sau3AI (NEB,MA) secara terpisah dengan kondisi reaksi : 5U enzim, 1x buffer, 100-200 ng DNA, ddH2O sampai 20 µl, dan diinkubasi pada 37°C semalam. Selanjutnya dilakukan desalting dengan QIAquick Nucleotide Removal Kit (Qiagen, Germany). DNA hasil digesti dilarutkan dalam 30 µl elution buffer.
Analisis T-RFLP
DNA hasil digesti dicampur dengan 1 µl HD-400[ROX] sebagai standar ukuran internal. DNA didenaturasi dengan pemanasan pada 95°C selama 5 menit kemudian segera ditempatkan pada es selama 5 menit. Selanjutnya campuran dimasukkan dalam 96-well plate dan dimasukkan dalam sistem elektroforesis kapiler pada ABIprism™ 310
Automated DNA Sequencer (PE Applied Biosystem). Panjang fragmen terlabel
dideterminasi dengan program GeneScan® (Perkin Elmer). Ukuran TRF yang diketahui dicocokkan dengan database pada situs Ribosomal Database Project II (Marsh et al., 2000). Untuk konfirmasi TRF hasil pemotongan dengan 2 enzim restriksi digunakan program Fragsort (www.oardc.ohio-state.edu/trflpfragsort).
Penghitungan Jumlah Bakteri
Sampel telur atau larva sebanyak 10 mg dicuci 3 kali dengan 100 ml 0,85% NaCl steril di atas kertas saring steril kemudian dihomogenisasi dalam 100 μl 0,85% NaCl steril. Dari homogenat sampel dibuat pengenceran berseri. Untuk tiap pengenceran, 0,1 ml disebar pada medium padat SWC 100% (Sea Water Complete; komposisi per liter medium terdiri dari Yeast extract 1 g, pepton 5 g, gliserol 3 ml, agar 15 g, air laut 750 ml, akuades sampai 1 l) dan medium TCBS (Oxoid) untuk menghitung kelompok Vibrio, kemudian diinkubasi semalam pada 28°C. Cawan dengan jumlah koloni antara 30-300 dipilih kemudian dihitung jumlah koloninya. Koloni dengan penampakan morfologi yang berbeda kemudian diambil dan dimurnikan. Isolat diidentifikasi dengan sekuens gen penyandi 16S rRNA. Pengkulturan juga dilakukan dari air tangki pemeliharaan larva.
Amplifikasi Gen 16S rRNA untuk ARDRA
Amplifikasi dilakukan dengan primer universal untuk kelompok bakteri, 63F (5’-CAGGCCTAACACATGCAAGTC-3’) dan 1387R (5’-CCCGGGAACGTATTC ACCGC -3’) (Marchesi et al. 1998). Kondisi PCR terdiri dari 1 siklus pada 94°C selama3 menit; 30 siklus pada 94°C selama 1 menit, 55°C selama 1 menit, 72°C selama 1 menit; 1 siklus pada 72°C selama 10 menit; diakhiri dengan penyimpanan pada 4°C. Produk PCR dilarikan pada gel agarosa 0,8% kemudian pita berukuran sekitar 1500 bp dipotong dan dipurifikasi dengan QIAquick Gel Extraction Kit (Qiagen, Germany).
Kloning Produk PCR 16S rRNA
Produk PCR diklon pada plasmid pGEM-T Easy Vector (Promega) dengan mengikuti prosedur yang dianjurkan, kemudian ditransformasikan ke dalam E.coli JM109 menggunakan CaCl2 dingin dan heat shock untuk membuat sel kompeten.
Seleksi Klon dilakukan pada medium LA yang ditambah Ampisilin 50 mg/ml dan substrat X-Gal. Selanjutnya, klon berwarna putih diambil, ditumbuhkan pada medium LB dengan Ampisilin 50 mg/ml pada 37°C, dikocok, selama 3-5 jam. Media yang mengandung sel ini digunakan sebagai template untuk amplifikasi insert menggunakan primer M13 dengan campuran reaksi yang terdiri dari 10% kultur, 0,8 mM dNTP, 1μM masing-masing primer,0,1 U/μl Taq DNA Polymerase, 1xbuffer. Kondisi PCR terdiri dari 3 siklus pada 94°C selama 70detik, 56°C selama 45 detik, 72°C selama 90 detik; 23 siklus pada 90°C selama 15 detik, 56°C selama 30 detik, 72°C selama 1 detik; 1 siklus pada 72°C selama 10 menit diakhiri dengan penyimpanan pada 4°C. Klon yang positif mengandung insert berukuran sekitar 1500 bp dipilih secara acak sebanyak 100 klon untuk digunakan dalam analisis berikutnya.
ARDRA (Amplified Ribosomal DNA Restriction Analysis)
Insert didigesti dengan enzim restriksi yang memotong dengan frekuensi tinggi, yaitu MspI dan RsaI (NEB, MA) dengan campuran reaksi 5U enzim, 1x buffer, 100-200 ng
DNA, ddH2O sampai 20 µl, kemudian diinkubasi pada 37°C semalam. Selanjutnya DNA hasil digesti dilarikan pada gel agarose 2%, dengan voltase 70V, selama 2 jam.
masing-masing dari keseluruhan klon yang dianalisis, selanjutnya klon dengan pola restriksi yang dominan disekuens gen penyandi 16S rRNA nya untuk identifikasi.
Sekuensing Gen Penyandi 16S rRNA
Sekuensing dilakukan dengan menggunakan ABI Big Dye Terminator kit (Perkin Elmer) dengan primer 63F dan 1387R secara terpisah. Siklus sekuensing dilakukan pada ABIprism™ 310 Automated DNA Sequencer (PE Applied Biosystem). Sekuens gen penyandi 16S rRNA berukuran sekitar 1300 bp dibandingkan dengan database menggunakan program BLAST pada situs web National Center for Biotechnology Information (http://www.ncbi.nlm.gov/BLAST/). Pohon filogenetika dikonstruksi berdasar sekuens yang telah dijajarkan dengan program MEGA 4 (www.megasoftware.net). Pohon konsensus dikonstruksi dengan metode
neighbor-joining dan 1000 kali bootstrap.
Analisis Keragaman
Bacterial phylotype richness (S) merupakan total puncak TRF berbeda/tipe restriksi
yang berbeda yang ditemukan pada tiap sampel. Indeks Shannon-Wiener (H’) dan
evenness (E) dihitung untuk menggambarkan keragaman komunitas dan nilai penting
relatif dari tiap filotipe dalam keseluruhan komunitas. H’ dihitung dengan rumus sebagai berikut: H’= -Σ (pi) (ln pi) dengan pi adalah kemelimpahan relatif dari fragment i.
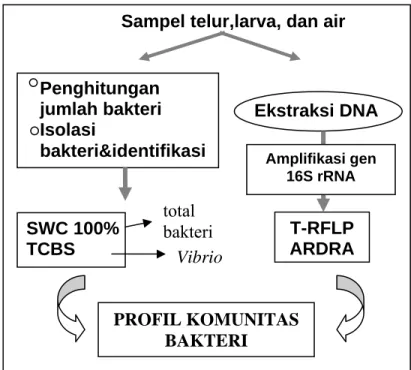
Diagram Alur Pekerjaan Dalam Penelitian Ini
Sampel telur,larva, dan air
SWC 100% TCBS Ekstraksi DNA T-RFLP ARDRA Amplifikasi gen 16S rRNA total bakteri Vibrio PROFIL KOMUNITAS BAKTERI Penghitungan jumlah bakteri Isolasi bakteri&identifikasi