PENGEMBANGAN ANALISIS LISTERIA MONOCYTOGENES
UNTUK JAJANAN PEMPEK DENGAN REAL-TIME
POLYMERASE CHAIN REACTION (rt-PCR)
ARIF MURTAQI AKHMAD MS
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
iii
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Pengembangan Analisis
Listeria monocytogenes untuk Jajanan Pempek dengan Real-time Polymerase Chain Reaction (rt-PCR) adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Januari 2016
Arif Murtaqi Akhmad MS
iv
RINGKASAN
ARIF MURTAQI AKHMAD MS. Pengembangan Analisis Listeria monocytogenes untuk Jajanan Pempek dengan Real-time Polymerase Chain Reaction (rt-PCR). Dibimbing oleh WINIATI P. RAHAYU dan SITI NURJANAH.
Listeria (L.) monocytogenes merupakan bakteri patogen yang dapat menyebabkan bahaya serius bagi manusia. L. monocytogenes dapat menyebabkan komplikasi meningitis. Pangan jajanan anak sekolah sebagai salah satu pangan siap saji memiliki risiko bahaya L. monocytogenes yang tinggi terutama bila dikonsumsi oleh anak sekolah dasar. Tujuan dari penelitian ini adalah untuk mengembangkan metode deteksi L. monocytogenes pada jajanan pempek menggunakan rt-PCR. Isolasi DNA L. monocytogenes dilakukan dengan membandingkan tiga metode ekstraksi yaitu: fenol:kloroform, pemanasan, dan kit komersial (QIAamp DNA Blood Mini Kit). Isolat DNA L. monocytogenes
diamplifikasi menggunakan PCR dengan dua variabel primer yaitu LIM 2 dan LIMRE untuk deteksi gen iap, dan primer DG69 dan DG74 untuk deteksi gen
hlyA. Pengujian dengan rt-PCR diawali dengan denaturasi awal pada 94 °C selama 5 menit diikuti dengan 30 siklus yang terdiri dari 45 detik denaturasi pada 94 °C, 45 detik annealing pada 55 °C, 45 detik ekstensi pada 72 °C dan ekstensi akhir pada 72 °C selama 7 menit. Kemudian dilanjutkan dengan program melting
dengantransisi suhu 72-94 °C dan laju 0.5 °C/detik.
Metode fenol:kloroform menunjukkan ekstraksi DNA terbaik yang lebih
murni. Primer DG69/DG74 lebih spesifik dibandingkan dengan primer LIM 2/LIMRE. Kondisi running yang digunakan menghasilkan kurva amplifikasi
spesifik sampai siklus ke-27. Deteksi DNA dari kultur murni L. monocytogenes
menghasilkan persamaan kurva standar CT = 37.9-3.11 C dengan nilai r sebesar -0.990 dan limit deteksi (LOD) 3.2 x 103 CFU/mL. Deteksi DNA pada sampel pempek menghasilkan persamaan kurva standar CT = 41.03-3.69 C dengan nilai r sebesar -0.997 dan limit deteksi (LOD) 6.3 x 103 CFU/g. Primer DG69 dan DG74 dapat dipertimbangkan sebagai metode spesifik dan sensitif yang dapat diandalkan untuk mendeteksi L. monocytogenes pada jajanan pempek.
Kata kunci: Listeria monocytogenes, metode deteksi, pempek, primer, real-time
v
SUMMARY
ARIF MURTAQI AKHMAD MS. Development of Real-time Polymerase Chain Reaction Analysis for DNA Detection of Listeria monocytogenes in Pempek Snack. Supervised by WINIATI P. RAHAYU and SITI NURJANAH
Listeria (L.) monocytogenes is an important foodborne pathogen which can cause serious human listeriosis. L. monocytogenes can cause meningitis
complications. School children snacks have high potential of risk of
L. monocytogenes because these foods are consumed by children. The aim of this study was to develop rt-PCR method of L. monocytogenes detection in fish based snack. DNA extraction of L. monocytogenes was conducted by comparing three extraction methods: phenol:chloroform, heating, and commercial kit (QIAamp DNA Blood Mini Kit). DNA isolate was amplified by rt-PCR using two variables which were primer LIM 2 and LIMRE for iap gene detection, and primer DG69 and DG74 for hlyA gene detection. The cycling conditions included an initial denaturation at 94 °C for 5 min followed by 30 cycles of 45 s denaturation at 94 °C, 45 s annealing at 55 °C, 45 s extension at 72 °C and 7 min final extension at 72 °C. The thermocycling program was followed by a melting program of transition from 72 to 94 °C with a rate of 0.5 °C/s.
Phenol:chloroform method gave the best extraction result, while primer DG69 and DG74 gave more specific result on standard PCR compared to primer LIM 2 and LIMRE. The running condition showed the fluorescence amplification curve up to 27th cycle. Amplification standard curve of DNA from pure culture resulted the equation CT = 37.9 – 3.11 C with r value -0.990 and limit of detection (LOD) 3.2 x 103 CFU / mL. Amplification standard curve of DNA from spiked pempek sample resulted the equation CT = 41.03 - 3.69 C with r value -0.997 and limit of detection (LOD) 6.3 x 103 CFU / g. In summary, rt-PCR using primer DG69 and DG74 should be considered as reliable method for specific and sensitive detection of L. monocytogenes in pempek.
vi
© Hak Cipta Milik IPB, Tahun 2016 Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjuan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
vii
PENGEMBANGAN ANALISIS LISTERIA MONOCYTOGENES UNTUK JAJANAN PEMPEK DENGAN REAL-TIME
POLYMERASE CHAIN REACTION (rt-PCR)
ARIF MURTAQI AKHMAD MS
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Pangan
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
viii
ix
Judul Tesis : Pengembangan Analisis Listeria monocytogenes untuk Jajanan Pempek dengan Real-time Polymerase Chain Reaction (rt-PCR)
Nama : Arif Murtaqi Akhmad MS
NIM : F251120181
Disetujui oleh Komisi Pembimbing
Prof. Dr. Winiati P Rahayu Dr. Ir. Siti Nurjanah, M.Si Ketua Anggota
Diketahui oleh
Ketua Program Studi Ilmu Pangan
Dekan Sekolah Pascasarjana
Prof. Dr. Ir. Ratih Dewanti-Hariyadi, M.Sc Dr Ir Dahrul Syah, M.Sc.Agr
x
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Mei 2014 sampai Mei 2015 ini ialah pengembangan metode deteksi mikroba patogen, dengan judul Pengembangan Analisis Listeria monocytogenes untuk Jajanan Pempek dengan
Real-time Polymerase Chain Reaction (rt-PCR).
Penulis menyampaikan penghargaan dan ucapan terima kasih yang tidak terhingga kepada :
1. Ibu Prof. Dr. Winiati P Rahayu dan Ibu Dr. Siti Nurjanah yang telah memberikan bimbingan, waktu dan perhatian selama penelitian dan penulisan tesis ini.
2. Bapak Dr. Puspo Edi Giriwono selaku penguji yang telah memberikan saran-saran guna menyempurnakan penulisan tesis ini.
3. Direktorat Jenderal Pendidikan Tinggi yang telah membiayai penelitian ini melalui Program Hibah Kompetensi.
4. Departemen ITP sebagai homebase IPN.
5. Program pascasarjana IPB yang telah membantu selama proses studi.
6. Pimpinan SEAFAST Center dan Departemen ITP yang telah memberikan ijin penggunaan fasilitas di laboratorium.
7. Seluruh pengajar di Program Studi Ilmu Pangan yang telah memberikan bekal ilmu bagi penulis.
8. Politeknik Gorontalo yang telah memberikan kesempatan dan dukungan dana selama menempuh studi di program studi Ilmu Pangan, Sekolah Pascasarjana Institut Pertanian Bogor.
9. Para teknisi di laboratorium SEAFAST Center dan di laboratorium ITP yang telah memberikan bantuan selama penulis melakukan penelitian.
10.Rifina Murtialmira dan Muhammad Afif Naufal yang telah memberikan semangat dan doa kepada penulis.
11.Ibu dan Bapak serta kakak-kakak dan keponakan-keponakan yang memberikan doa dan dukungan kepada penulis.
12.Teman-teman yang telah membantu selama penelitian. Penulis berharap tesis ini dapat bermanfaat.
Bogor, Januari 2016
xi
DATAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xii
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 1
Tujuan Penelitian 2
Manfaat Penelitian 2
2 TINJAUAN PUSTAKA 2
Karakteristik L. monocytogenes 2
Metode Analisis Polymerase Chain Reaction (PCR) 3
3 METODE 5
Bahan 5
Alat 6
Tahapan Penelitian 6
4 HASIL DAN PEMBAHASAN 11
Pemilihan Metode Ekstraksi DNA 11
Konfirmasi Spesifisitas Primer 13
Penentuan Limit Deteksi DNA L. monocytogenes 16
SIMPULAN 18
SARAN 19
DAFTAR PUSTAKA 19
LAMPIRAN 22
xii
DAFTAR TABEL
1. Penelitian tentang pengembangan deteksi L. monocytogenes 4
2. Sitotoksisitas mikroba yang digunakan 6
3. Kemurnian dan konsentrasi DNA 12
4. Hasil analisis kurva melting 15
5. Hasil analisis rt-PCR DNA L. monocytogenes 16
6. Hubungan nilai CT terhadap sampel yang diuji 18
DAFTAR GAMBAR
1. L. monocytogenes 2
2. Grafik sigmoidal proses amplifikasi dengan rt-PCR 5
3. Skema penelitian 7
4. Hasil elektroforesis pada tahap ekstraksi DNA 12 5. Hasil elektroforesis produk PCR dengan primer (a) LIM 2 /LIMRE dan
(b) DG69/DG74 14
6. Kurva amplifikasi uji spesifisitas primer DG69/DG74 dengan rt-PCR 15 7. Kurva standar hubungan log konsentrasi kultur murni L.
monocytogenes dan nilai CT 17
8. Kurva standar hubungan log konsentrasi L. monocytogenes padasampel
pangan spike dan nilai CT 17
DAFTAR LAMPIRAN
1. Komposisi dalam PCR standar dengan Primer LIM 2 / LIMRE 22 2. Kondisi running PCR standar dengan Primer LIM 2 / LIMRE 22 3. Komposisi bahan dalam PCR standar dengan Primer DG69 / DG74 22 4. Kondisi running PCR standar dengan Primer DG69 / DG74 22 5. Komposisi bahan untuk rt-PCR dengan Primer LIM 2 / LIMRE 23 6. Kondisi running rt-PCR dengan Primer LIM 2 / LIMRE 23 7. Komposisi bahan untuk rt-PCR dengan Primer DG69 / DG74 23 8. Kondisi running rt-PCR dengan Primer DG69 / DG74 23
9. Analisis BLAST primer LIM 2 24
10. Analisis BLAST primer LIMRE 24
11. Analisis BLAST primer DG69 25
1
PENDAHULUAN
Latar Belakang
Listeria monocytogenes sering mengontaminasi produk-produk pangan segar maupun pangan dengan minimal proses, seperti produk susu, ikan atau sayuran/buah. L. monocytogenes dapat menyebabkan keracunan darah dan komplikasi meningitis. L. monocytogenes memiliki kemampuan hidup yang luas antara suhu 1-45 °C. Oleh sebab itu, L. monocytogenes sering dijumpai pada pangan siap saji dan pangan yang disimpan pada suhu rendah (Moreno et al.
2012).
Perkembangan teknologi identifikasi DNA yang pesat saat ini dapat membantu untuk mengetahui pencemaran pangan terutama cemaran biologi dan mikrobiologi. Salah satu metode cepat deteksi mikroba yaitu Polymerase Chain Reaction (PCR). Salah satu kelebihan metode PCR yaitu mampu mendeteksi
L. monocytogenes yang masih hidup namun tidak terdeteksi dengan metode pencawanan (viable but not culturable). Aplikasi PCR dalam deteksi bakteri
L. monocytogenes telah banyak diaplikasikan contohnya pada sampel susu dan produk turunannya (Aurora et al. 2008), daging dan produk turunannya (Meyer et al. 2011; Marian et al. 2012), daging ikan dan produk turunannya (Marian et al. 2012), sayuran (Moreno et al. 2012), dan beberapa produk pangan siap saji lainnya (Jamali et al. 2013). Teknologi deteksi DNA dengan real-time PCR (rt-PCR) merupakan pengembangan metode PCR yang dapat mendeteksi dan mengkuantifikasi DNA bakteri secara aktual selama proses berjalan. Oleh karena itu, metode rt-PCR ini relatif lebih cepat dibandingkan dengan PCR standar.
Pangan jajanan anak sekolah (PJAS), khususnya yang berbahan dasar daging ikan, perlu mendapat perhatian lebih. Hal ini disebabkan komposisi daging ikan yang banyak mengandung nutrisi bagi pertumbuhan mikroba dan ditambah lagi praktik penanganan para penjual PJAS yang masih jauh dari higienis. Salah satu contoh PJAS berbasis ikan yang cukup terkenal yaitu pempek. Pempek merupakan makanan khas dari Palembang, Sumatra Selatan yang terbuat dari adonan ikan dan tapioka. Pempek sudah banyak ditemui di tiap-tiap sekolah, perkantoran, pusat perbelanjaan, dan tempat-tempat lainnya di seluruh Indonesia. Pengembangan deteksi L. monocytogenes untuk jajanan pempek dengan rt-PCR perlu dilakukan mengingat masih belum adanya penelitian tersebut baik di dalam maupun luar negeri.
Perumusan Masalah
Penelitian tentang pengembangan metode deteksi L. monocytogenes secara kuantitatif pada PJAS berbasis ikan, khususnya pempek, sangat penting dilakukan mengingat belum adanya penelitian tersebut sampai saat ini. Pengembangan metode ini dimaksudkan untuk memberi informasi metode ekstraksi DNA, penggunaan primer, kondisi running PCR standar dan rt-PCR, sehingga diperoleh
serangkaian metode yang optimal untuk deteksi secara kuantitatif
2
Tujuan Penelitian
Tujuan dari penelitian ini adalah:
1. Menentukan metode ekstraksi DNA optimum untuk L. monocytogenes pada PJAS berbasis ikan, khususnya pempek
2. Menentukan primer yang spesifik untuk pengujian L. monocytogenes
3. Menguji limit deteksi rt-PCR ESCO Swift 48 untuk kultur murni
L. monocytogenes dan sampel pempek yang di-spike dengan
L. monocytogenes
Manfaat Penelitian
Manfaat dari penelitian ini yaitu tersedianya metode analisis yang valid untuk mengidentifikasi dan mengkuantifikasi L. monocytogenes pada sampel PJAS berbasis ikan, khususnya pempek, secara cepat sehingga dapat dimanfaatkan oleh berbagai pihak khususnya pemerintah dan peneliti yang terkait dalam bidang keamanan pangan.
2
TINJAUAN PUSTAKA
Karakteristik L. monocytogenes
L. monocytogenes termasuk bakteri berbentuk batang (Gambar 1), Gram positif, psikotropik, fakultatif anaerob, katalase positif, oksidase-negatif (Campos
et al. 2011). L. monocytogenes umum ditemukan di lingkungan dan berbagai pangan mentah. L. monocytogenes tergolong bakteri yang mampu beradaptasi pada kondisi lingkungan yang luas (Acciari et al. 2011). L. monocytogenes dapat
bertahan pada pH sekitar 4.4 dan konsentrasi garam di atas 14 %.
L. monocytogenes mampu hidup pada suhu 1-45 °C, sehingga makanan yang
disimpan di refrigerator justru memberi kesempatan hidup bagi
L. monocytogenes yang bagi bakteri lain tidak dapat tumbuh (Kramarenko et al.
2013)
3
Listeria monocytogenes dapat menyebabkan penyakit pada manusia melalui infeksi yang disebut listeriosis. Listeriosis dapat menyebabkan penyakit pada orang sehat, terlebih lagi pada balita, penderita immunocompromised, golongan wanita hamil, dan orang yang kehilangan imunitas akibat suatu penyakit (Todd et al. 2011). Listeria monocytogenes sering menyebabkan infeksi fatal seperti meningitis, sepsis, atau infeksi pada janin dan keguguran. Bakteri ini dapat menyebabkan penyakit pada orang sehat seperti gastroenteritis dan demam yang akan sembuh dengan sendirinya (Schuppler et al. 2010). Identifikasi dan kuantifikasi L. monocytogenes pada pangan sangat perlu dilakukan mengingat adanya regulasi dari Eropa yang mensyaratkan jumlah cemaran kurang dari 100 CFU/g L. monocytogenes pada pangan siap saji terutama yang ditujukan pada konsumsi anak kecil dan keperluan medis (Moreno et al. 2012).
Metode Analisis Polymerase Chain Reaction (PCR)
Metode klasik dalam deteksi cemaran mikroba di dalam produk pangan awalnya menggunakan isolasi di media dan konfirmasi melalui tes biokimia dan serologi. Pengembangan metode deteksi mikroba selanjutnya yaitu menggunakan PCR yang menghasilkan data yang lebih akurat dan relatif cepat. Metode konvensional meliputi persiapan media kultur (pengayaan, pengayaan selektif, dan penumbuhan pada agar selektif), penghitungan koloni, pengkarakterisasian dengan uji biokimia, yang dapat dilanjutkan dengan penetapan serotipe (serovar) dengan uji serologi. Hasil uji biokimia yang sama dari genus mikroba yang sama, tidak menjamin bahwa mikroba-mikroba tersebut berasal dari serovar yang sama. Untuk mengatasinya, dilakukan uji serologi untuk mengonfirmasi koloni tipikal yang telah diperoleh dari uji morfologi dan uji biokimia. Cara ini juga digunakan untuk mengidentifikasi mikroba pada tingkat subspesies. Kekurangan dari metode ini ialah memerlukan keahlian yang baik, waktu yang lama, dan mahal. Kekurangan tersebut dapat diatasi dengan metode deteksi cepat terhadap mikroba cemaran yaitu teknik PCR.
Metode PCR dapat melipatgandakan sekuens DNA atau RNA menjadi ribuan sampai jutaan copy, sehingga PCR dapat membantu mendeteksi cemaran mikroba pada pangan (Pochop et al. 2013). Pada mulanya, PCR memiliki kelemahan yaitu tidak dapat membedakan DNA dari organisme hidup dan organisme mati. Tahap penting sebelum dilakukannya metode PCR adalah tahap ekstraksi DNA. Hal ini disebabkan pada matriks sampel pangan, khususnya yang berbasis daging, mengandung beberapa komponen seperti produk reaksi mailard, glikogen, lemak, kolagen, asam fulvat, dan besi yang dapat ikut terpurifikasi bersama DNA target. Keberadaan komponen-komponen tersebut memungkinkan terjadinya penghambatan amplifikasi asam nukleat dengan PCR (Camma et al.
2012). Penelitian tentang deteksi L. monocytogenes dengan PCR sudah cukup banyak dilakukan terutama pada jenis pangan siap saji, susu dan produk hasil susu, sayuran, daging dan produk hasil daging, serta produk ikan (Tabel 1).
4
basa tertentu. Kelebihan rt-PCR dibanding PCR standar antara lain: lebih mudah, akurat, cepat, dan sensitif.
Kuantifikasi rt-PCR secara umum dapat dilakukan dengan dua cara: (1) menggunakan dye fluoresence yang berikatan dengan amplikon, dan (2) menggunakan probe oligonukleotida yang dimodifikasi sedemikian rupa
sehingga akan berpendar ketika terhibridisasi dengan DNA komplementer. Jenis
dye fluorescence yang banyak digunakan sampai saat ini yaitu SYBR® Green sedangkan jenis probe yang digunakan sampai saat ini yaitu TaqMan®, molecular beacons, dan Scorpions® (Dharmaraj 2009). Penggunaan dye fluorescence relatif lebih ekonomis namun spesifisitasnya lebih rendah dibanding probe.
Tabel 1. Penelitian tentang pengembangan deteksi L. monocytogenes
Tujuan Penelitian Sampel Pustaka
Deteksi L. monocytogenes dengan metode PCR kompetitif Perbandingan antara uji PI-PLC dan PCR
dalam deteksi patogenisitas L. Deteksi L. monocytogenes dengan metode
immunoassay VIDAS LMO2 Deteksi L. monocytogenes dengan metode
MPN-PCR
viabel pada pangan nabati siap saji dengan metode pengkulturan, PCR, dan DVC-FISH
Sayuran Moreno et al. 2012
Deteksi L. monocytogenes viabel dengan real-time Reverse Transcriptase PCR
Daging babi Ye et al, 2012 Deteksi dan isolasi Listeria spp dan
L. monocytogenes dalam berbagai jenis media agar selektif
Deteksi L. monocytogenes dengan metode
biosensor multiplex fiber optic
Daging siap saji
Ohk dan Bhunia.
2013
5
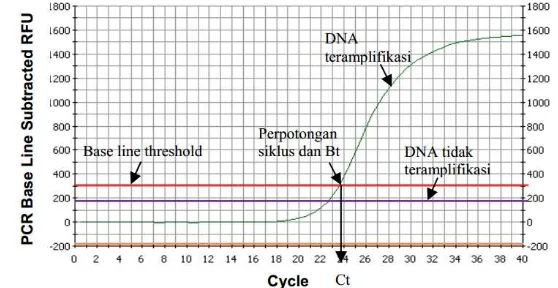
Grafik amplifikasi menunjukkan ada tidaknya DNA target dengan terbentuknya kurva sigmoidal (Gambar 2). Semakin banyak amplikon yang terbentuk maka akan semakin meningkatkan akumulasi flouresen yang terbaca. Akumulasi fluoresen tersebut akan membentuk kurva sigmoidal. Analisis kurva amplifikasi terhadap base line threshold akan menghasilkan nilai yang disebut threshold cycle
(CT) (Edwards et al. 2004).
Kurva standar merupakan hubungan antara log konsentrasi bakteri dan CT. Kurva ini dapat digunakan dalam penetapan limit deteksi dan menghitung konsentrasi bakteri yang belum diketahui pada suatu sampel pangan. Sampel yang digunakan untuk pembuatan kurva standar dibuat serial pengenceran dari pengenceran tertinggi sampai terendah yang masih dapat dideteksi, dengan konsentrasi bakteri dalam kultur antara 100 - 109 CFU/mL. Masing-masing pengenceran diekstrak DNA nya untuk ditentukan nilai CT nya menggunakan rt-PCR. Pembuatan kurva standar hanya dilakukan untuk deteksi mikroba secara kuantitatif.
Gambar 2. Grafik sigmoidal proses amplifikasi dengan rt-PCR
Kurva melting (peleburan) merupakan kurva turunan dari hubungan antara fluoresen dan suhu (dF/dT). Suhu melting (Tm) merupakan suhu saat 50 % amplikon DNA untai ganda terpisah menjadi dua untai tunggal. Nilai Tm diketahui sebagai suhu pada saat munculnya puncak pada kurva melting. Analisis kurva
melting sangat disarankan untuk metode rt-PCR menggunakan SYBR Green® karena dapat membantu mengidentifikasi adanya produk non-spesifik maupun primer dimer (Brisson et al. 2000).
3
METODE
Bahan
Mikroba standar yang digunakan adalah L. monocytogenes ATCC 7644.
Sedangkan untuk penentuan spesifisitas primer digunakan kontrol
6
α-hemolitik Choi dan Hong (2003)
E. coli ATCC 25922 β-hemolitik Snyder dan Carson (2010)
S. aureus ATCC 25923 β-hemolitik Mak et al. (2008)
C. sakazakii ATCC 29544 β-hemolitik Fakruddin et al. (2014)
L. plantarum ATCC 8014 - -
Bahan kimia yang digunakan adalah lisozim ( ≥ 20000 unit/mg protein) (Bio Basic, Canada), proteinase K ( > 30 unit/mg protein) (Peqlab, Jerman), cetyl trimethylammonium bromide (CTAB) (Bio Basic, Kanada), ethylenediaminetetraacetic acid (EDTA) (Amersham BioSciences, UK), QIAamp DNA Blood Mini Kit (Qiagen, Jerman), agarose (Bio Basic, Kanada), larutan Tris-HCl (Promega, US), buffer tris-EDTA (TE), larutan sodium dodecyl sulfate (SDS) (Merck, Jerman), isopropanol (Merck, Jerman), SYBR® Green (Thermo Fisher Scientific, US), amonium asetat (Merck, Jerman), fenol (MP Bio, US), kloroform (J.T. Baker, Meksiko), NaCl (Merck, Jerman), dan etanol (Merck, Jerman). Primer yang digunakan yaitu primer forward LIM 2
(CTAAAGCGGGAATCTCCCTT) / primer reverse LIMRE
(CCATTGTCTTGCGCGTTAAT) (Alpha DNA, US) untuk gen iap serta primer forward DG69 (GTGCCGCCAAGAAAAGGTTA) / primer reverse DG74 (CGCCACACTTGAGATAT) (Alpha DNA, US) untuk gen hlyA. Bahan PJAS berbasis ikan yang digunakan sebagai contoh adalah pempek.
Alat
Alat-alat yang digunakan antara lain adalah rt-PCR Swift™ Spectrum 48 (ESCO, Singapura), AB 2720 PCR Thermal Cycler (Applied Biosystems, US), Bio-Rad Universal Hood II Gel Doc (Bio-Rad, US), Gel Elektroforesis PowerPac® Basic (Bio-Rad, US), kamera digital Canon® EOS 600D (Canon, Jepang), Spektofotometer UV 2450 (Shimadzu, Jepang), magnetic stirrer, stomacher, inkubator, bunsen, pipet mikro, water bath, laminar air flow, hot plate, autoklaf, tabung mikrosentrifus, jangka sorong, dan alat gelas lainnya.
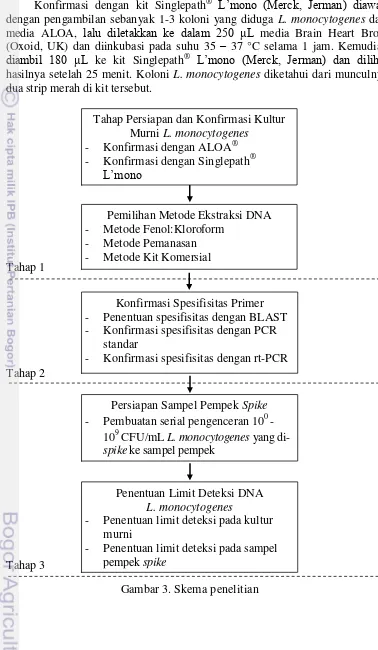
Tahapan Penelitian
Tahapan penelitian utama pada penelitian ini meliputi 3 tahap utama (Gambar 3). Tahap-tahap utama tersebut adalah; (1) pemilihan metode ekstraksi terbaik, (2) konfirmasi spesifisitas primer, (3) penentuan limit deteksi DNA
L. monocytogenes.
1. Persiapan dan Konfirmasi Kultur Murni L. monocytogenes
Konfirmasi L. monocytogenes dilakukan dengan media selektif Agar Listeria Ottaviani Agosti (ALOA) (Merck, Jerman) dan kit immuno assay
7
selama 18 jam pada suhu 37 °C. Selanjutnya kultur murni diambil 0.1 mL untuk ditumbuhkan pada media ALOA, kemudian diinkubasi pada suhu 37 °C selama 24 jam. Koloni L. monocytogenes ditandai dengan warna hijau dengan lingkaran (halo) putih keruh.
Konfirmasi dengan kit Singlepath® L’mono (Merck, Jerman) diawali dengan pengambilan sebanyak 1-3 koloni yang diduga L. monocytogenes dari media ALOA, lalu diletakkan ke dalam 250 μL media Brain Heart Broth (Oxoid, UK) dan diinkubasi pada suhu 35 – 37 °C selama 1 jam. Kemudian
diambil 180 μL ke kit Singlepath® L’mono (Merck, Jerman) dan dilihat hasilnya setelah 25 menit. Koloni L. monocytogenes diketahui dari munculnya dua strip merah di kit tersebut.
Tahap 1
Tahap 2
Tahap 3
Gambar 3. Skema penelitian Tahap Persiapan dan Konfirmasi Kultur
Murni L. monocytogenes
- Konfirmasi dengan ALOA® - Konfirmasi dengan Singlepath®
L’mono
Pemilihan Metode Ekstraksi DNA - Metode Fenol:Kloroform
- Metode Pemanasan - Metode Kit Komersial
Konfirmasi Spesifisitas Primer - Penentuan spesifisitas dengan BLAST - Konfirmasi spesifisitas dengan PCR
standar
- Konfirmasi spesifisitas dengan rt-PCR
Persiapan Sampel Pempek Spike
- Pembuatan serial pengenceran 100 - 109 CFU/mL L. monocytogenes yang
di-spike ke sampel pempek
Penentuan Limit Deteksi DNA
L. monocytogenes
- Penentuan limit deteksi pada kultur murni
8
2. Pemilihan Metode Ekstraksi DNA untuk Kultur Murni
Metode ekstraksi yang digunakan sebagai perlakuan terdiri dari tiga metode, yaitu 1) metode fenol-kloroform, 2) metode pemanasan 3) metode
kit komersial. Penentuan metode terbaik diawali dengan inokulasi
L. monocytogenes pada media TSB selama 18 jam pada suhu 37°C. Selanjutnya kultur murni overnight dalam TSB diisolasi DNA-nya dengan tiga metode tersebut.
1. Metode fenol:kloroform oleh Sambrook dan Russel (2001) yang dimodifikasi
Modifikasi yang dilakukan terhadap metode fenol:kloroform oleh Sambrook dan Russel (2001) adalah penggunaan buffer TE yang digunakan pada 1 mL kultur bakteri untuk menggantikan buffer lisis. Campuran buffer lisis dan 0.2 mg/mL proteinase K diganti dengan campuran antara 500 µL buffer TE 1X dan 100 µL lisozim. Pelarut fenol:kloroform (1:1 v/v) dipakai sebagai ganti dari pelarut fenol:kloroform:isoamil alkohol (25:24:1 v/v/v). Garam 10 M ammonium asetat pH 7.4 digunakan sebagai pengganti 0.3 M natrium asetat pH 4.8. Selain itu, etanol absolut diganti dengan isopropanol. Cara pengeringvakuman pelet diganti dengan cara pengeringudaraan.
Sampel diambil 1 mL, lalu disentrifus dan pelet diresuspensi dengan
500 μL buffer TE 1X sebanyak 2 kali. Setelah disentrifus, pelet diresuspensi dengan 500 μL buffer TE 1X dan 100 μL lisozim, diinkubasi pada suhu 4 °C selama 5 menit. Kemudian ditambahkan 25 μL SDS 10%, 50 μL NaCl
5M, dan 100 μL proteinase K (20 mg/mL), divortex dan diinkubasi pada suhu 65 °C selama 2 jam. Selanjutnya ditambahkan 500 μL fenol:kloroform (1:1 v/v), divortex dan diinkubasi -20 °C selama 30 menit, lalu disentrifus pada suhu 4 °C, 12000 rpm selama 10 menit. Supernatan diambil 500 μL dan dipindahkan ke tabung eppendorf baru, ditambahkan 500 μL fenol:kloroform (1:1 v/v), divortex dan diinkubasi -20 °C selama 30 menit, lalu disentrifus pada suhu 4°C, 12000 rpm selama 10 menit. Supernatan diambil 500 μL dan dipindahkan ke tabung eppendorf baru, ditambahkan
500 μL kloroform, disentrifus pada suhu 4 °C, 12000 rpm selama 10 menit. Supernatan diambil 500 μL dipindahkan ke tabung eppendorf baru, ditambahkan ammonium asetat 10 M pH 7.4 sebanyak 150 μL volume dan isopropanol dingin 500 μL, diinkubasikan -20 °C selama semalam, kemudian disentrifus 14000 rpm selama 30 menit. Selanjutnya pelet
ditambah dengan 500 μL etanol 70 % dan disentrifus 12000 rpm selama 5 menit. Supernatan dibuang dan pelet dikeringudarakan, kemudian
dilarutkan dalam 50 μL buffer TE 1X.
2. Metode pemanasan oleh Rahayu et al. (2009) yang dimodifikasi
Metode ini merupakan metode dari Rahayu et al. (2009) yang mengacu pada metode dari Hein et al. (2001). Modifikasi yang dilakukan pada metode yang digunakan oleh Rahayu et al. (2009) adalah penurunan suhu pemanasan dari 100 menjadi 95 °C.
9
diresuspensi dengan 500 μL buffer TE lalu dihomogenkan dengan cara
divortex. Suspensi tersebut disentrifus kembali pada mikrosentrifus selama 3 menit 14.000 rpm pada suhu 20 °C. Pelet yang dihasilkan diresuspensi
dengan 100 μL lisozim dan dihomogenisasi dengan cara divortex. Kemudian suspensi tersebut diinkubasi pada suhu 25 °C selama 15 menit.
Setelah diinkubasi, sebanyak 100 μL proteinase K ditambahkan ke dalam
suspensi dan divortex sampai homogen. Selanjutnya suspensi diinkubasi pada suhu 65 °C selama 1 jam dan dinkubasi kembali pada suhu 95 °C selama 15 menit. Proses tersebut bertujuan untuk melisiskan sel bakteri dan menghilangkan protein yang terkandung di dalamnya. Penambahan buffer TE berfungsi untuk menjaga kestabilan DNA pada saat melisiskan sel. Suspensi yang telah diinkubasi didinginkan dalam es sampai membeku dan di-thawing dan disentrifus selama 5 menit 14.000 rpm pada suhu 20 °C. Supernatan yang dihasilkan dipindahkan pada tabung mikrosentrifus baru, disimpan pada suhu -20 °C sebagai DNA target.
3. Metode Kit Komersial QIAamp® DNA Blood Mini Kit yang dimodifikasi Modifikasi yang dilakukan terhadap metode dari produsen yaitu penggunaan 1 mL CTAB untuk preparasi awal sampel, kemudian divortex hingga homogen. Suspensi tersebut disentrifus selama 1 menit dengan kecepatan 13000 rpm dan supernatan yang dihasilkan dibuang sehingga menyisakan pelet pada tabung mikrosentrifus. Tahap selanjutnya sesuai dengan petunjuk dari produsen.
QIAamp® DNA Blood Mini Kit tersebut terdiri dari empat jenis buffer (AW1, AW2, AE, dan AL), collection tube, dan kolom mini dimana di dalamnya terdapat filter putih. Teknis pengisolasian DNA dengan kit komersial pertama-tama adalah masing-masing satu mL suspensi kultur murni mikroba spesifik yang terdapat dalam media NB dimasukkan ke dalam tabung mikrosentrifus. Kemudian ditambahkan dengan 1 mL CTAB sebagai suatu modifikasi metode dari prosedur yang ditunjukkan pada
handbook produsen kit, kemudian divortex hingga homogen. Suspensi tersebut disentrifus selama 1 menit dengan kecepatan 13000 rpm dan supernatan yang dihasilkan dibuang sehingga menyisakan pelet pada tabung mikrosentrifus. Jika tidak dihasilkan pelet, maka suspensi disisakan
sebanyak 200 μL supernatan pada tabung mikrosentrifus. Kemudian
ditambahkan proteinase K sebanyak 30 μL ke dalam tabung mikrosentrifus,
dan dihomogenkan dengan vortex kemudian diinkubasi selama 20 menit pada suhu 65 °C.
Setelah itu, ditambahkan buffer AL sebanyak 300 μL dan
dihomogenkan dengan vortex. Suspensi diinkubasi selama 10 menit di dalam water bath pada suhu 65 °C. Setelah itu, ditambahkan 500 μL etanol (100 %), dihomogenkan dengan vortex dan disentrifus selama 2 menit dengan kecepatan 10000 rpm. Supernatan yang dihasilkan dipipet dan dimasukkan ke dalam kolom mini yang sudah dipasang pada collection tube, kemudian disentrifus 8000 rpm selama 1 menit. Sebanyak 500 μL buffer AW1 ditambahkan ke dalam kolom mini yang masih terpasang pada
collection tube dan disentrifus 8000 rpm selama 1 menit.
10
kolom mini, kemudian disentrifus 13000 rpm selama 3 menit. Kolom mini dipindahkan kembali ke dalam collection tube dan ditambah 500 μL buffer AW2 dan disentrifus kembali selama 1 menit 13000 rpm. Kolom mini dipindahkan ke dalam tabung mikrosentrifus steril dan ditambahkan ke
dalamnya sebanyak 80 μL buffer AE yang kemudian disentrifus 8000 rpm
selama 1 menit. Cairan yang diperoleh pada tabung mikrosentrifus disimpan pada freezer dengan suhu -20 °C sebagai DNA target.
Visualisasi Hasil Ekstraksi DNA
Hasil ekstraksi DNA dari tiga metode tersebut dinilai melalui penentuan kualitas dan kemurnian DNA. Tahap ini diawali dengan elektroforesis DNA menggunakan 1.5 % gel agarose pada tegangan 100 volts. Setelah dimigrasi, gel berisi DNA direndam dalam larutan Ethidium Bromida (EtBr) kemudian dicuci menggunakan air selama kurang lebih 10 menit. Setelah itu, gel dimasukkan ke dalam Bio-RAD gelldoc untuk melihat pita DNA genom. Teknis pengukurannya adalah dengan membandingkan nilai absorbansi pada panjang gelombang 260 dan 280 nm (A260/A280). Metode ekstraksi terpilih didasarkan pada jumlah konsentrasi DNA genom yang paling tinggi.
3. Konfirmasi Spesifisitas Primer
Tahap awal yang dilakukan sebelum pemilihan primer yaitu penentuan spesifisitas masing-masing primer menggunakan BLAST. Pemilihan primer dilakukan dengan prosedur kerja PCR standar (Lampiran 1, 2, 3 dan 4). Amplikon yang dihasilkan dari PCR standar dinilai dengan pengamatan menggunakan gel elektroforesis. Primer yang dijadikan perlakuan adalah : a. Primer LIM 2 dan LIMRE yang merupakan sekuens dari gen iap (Dadkhah
et al. 2012).
b. Primer DG69 dan DG74 yang merupakan sekuens dari gen hlyA (Choi dan Hong 2003).
DNA yang digunakan yaitu DNA L. monocytogenes sebagai DNA target, dibandingkan dengan DNA S. aureus dan C. sakazakii sebagai DNA bakteri lain. Primer terbaik didasarkan pada spesifisitasnya menggunakan PCR standar. Primer terpilih digunakan untuk tahap-tahap selanjutnya.
Spesifisitas primer dinilai menggunakan prosedur kerja rt-PCR (Lampiran 5, 6, 7 dan 8). DNA yang digunakan yaitu DNA L. monocytogenes
sebagai DNA target, dibandingkan dengan DNA E. coli, S. aureus, L. plantarum, dan C. sakazakii sebagai DNA bakteri lain. Parameter yang
diamati adalah pola kurva amplifikasi dan kurva melting. Spesifisitas primer ditunjukkan oleh tidak adanya amplifikasi dari DNA lain. Spesifisitas primer pada kurva melting ditunjukkan dengan nilai puncak kurva melting (Tm) yang berbeda antara DNA target, kontrol negatif dan kontrol DNA bakteri lain.
4. Persiapan Sampel Pempek Spike
Sampel yang digunakan yaitu pempek yang telah direbus dan belum digoreng. Langkah pertama dilakukan pengambilan sampel pempek sebanyak 25 g untuk dihomogenkan dengan 75 mL BPW yang mengandung konsentrasi
11
yang telah di-spike digunakan sebagai sampel untuk penentuan limit deteksi DNA L. monocytogenes pada sampel pangan spike.
5. Penentuan Limit Deteksi DNA L. monocytogenes
Pengujian dilakukan untuk deteksi DNA L. monocytogenes kultur murni dan sampel pangan spike (SPS). Sensitivitas dari realtime PCR untuk deteksi
L. monocytogenes ditentukan dengan pembuatan kurva standar hubungan nilai
threshold cycle (CT) dan log konsentrasi kultur murni. Kurva standar memberikan persamaan linear yang digunakan untuk mengukur limit deteksi
L. monocytogenes dalam sampel pangan. Persamaan linear kurva standar digunakan untuk menghitung konsentrasi bakteri yang belum diketahui dalam suatu kultur. Konsentrasi kultur L. monocytogenes dapat dihitung dengan memasukkan nilai CT hasil amplifikasi sebagai nilai y pada persamaan linear, kemudian nilai x yang diperoleh dicari nilai antilog-nya.
Tahapan pertama persiapan sampel untuk kultur murni diawali dengan membuat serial pengenceran dari pengenceran tertinggi sampai terendah yang
masih dapat dideteksi, dengan konsentrasi bakteri dalam kultur antara 100 - 109 CFU/mL. Langkah selanjutnya dilakukan pengambilan 1 mL untuk
ekstrasi DNA dan 1 mL lainnya untuk dihitung konsentrasi bakterinya dengan metode pencawanan (FDA 2001). DNA y a n g diekstrak dari beberapa tingkat pengenceran tersebut kemudian diamplifikasi dengan rt-PCR. Perpotongan kurva amplifikasi yang dihasilkan dengan basement-threshold
menghasilkan nilai CT. Sampel yang dipakai untuk pembuatan kurva standar L.
monocytogenes ditentukan berdasarkan :
a. Sampel yang masih terdeteksi jumlah koloninya pada saat pencawanan b. Sampel yang nilai CT nya di bawah nilai siklus tertinggi yang masih
menunjukkan spesifisitas primer (berdasarkan uji spesifisitas primer)
4
HASIL DAN PEMBAHASAN
Pemilihan Metode Ekstraksi DNA
Salah satu tahap penting dalam analisis DNA dengan metode PCR yaitu tahap ekstraksi DNA. Ekstraksi DNA menjadi titik kritis yang sangat mempengaruhi hasil analisis. Jumlah dan mutu DNA hasil ekstraksi akan mempengaruhi analisis lebih lanjut dengan PCR. Hasil ekstraksi DNA menggunakan tiga metode pada penelitian ini dievaluasi melalui tahap elektroforesis menggunakan gel agarose. Hasil visualisasi DNA dengan gel elektroforesis berupa pita DNA genom dari masing-masing metode ekstraksi (Gambar 4).
12
pita yang terdapat di antara marker 500 bp dan 1000 bp, di antara marker 1000 bp dan 1500 bp, serta di atas 10000 bp.
Keterangan : a. Kontrol
b. DNA hasil isolasi metode fenol:kloroform
c. DNA hasil isolasi metode pemanasan
d. DNA hasil isolasi metode kit komersial
e. DNA ladder 1kb
Gambar 4. Hasil elektroforesis pada tahap ekstraksi DNA Tabel 3. Kemurnian dan konsentrasi DNA
Metode Ekstraksi
Kultur murni Sampel pempek spike
A260/A280 Konsentrasi DNA (ng/µ L)
A260/A280 Konsentrasi DNA (ng/µ L)
Fenol:kloroform 1.24 1857 1.28 1406
Pemanasan 0.40 1027 - -
Kit komersial 0.76 915 0.70 656
DNA dikatakan murni jika rasio diantara kedua nilai absorbansi 260 dan 280 berada di kisaran 1.8 (Nolan et al. 2007). Pengukuran kemurnian DNA (Tabel 3) menunjukkan bahwa DNA yang dihasilkan dari metode fenol:kloroform untuk sampel kultur murni dan sampel pempek spike masih terdapat pengotor lain ditunjukkan oleh nilai rasio A260 dan A280 di bawah 1.8. Pengotor yang diduga menyebabkan hal tersebut diduga adalah protein. Hal ini dapat disebabkan karena kurangnya proses pencucian menggunakan isopropanol. Bettelheim et al. (2013) menjelaskan bahwa isopropanol dapat meningkatkan kemurnian DNA dengan mengoptimalkan proses pembuangan residu protein.
13
Metode kit komersial QIAmp DNA Blood Mini Kit telah digunakan sebelumnya oleh Dauphin et al. (2009), Nur’utami et al. (2011) dan Dibbern et al. (2015). Metode kit komersial oleh Dauphin et al. (2009) yang digunakan untuk bakteri Brucella menghasilkan DNA yang memiliki kemurnian yang kurang baik dimana nilai rasio A260/A280 lebih kecil dari 1.8, sedangkan penelitian Dibbern et al. (2015) pada bakteri S. aureus justru menunjukkan bahwa metode kit QIAmp DNA Blood Mini Kit justru menghasilkan kemurnian DNA yang lebih baik dari metode lainnya pada rasio A260/A280 sebesar 1.76.
Metode yang dikembangkan oleh Nur’utami et al. (2011) dengan modifikasi penggunaan CTAB untuk pelisisan awal menghasilkan kemurnian DNA
Salmonella Typhimurium yang baik sekitar 2.0. Hal ini berbeda dengan hasil penelitian kali ini yang menghasilkan kemurnian DNA di bawah 1.0. Konsentrasi yang dihasilkan metode kit juga lebih rendah dibanding metode fenol:kloroform baik untuk sampel kultur murni maupun sampel pempek spike. Hal ini dimungkinkan karena CTAB yang digunakan sebagai pengganti tissue lysis buffer (buffer ATL) kurang optimal jika digunakan untuk bakteri Gram positif.
Buffer ATL direkomendasikan oleh produsen sebagai buffer yang digunakan dalam pelisisan jaringan sel. Ekstraksi DNA selanjutnya dilakukan dengan metode fenol:kloroform.
Konfirmasi Spesifisitas Primer
Tahapan awal sebelum penentuan spesifisitas primer yaitu pemilihan primer yang sesuai untuk deteksi L. monocytogenes. Penelitian ini menggunakan dua pasang primer berbeda. Masing-masing pasangan primer diuji menggunakan PCR standar. Masing-masing primer dianalisis dengan menggunakan Basic Local Alignment Search Tool (NCBI) dan ditentukan data spesies yang memiliki kecocokan 100% terhadap urutan sekuens primer yang dianalisis (Lampiran 9, 10, 11 dan 12). Untuk mengkonfirmasi bahwa kedua primer tersebut spesifik terhadap L. monocytogenes maka dilakukan pengujian dengan PCR standar dan rt-PCR.
Primer LIM 2/LIMRE digunakan untuk mengamplifikasi gen iap. Gen iap
(invasion associated secreted endopeptidase) merupakan gen yang memegang peranan penting pada viabilitas sel L. monocytogenes. Gen iap memproduksi protein p60 yang mempunyai aktivitas bakteriolitik (Kohler et al. 1991). Gen
iap hanya terdapat pada L. monocytogenes dan tidak terdapat pada bakteri lain yang digunakan sebagai kontrol. Hal ini sesuai dengan hasil BLAST yang menunjukkan bahwa kecocokan 100% dari primer forward (LIM 2) dan reverse
(LIMRE) hanya berasal dari spesies Listeria saja (Lampiran 9 dan 10). Hasil PCR standar dengan primer LIM 2/LIMRE menunjukkan adanya pita (band) yang terbentuk pada hasil elektroforesis produk amplifikasi PCR dengan ukuran di bawah 200 bp pada semua sampel (Gambar 5). Hal ini menunjukkan bahwa kondisi running untuk primer LIM 2/LIMRE belum baik karena masih menghasilkan produk non spesifik.
14
kategori α-hemolitik. Bakteri lain yang digunakan sebagai kontrol juga mempunyai kemampuan hemolitik, antara lain E. coli ATCC 25922 (Snyder dan Carson 2010),
S. aureus ATCC 25923 (Mak et al. 2008) dan C. sakazakii ATCC 29544 (Fakruddin et al. 2014) yang ketiga-tiganya termasuk kategori β-hemolitik. Sedangkan L. plantarum ATCC 8014 tidak mempunyai sifat hemolitik. Hasil PCR standar dengan primer DG69/DG74 menunjukkan adanya pita 635 bp hanya pada DNA L. monocytogenes (Gambar 5). Hal ini sesuai dengan hasil BLAST yang menunjukkan bahwa kecocokan 100% dari primer forward (DG69) dan reverse (DG74) hanya berasal dari spesies Listeria saja (Lampiran 11 dan 12).
Perbandingan hasil kedua primer (Gambar 5) menunjukkan bahwa primer DG69/DG74 lebih cocok digunakan untuk deteksi L. monocytogenes. Selain itu, primer DG69/DG74 lebih banyak dipakai pada penelitian-penelitian sebelumnya untuk deteksi L. monocytogenes dibanding primer LIM 2/LIMRE. Amplikon primer DG69/DG74 berada pada ukuran 635 bp sehingga lebih mudah dibedakan dari produk non spesifik maupun primer dimer.
Keterangan:
Pengujian spesifisitas primer dilakukan untuk menentukan kemampuan primer dalam membedakan DNA bakteri target dengan bakteri lain. Hasil uji spesifisitas primer DG69/DG74 dengan rt-PCR menunjukkan bahwa kondisi
running yang dipakai dapat mendeteksi DNA L. monocytogenes dengan hasil kurva amplikasi yang baik sampai siklus 27 (Gambar 6). Setelah siklus ke-27, kondisi running tersebut tidak spesifik lagi karena dapat menghasilkan produk non-spesifik pada sampel L. plantarum, S. aureus, C. sakazakii, E. coli, dan kontrol tanpa DNA.
15
yang teramplifikasi dan terkuantifikasi sehingga menghasilkan data false-positive (Pestana et al. 2010). Menurut Altshuler (2006), munculnya primer dimer dan produk non-spesifik dapat dikurangi dengan optimasi kondisi running. Oleh karena itu, metode rt-PCR menggunakan SYBR Green biasanya diikuti dengan analisis kurva melting untuk melihat suhu pelelehan (Tm) dari masing-masing sampel.
Keterangan :
a. L. monocytogenes ATCC 7644
b. L. monocytogenes ATCC 13932
c. L. plantarum ATCC 8014 d. S. aureus ATCC 25923 e. C. sakazakii ATCC
29544
f. Kontrol tanpa DNA g. E. coli ATCC 25922
Gambar 6. Kurva amplifikasi uji spesifisitas primer DG69/DG74 dengan rt-PCR Nilai Tm merupakan suhu ketika 50 % DNA untai ganda telah terdenaturasi menjadi DNA untai tunggal (Esco 2009). Hasil analisis nilai Tm dengan primer DG69/DG74 (Tabel 4) menunjukkan bahwa L. monocytogenes dapat dibedakan dari C. sakazakii dan E. coli; sedangkan L. monocytogenes, L. plantarum, dan S. aureus tidak dapat dibedakan karena memiliki Tm yang sama (79 °C).
Tabel 4. Hasil analisis kurva melting
Kode Sampel DNA Nilai Tm ( °C)
a. L. monocytogenes ATCC 7644 79.0 b. L. monocytogenes ATCC 13932 79.0
c. L. plantarum ATCC 8014 79.0
d. S. aureus ATCC 25923 79.0
e. C. sakazakii ATCC 29544 75.5
f. Kontrol tanpa DNA 76.5
g. E. coli ATCC 25922 73.5
Analisis nilai Tm setelah tahap amplifikasi merupakan metode alternatif dari analisis gel elektroforesis untuk penentuan kemurnian hasil amplikon (Dwight et al. 2011). Oleh karena itu, analisis nilai Tm dapat mendeteksi adanya
16
Penentuan Limit Deteksi DNA L. monocytogenes
Limit deteksi merupakan nilai pengenceran tertinggi yang masih dapat teramplifikasi yang menunjukkan sensitivitas deteksi suatu metode. Limit deteksi rt-PCR menggunakan primer DG69/DG74 ditentukan dari konsentrasi saat nilai CT = 27 (berdasarkan tahap konfirmasi spesifisitas primer). Hasil analisis limit deteksi pada sampel kultur murni dan sampel pempek spike dengan rt-PCR
menghasilkan data nilai CT dan nilai Tm dari masing-masing konsentrasi
L. monocytogenes (Tabel 5).
Tabel 5. Hasil analisis rt-PCR DNA L. monocytogenes
Kultur murni Sampel pempek spike
Konsentrasi menunjukkan bahwa semakin besar konsentrasi L. monocytogenes maka nilai CT akan semakin kecil. Hal ini terjadi karena konsentrasi bakteri yang semakin tinggi akan menghasilkan DNA terekstrak yang lebih banyak, sehingga semakin tinggi DNA yang terekstrak maka semakin cepat mencapai garis basement-threshold
saat proses amplifikasi berjalan (Applied Biosystems 2015). Hasil analisis nilai Tm pada kultur murni dan sampel pempek spike yang berada pada nilai 79 ± 0.5 menunjukkan bahwa bahwa kondisi running yang diterapkan telah spesifik dan tidak terjadi mispriming.
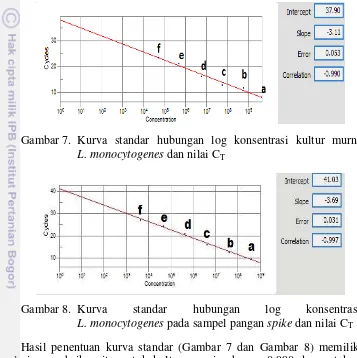
Hasil analisis deteksi L. monocytogenes dengan rt-PCR (Tabel 5) dapat dibuat kurva standar hubungan nilai CT dan konsentrasi L. monocytogenes. Kurva standar yang dihasilkan pada kultur murni (Gambar 7) memiliki persamaan:
CT : Nilai CT
C : Nilai Log Konsentrasi bakteri (CFU/mL)
Sedangkan kurva standar yang dihasilkan pada sampel pempek spike (Gambar 8) memiliki persamaan :
CT : Nilai CT
C : Nilai log konsentrasi bakteri pada SPS
Nilai konstanta persamaan kurva standar kultur murni (37.9) lebih kecil dibanding nilai konstanta SPS (41.03). Sedangkan nilai koefisien persamaan
CT = 37.9 – 3.11 C
17
kurva standar kultur murni (3.11) sedikit lebih rendah dibanding nilai koefisien SPS (3.69). Ini menunjukkan bahwa pada konsentrasi L. monocytogenes yang rendah, sampel kultur murni lebih dulu teramplifikasi dibanding SPS. Sedangkan pada konsentrasi L. monocytogenes yang semakin tinggi, SPS justru lebih cepat teramplifikasi. Hal ini menunjukkan bahwa matriks pangan pempek mempengaruhi hasil amplifikasi di rt-PCR. Menurut Dadkhah et al. (2012) dan Camma et al. (2012), adanya komponen lain dari pangan dapat menjadi sebab terjadinya perbedaan hasil amplifikasi, baik yang disebabkan karena pengaruh saat ekstraksi maupun pengaruh saat proses amplifikasi rt-PCR.
Gambar 7. Kurva standar hubungan log konsentrasi kultur murni
L. monocytogenes dan nilai CT
Gambar 8. Kurva standar hubungan log konsentrasi
L. monocytogenes padasampel pangan spike dan nilai CT
Hasil penentuan kurva standar (Gambar 7 dan Gambar 8) memiliki nilai korelasi yang baik yaitu untuk kultur murni sebesar -0.990 dan untuk sampel pempek spike sebesar -0.997. Hasil nilai korelasi mendekati ± 1.0 menunjukkan hubungan yang sangat kuat antara konsentrasi L. monocytogenes terhadap nilai CT yang dihasilkan (Taylor 1990). Nilai korelasi yang negatif menunjukkan bahwa semakin besar nilai pada sumbu x (konsentrasi L. monocytogenes) maka semakin kecil nilai pada sumbu y (nilai CT).
Persamaan kurva standar yang telah dihasilkan dapat digunakan untuk kuantifikasi sampel yang belum diketahui konsentrasi L. monocytogenes-nya. Selain itu, penentuan limit deteksi dari sampel kultur murni dan sampel pempek
18
didapat dari nilai C dari persamaan kurva standar tersebut. Hasil limit deteksi pada sampel kultur murni dan sampel pempek spike masing-masing adalah 3.2 x 103 CFU/mL dan 6.3 x 103 CFU/g. Hasil limit deteksi ini mendekati hasil dari metode yang digunakan oleh Dadkhah et al. (2012) yang memiliki limit deteksi pada sampel kultur murni sebesar 1.2 x 103 CFU/mL dan limit deteksi pada sampel susu sebesar 2.1 x 103 CFU/mL.
Hasil penentuan limit deteksi untuk kultur murni dan sampel pempek spike
memiliki kepekaan 3 kali lebih rendah dibanding hasil penelitian Dadkhah et al.
(2012). Salah satu penyebabnya yaitu nilai kemurnian DNA hasil ekstraksi yang tidak baik (Siebert 1999). Selain itu, perbedaan kehomogenan sampel pempek
spike yang lebih rendah dibanding sampel susu spike pada penelitian Dadkhah et al. (2012) merupakan penyebab menurunnya sensitivitas deteksi penelitian ini.
Aplikasi penerapan deteksi sampel dengan rt-PCR antara kultur murni dan sampel pempek spike akan berbeda ketika nilai CT berada di atas 27, sedangkan
jika nilai CT di bawah angka 27 dapat dinyatakan bahwa sampel positif terdapat
L. monocytogenes (Tabel 6). Sampel kultur murni yang memiliki nilai CT di atas 27 dapat dinyatakan negatif L. monocytogenes berdasarkan hasil konfirmasi spesifisitas sebelumnya, sedangkan untuk sampel pempek spike yang memiliki nilai CT di atas 27 masih perlu adanya uji rt-PCR lanjut dengan perlakuan
enrichment sampel. Enrichment sampel pempek spike tersebut dimaksudkan untuk meningkatkan jumlah konsentrasi awal L. monocytogenes sehingga memungkinkan pengujian kualitatif dengan rt-PCR pada sampel spike tersebut. Tabel 6. Hubungan nilai CT terhadap sampel yang diuji
Nilai CT
Kultur murni Sampel pempek spike
Status Status Langkah lanjut
≤ 27 Positif L. monocytogenes Positif L. monocytogenes Tidak ada
> 27 Negatif Perlu uji lanjut Uji rt-PCR dengan
enrichment sampel
SIMPULAN
Metode ekstraksi yang disarankan untuk analisis L. monocytogenes adalah metode fenol:kloroform dengan hasil DNA genom yang lebih tinggi dibanding metode pemanasan dan metode kit komersial. Primer yang sesuai untuk analisis
L. monocytogenes yaitu primer DG69/DG74 dengan hasil kespesifikan lebih baik dibanding primer LMS 2/LIMRE. Kondisi running yang dipakai mendeteksi DNA L. monocytogenes menghasilkan kurva amplikasi yang baik sampai siklus ke-27. Deteksi DNA kultur murni L. monocytogenes menghasilkan kurva standar CT = 37.9 – 3.11 C dengan sensitivitas 3.2 x 103 CFU/mL, sedangkan pada SPS
19
SARAN
Pengembangan metode ekstraksi perlu dilakukan untuk mendapatkan metode ekstraksi yang memiliki kemurnian dan kualitas DNA yang lebih baik. Hasil pengembangan metode menunjukkan adanya perbedaan kurva standar antara kultur murni dan sampel pangan, sehingga perlu ditentukan kurva standar baru untuk penelitian tentang deteksi L. monocytogenes pada sampel pangan yang berbeda. Selain itu, pengembangan rt-PCR juga perlu dilakukan untuk deteksi
L. monocytogenes pada jajanan pempek dengan metode lain seperti TaqMan®.
DAFTAR PUSTAKA
Acciari VA, Torresi M, Migliorati G, Giannatale ED, Semprini P, Prencipe P. 2011. Characterization of L. monocytogenes strains isolated from soft and semi-soft cheese sampled in a region of Italy. Veterinaria Italiana. 47(1): 15-23 Altshuler ML. 2006. PCR Troubleshooting: The Essential Guide. Caister
Academic Press. Moscow
Applied Biosystems (AB). 2015. Essentials of Real Time PCR. US
Aurora R, Prakash A, Prakash S, Rawool DB, Barbuddhe SB. 2008. Comparison of PI-PLC based assay and PCR along with in vivo pathogenicity test for rapid detection of pathogenic L. monocytogenes. Food Control. 19: 641-647
Bettelheim FA, Brown WH, Campbell MK, Farrell SO, Torres OJ. 2013.
Introduction to General, Organic, and Biochemistry. Brooks/Cole. Australia Brisson M, Tan L, Park R, Hamby K. 2000. Identification of nonspecific products
using melt-curve analysis on the iCycler iQ™ detection system. Bio-Rad antimicrobials for the control of Listeria monocytogenes in foods. Formatex. 1111-1123
Coral D - P, Rosales-Rivera A, Rodriguez-Garcia ME. 2009. Determination of the gelatinization temperature of starch presented in maize flours. Journal of Physics. 167: 1-5
Choi WS, and Hong CH. 2003. Rapid enumeration of Listeria monocytogenes in milk using competitive PCR. Int. J. Food Microbiol. 84: 79-85
Dadkhah H, Bassami MR, Hashemi S, Shahraz F, Hosseini H, Karatzas KAG, Khaksar R. 2012. Evaluation and comparison of SYBR Green I real-time PCR dan TaqMan real-time PCR methods for quantitative assay of Listeria monocytogenes in nutrient broth and milk. African Journal of Microbiology Research. 69: 1908-1917
Dharmaraj, S. 2015. RT-PCR: The Basics. http://www.lifetechnologies.com [12 Juni 2015]
Dauphin LA, Hutchins RJ, Bost LA, Bowen MD. 2009. Evaluation of automated and manual commercial DNA extraction methods for recovery of Brucella
20
Dibbern AG, Botaro BG, Viziack MP, Silva LFP, Santos MV. 2015. Evaluation of methods of DNA extraction from Staphylococcus aureus in milk for use in real-time PCR. Genet. Mol. Res. 14(1): 227-233
Dwight Z, Palais R, Wittwer CT. 2011. uMELT: prediction of high-resolution melting curves and dynamic melting profiles of PCR products in a rich web application. Bioinformatics. 27(7):1019–1020
Esco. 2009. User Manual Swift Spectrum 48 Fluorescence Quantitative PCR Detection System. Esco Healthcare Pte. Ltd.
Erali M, Wittwer CT. 2010. High resolution melting analysis for gene scanning. Methods. 50: 250-261
Fakruddin M, Rahaman M, Ahmed MM, Hoque MM. 2014. Stress tolerant virulent strains of Cronobacter sakazakii from food. Biological Research. 47(1): 63
Food and Drug Admission (FDA). 2001. Bacteriological Analytical Manual. Food and Drug Administration.
Hein I, Lehner A, Rieck P, Klein K, Brandl E, Wagner M. 2001. Comparison of different approaches to quantify Staphylococcus aureus cells by real-time quantitative pcr and application of this technique for examination of cheese. Applied and Environmental Microbiology. 67(7): 3122–3126.
Jamali H, Chai LC, Thong KL. 2013. Detection and isolation of Listeria monocytogenes in ready to eat foods with various selective culture agar. Food Control. 32: 19-24
Jenie BSL, Kusumaningrum HD, Nurjanah S. 2013. Deteksi bakteri patogen dan fermentatif dari pangan menggunakan real-time polymerase chain reaction. Prosiding Seminar Hasil-Hasil PPM IPB. 1: 292- 308
Kohler S, Bubert A, Vogel M, Goebel W. 1991. Expression of the iap gene coding for protein p60 of Listeria monocytogenes is controlled on the posttranscriptional level. Journal of Bacteriology. 173: 4668-4674
Kramarenko T, Roasto M, Meremae K, Kuningas M, Poltsama P, Elias T. 2013.
Lysteria monocytogenes prevalence and serotype diversity in various foods. Food Control. 30: 24-29
Kunkel D. 2013. Listeria monocytogenes - rod prokaryote (bacterium). http://www.denniskunkel.com/DK/Bacteria/20229A.html [12 November 2013] Mak P, Maszewska A, Rozalska M. 2008. The amino acid sequences and
activities of synergistic hemolysins from Staphylococcus cohnii. FEMS Microbiology Letters. 287: 230-235
Marian MN, Aminah SMS, Zuraini MI, Son R, Maimunah M, Lee YH, Wong WC. Elexson N. 2012. MPN-PCR detection and antimicrobial resistance of
Listeria monocytogenes isolated from raw and ready to eat foods in Malaysia. Food Control. 28: 309-314
Meyer C, Ahomaa MF, Sperner B, Martlbauer E. 2011. Detection of Listeria monocytogenes in pork and beef using the VIDAS LMO2 automated enzyme linked immunoassay method. Meat Science. 88: 594-596
Montgomery JL, Sanford LN, Wittwer CT. 2010. High-resolution DNA melting analysis in clinical research and diagnostics. Expert Rev Mol Diagn. 10: 219-240
21
ready-to-eat and processed vegetable foods by culture and DVC-FISH. Food Control. 27: 374-379
Nolan T, Mueller R, Bustin S. 2007. QPCR: target preparation. In: Mackay IM (ed.). Real-time PCR in Microbiology From Diagnosis to Characterization. UK: Caister Academik Press
Nur’utami DA, Nurwitri CC, Rahayu WP, Panggabean RIL, Yuliangsih S. 2011.
Metode analisis isolasi dan identifikasi Salmonella Typhimurium pada susu dengan metode real-time PCR (Polymerase Chain Reaction) [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Ohk SH, Bhunia AK. 2013. Multiplex fiber optic biosensor for detection of
Listeria monocytogenes, Escherichia coli O157:H7 and Salmonella enteritica
from ready to eat meat samples. Food Microbiology. 33: 166-171
Pestana EA, Belak S, Diallo A, Crowther JR, Viljoen GJ. 2010. Early, Rapid, and Sensitive Veterinary Molecular Diagnostics Real-Time PCR Application. Springer. Dordrecht
Pochop J, Kacaniova M, Hleba L, Petrova J, Lopasovsky L, Pavelkova A, Bobkova A. 2013. Real-time PCR detection of Listeria monocytogenes in Food samples of animal origin. International Journal of Microbiology, Biotechnology And Food Sciences. 141: 1550-1558
Rahayu WP, Panggabean RIL, Yuliangsih S, Pusparini N, Khotimah K. 2009. Comparison of three extraction methods for identification Bacillus cereus and
Staphylococcus aureus in fried rice. Research Center for Drug and Food. National Agency of Drug and Food Control.
Rawool DB, Malik SVS, Shakuntala I, Sahare AM, Barbuddhe SB. Detection of multiple virulence-associated genes in Listeria monocytogenes isolated from bovine mastitis cases. International Journal of Microbiology. 113: 201-207 Sambrook J and Russel DW. 2001. Molecular Cloning: A Laboratory Manual.
Cold Spring Harbor Laboratory Press, New York.
Schuppler M, Loessner MJ. 2010. The opportunistic pathogen Listeria monocytogenes: pathogenicity and interaction with mucosal immune system. Int. Journal of Inflammation. 2010: 1-12
Siebert PD. 1999. Quantitative RT-PCR. In: Kochanowski B, Reischl U (eds.). Methods in Molecular Medicine: Quantitative PCR Protocols. Humana Press. New Jersey
Snyder RO, Carson KL. 2010. New viral gene vector reference material. ATCC Connection. 30(1): 3-5
Taylor R. 1990. Interpretation of the Correlation Coefficient: A Basic Review. Journal of Diagnostic Medical Sonography. 6: 35-39
Todd ECD, Notermans S. 2011. Surveillance of listeriosis and its causative pathogen, Listeria monocytogenes. Food Control. 22: 1484–1490
Ye K, Zhang Q, Jiang Y, Xu X, Cao J, Zhou G. 2012. Rapid detection of viable
22
LAMPIRAN
Lampiran 1. Komposisi dalam PCR standar dengan Primer LIM 2 / LIMRE
Bahan Jumlah
Maxima Hotstart Master Mix 10 µL
Water (Nuclease Free) 13 µL
Template DNA 2 µL
Primer forward LIM 2 (CTA AAG CGG GAA TCT CCC TT) 250 nM Primer reverse LIMRE (CCA TTG TCT TGC GCG TTA AT) 250 nM Lampiran 2. Kondisi running PCR standar dengan Primer LIM 2 / LIMRE
Tahap Suhu ( °C ) Waktu Jumlah Siklus
Denaturasi awal 95 5 menit *) 1
Tahap amplifikasi : 40
Denaturasi 95 15 detik
Annealing 54 1 menit
Extension 72 30 detik
Entension Akhir 72 5 menit 1
Lampiran 3. Komposisi bahan dalam PCR standar dengan Primer DG69 / DG74
Bahan Jumlah
DreamTaq Green PCR Master Mix 10 µL
Water (Nuclease Free) 4 µL
Template DNA 2 µL
primer forward DG69 (GTGCCGCCAAGAAAAGGTTA) 500 nM* primer reverse DG74 (CGCCACACTTGAGATAT) 500 nM* Lampiran 4. Kondisi running PCR standar dengan Primer DG69 / DG74
Tahap Suhu ( °C ) Waktu Jumlah Siklus
Denaturasi awal 94 5 menit 1
Tahap amplifikasi : 30
Denaturasi 94 45 detik
Annealing 55 45 detik
Extension 72 45 detik
23
Lampiran 5. Komposisi bahan untuk rt-PCR dengan Primer LIM 2 / LIMRE
Bahan Jumlah
DyNamo ColorFlash Probe qPCR Kit 10 µL
Water (Nuclease Free) 13 µL
Template DNA 2 µL
Primer forward LIM 2 (CTA AAG CGG GAA TCT CCC TT) 250 nM Primer reverse LIMRE (CCA TTG TCT TGC GCG TTA AT) 250 nM
Lampiran 6. Kondisi running rt-PCR dengan Primer LIM 2 / LIMRE
Tahap Suhu ( °C ) Waktu Jumlah Siklus
Denaturasi awal 95 5 menit *) 1
Tahap amplifikasi : 40
Denaturasi 95 15 detik
Annealing 57 1 menit
Extension 72 30 detik
Entension Akhir 72 5 menit 1
Lampiran 7. Komposisi bahan untuk rt-PCR dengan Primer DG69 / DG74
Bahan Jumlah
DyNAmo ColorFlash SYBR Green qPCR Kit 10 µL
Water (Nuclease Free) 4 µL
Template DNA 2 µL
primer forward DG69 (GTGCCGCCAAGAAAAGGTTA) 500 nM* primer reverse DG74 (CGCCACACTTGAGATAT) 500 nM*
Lampiran 8. Kondisi running rt-PCR dengan Primer DG69 / DG74
Tahap Suhu ( °C ) Waktu Jumlah Siklus
Denaturasi awal 94 5 menit 1
Tahap amplifikasi : 30
Denaturasi 94 45 detik
Annealing 55 45 detik
Extension 72 45 detik
Entension Akhir 72 7 menit 1
Tahap Melting 72 – 94 ( kenaikan suhu
tiap 0.5°C)
10 detik (tiap kenaikan suhu
0.5°C)
24
Lampiran 9. Analisis BLAST primer LIM 2
No Deskripsi Kecocokan
sekuens 1 Dendroctonus ponderosae Seq01042496, whole genome
shotgun sequence
100% 2 Human DNA sequence from clone RP11-49F20 on
chromosome 13, complete sequence
100%
3 L. monocytogenes iap gene for protein p60 100%
150 Listeria monocytogenes WSLC1042, complete genome 100% 151 Triticum aestivum chromosome 3B, genomic scaffold, cultivar
Chinese Spring
100% Keterangan : Data sekuens DNA no. 4 – 149 berasal dari genus Listeria (data
tidak ditampilkan)
Lampiran 10. Analisis BLAST primer LIMRE
No Deskripsi Kecocokan
sekuens
1 L. monocytogenes iap gene for protein p60 100%
166 Listeria welshimeri strain TUMLis35 invasion associated protein p60 (iap) gene, complete cds
100%
167 Marinovum algicola DG 898, complete genome 100%
168 Schistocephalus solidus genome assembly
S_solidus_NST_G2, scaffold SSLN_scaffold0001343
100% 169 Vibrio vulnificus MO6-24/O chromosome I, complete
sequence
100% Keterangan : Data sekuens DNA no. 2 – 165 berasal dari genus Listeria (data
25
Lampiran 11. Analisis BLAST primer DG69
No Deskripsi Kecocokan
sekuens 1 L. monocytogenes lisA gene for listeriolysin and part of prfA
and mpl genes
100%
296 Listeria monocytogenes WSLC1042, complete genome 100% 297 Triticum aestivum chromosome 3B, genomic scaffold, cultivar
Chinese Spring
100% 298 Vitis vinifera, whole genome shotgun sequence, contig
VV78X198057.23, clone ENTAV 115
100% Keterangan : Data sekuens DNA no. 2 – 295 berasal dari genus Listeria (data
tidak ditampilkan)
Lampiran 12. Analisis BLAST primer DG74
No Deskripsi Kecocokan
sekuens
1 Drosophila busckii chromosome 3R sequence 100%
2 L.monocytogenes listeriolysin O (hlyA) gene, complete cds 100%
188 Listeria monocytogenes WSLC1001, complete genome 100% 189 Nippostrongylus brasiliensis genome assembly
N_brasiliensis_RM07_v1_5_4, scaffold NBR_scaffold0001085
100%
190 Oryza sativa Japonica Group DNA, chromosome 10, cultivar: Nipponbare, complete sequence
100% 191 Phanerochaete carnosa HHB-10118-sp hypothetical protein
(PHACADRAFT_250592), partial mRNA
100% 192 PREDICTED: Esox lucius galactosidase, beta 1 (glb1), mRNA 100% 193 Solanum lycopersicum chromosome ch04, complete genome 100% 194 Solanum pennellii chromosome ch04, complete genome 100% 195 Synthetic construct hemolysin BL-listeriolysin O-enterotoxin
B fusion protein gene, partial cds
100% 196 Synthetic construct lytic 1/listeriolysin O fusion protein gene,
partial cds
100% 197 TPA: Oryzias latipes strain Hd-rR, complete genome
assembly, chromosome 16
100% Keterangan : Data sekuens DNA no. 3 – 187 berasal dari genus Listeria (data
26
RIWAYAT HIDUP
Penulis dilahirkan di Surakarta pada tanggal 17 Desember 1986 sebagai anak ke dua dari lima bersaudara dari pasangan Bapak Ngimri dan Ibu Pasyektini. Penulis lulus dari SMU Al-Islam 1 Surakarta pada tahun 2004 dan melanjutkan studi pada tahun yang sama di Institut Petanian Bogor (IPB) melalui jalur undangan. Penulis meraih gelar Sarjana Teknologi Pertanian pada program studi Teknologi Pangan dan Gizi, Fakultas Teknologi Pertanian, Institut Pertanian Bogor pada tahun 2009. Pada tahun 2009 penulis bekerja sebagai programmer
SAP di PT Jogja Global Technology. Kemudian pada tahun 2011, penulis bekerja sebagai dosen tetap di Politeknik Gorontalo. Pada tahun 2012, penulis memiliki kesempatan untuk melanjutkan pendidikan S2 pada program studi Ilmu Pangan, Sekolah Pascasarjana, IPB. Selama masa studi penulis meraih beasiswa dari Tanoto Foundation tahun ajaran 2013/2014. Penulis mendapatkan dana bantuan penelitian Direktorat Jenderal Pendidikan Tinggi melalui Program Hibah Kompetensi atas nama Prof. Dr. Winiati P. Rahayu. Sebagai salah satu syarat untuk mendapatkan gelar Magister Sains, penelitian ini telah diterima untuk dipublikasikan dalam Malaysian Journal of Microbiology berjudul “Detection of
Listeria monocytogenes in Indigenous Snack Using Real-time Polymerase Chain