ISOLATION AND IDENTIFICATION ANALYSIS METHOD FOR
Salmonella Typhimurium IN MILK USING REAL-TIME PCR (Polymerase Chain Reaction)
Dwi Aryanti Nur’utami1, C. C. Nurwitri1, Winiati P. Rahayu1, Riolina Ida L. Panggabean2, and Suci Yuliangsih2 1
Department of Food Science and Technology, Faculty of Agricultural Engineering and Technology, Bogor Agricultural University, IPB Darmaga
Campus, PO Box 220, Bogor, West Java, Indonesia. 2
National Agency of Drug and Food Control Republic of Indonesia, Indonesia Phone: +62 857 23 521259, e-mail: [email protected]
ABSTRACT
Salmonella Typhimurium is one of pathogenic bacteria that cause foodborne disease. Conventional culture methods which is traditionally been considered as a ‘‘gold standards’’ are a common method to identified this pathogen. However, conventional culture methods are labor-intensive and time-consuming. This study aimed to obtain a rapid and precise analytical method for Salmonella Typhimurium, to compare boiling method and commercial kit method using QIAamp® DNA Blood Mini Kit for extraction of DNA, identify and quantify Salmonella Typhimurium on sterilized milk using real-time PCR. Stages of this research are doing enrichment in pure culture and spiked sample, isolating the DNA of Salmonella Typhimurium by boiling method and commercial kit method, and then running a real-time PCR for determining specificity of the primers, determining primers concentration and doing quantification of Salmonella Typhimurium in sterilized milk. Commercial kit method produces DNA template purer than boiling method. So it affects their efficiency value. Commercial kit method assay has better efficiency than boiling method assay. It is 100% for commercial kit method and 386,8% for boiling method. The concentration of primers which is used in real-time PCR assay is 0.125 μM and its primers are specific for Salmonella Typhimurium. The result of Salmonella Typhimurium quantification in sterilize milk are not the same as quantification result from conventional method. But it has the same result with concentration value of Salmonella Typhimurium that is added to sterilize milk which is counted by Petroff-Hausser.
I.
PENDAHULUAN
A.
Latar Belakang
Pangan merupakan kebutuhan pokok bagi setiap individu, karena di dalamnya terkandung senyawa-senyawa yang sangat diperlukan untuk memulihkan dan memperbaiki jaringan tubuh yang rusak, mengatur proses di dalam tubuh, dan menghasilkan energi untuk kepentingan berbagai kegiatan dalam kehidupan. Komposisi umum bahan pangan terdiri atas protein, karbohidrat, lemak, vitamin dan mineral. Bahan pangan dengan komposisi tersebut merupakan media yang baik untuk pertumbuhan mikroba.
Populasi mikroba khususnya mikroba patogen dapat meningkat pada pangan yang dikonsumsi, sehingga dapat menimbulkan berbagai permasalahan salah satunya adalah Kejadian Luar Biasa (KLB) keracunan pangan (foodborne disease) yang tidak jarang dapat menimbulkan kematian (Supardi dan Sukamto 1999). Salah satu penyebab KLB keracunan pangan adalah adanya cemaran biologis mikroba patogen dari jenis Salmonella. Selama tiga tahun berturut-turut Salmonella dijumpai sebagai penyebab keracunan pangan di Indonesia dan kemungkinan terjadinya berkisar antara 12,5 hingga 25,0% dari cemaran mikroba (Badan POM RI 2008, 2009, 2010).
Setiap individu berhak untuk memperoleh pangan yang senantiasa tersedia setiap waktu, bergizi, dan aman untuk dikonsumsi. Pangan yang aman dikonsumsi merupakan pangan yang bebas (di bawah toleransi maksimum yang dipersyaratkan) dari cemaran berbahaya seperti cemaran biologis, kimia, dan benda asing yang dapat mengganggu, merugikan, dan membahayakan kesehatan manusia (Fardiaz 1994). Oleh karena itu, untuk mengetahui tingkat cemaran suatu pangan, khususnya cemaran biologis maka perlu dilakukan suatu pengujian baik kualitatif maupun kuantitatif, namun pengujian yang biasa digunakan untuk mengidentifikasi mikroba patogen adalah dengan metode konvensional/tradisional yang bergantung pada identifikasi bakteriologikal dan serologikal yang melelahkan dimana membutuhkan waktu empat sampai tujuh hari untuk memperoleh uji yang positif yang bergantung pada konfirmasi secara biokimia dan serologikal (Elizaquível et al. 2010).
Penggunaan metode uji yang lebih cepat menjadi hal yang menarik untuk dikembangkan dan dipelajari khususnya untuk pengawasan keamanan pangan. Diantara banyak metode cepat yang sedang dikembangkan untuk mendeteksi dan mengidentifikasi Salmonella dan mikroba patogen lainnya, Polymerase Chain Reaction (PCR) telah sering dipelajari selama dekade terakhir karena selain cepat dan mudah, metode ini juga sangat spesifik dan sensitif (Abubakar et al. 2007). Baru-baru ini, metode real-time PCR banyak digunakan karena hasilnya dapat langsung dimonitor secara real-time (diamati seketika) dan data yang dihasilkan dapat dianalisis secara kuantitatif (Chen et al. 2009).
Pusat Riset Obat dan Makanan (PROM) Badan POM RI sedang mengembangkan metode dengan
real-time PCR. Pengembangan metode ini penting dilakukan untuk memperoleh metode yang cocok (efektif, spesifik, dan sensitif) bagi pangan yang memiliki komposisi beragam yang bisa menghambat pengidentifikasian Salmonella Typhimurium. Pengujian dilakukan dengan menggunakan bakteri uji
Salmonella Typhimurium sebagai kontrol positif dan Shigella sonnei sebagai kontrol negatif di dalam sampel pangan susu UHT dengan gen target adalah InvA. Gen target InvA bertanggung jawab terhadap proses invasi dari sel epitel dan lokus gen InvA diketahui terdistribusi luas pada berbagai strain
Salmonella (Bohaychuk et al. 2006).
B.
Tujuan Penelitian
menggunakan metode pendidihan dengan metode menggunakan kit komersial QIAamp® DNA Blood Mini Kit, (3) mengidentifikasi dan mengkuantifikasi Salmonella Typhimurium pada susu UHT yang berbasiskan amplifikasi DNA dengan menggunakan real-time PCR.
C.
Manfaat Penelitian
Manfaat dari hasil penelitian ini adalah memberikan informasi awal mengenai metode cepat, spesifik, dan efisien untuk mengidentifikasi Salmonella Typhimurium pada sampel pangan susu UHT yang berbasiskan amplifikasi DNA dengan menggunakan real-time PCR dengan gen target InvA
3
I.
TINJAUAN PUSTAKA
A.
Keracunan Pangan
Secara umum bahaya yang timbul dari pangan disebut foodborne disease atau sering disebut keracunan pangan. Menurut World Health Organization (WHO), kejadian luar biasa (KLB) keracunan pangan (foodborne disease outbreak) didefinisikan sebagai suatu kejadian dimana terdapat dua orang atau lebih yang menderita sakit setelah mengkonsumsi pangan yang secara epidemiologi terbukti sebagai sumber penularan (Sparringa 2002). Foodborne disease adalah penyakit yang disebabkan infeksi atau intoksikasi akibat mengkonsumsi makanan, minuman atau air yang telah terkontaminasi (Sharp dan Reilly 2000).
Penyakit yang timbul bila seseorang mengkonsumsi suatu pangan dapat disebabkan oleh dua hal, yaitu pertama pangan tersebut mungkin mengandung komponen beracun (komponen anorganik seperti sianida, gosipol, dsb). Kedua, pangan mengandung mikroba dalam jumlah yang cukup untuk dapat menimbulkan gejala sakit. Berdasarkan kedua hal tersebut, penyakit yang ditimbulkan oleh pangan dapat digolongkan dalam dua kelompok besar menurut penyebabnya yaitu (1) keracunan, dan (2) infeksi mikroba.
Istilah keracunan pangan digunakan secara umum untuk menyatakan semua gejala penyakit yang ditimbulkan sebagai akibat dari mengkonsumsi suatu pangan, baik penyakit tersebut disebabkan oleh toksin maupun oleh mikroba penyebab infeksi yang terdapat dalam pangan tersebut. Selain itu, keracunan pangan juga dapat terjadi karena tertelannya toksin yang merupakan hasil metabolisme sel-sel mikroba tertentu. Gejala-gejala keracunan karena toksin tersebut diatas disebut intoksikasi. Gambaran penyakit yang dapat disebarkan melalui pangan adalah sebagai berikut ini (Gambar 1.).
Gambar 1. Skema penyakit yang disebarkan oleh pangan (foodborne disease)
Infeksi mikroba adalah tertelannya atau masuknya mikroba ke dalam tubuh, kemudian dapat menembus sistem pertahanan tubuh dan hidup serta berkembang biak di dalam tubuh. Dengan kata
Infeksi
Enterotoksigenik Invasif
Tumbuh dan Lisis Sporulasi
Mukosa usus Sistemik (perut) Tenunan lainnya Hati Otot Menggangu metabolisme karbohidrat Neurotoksin Toksin bakteri Mikotoksin Keracunan
Racun anorganik Intoksikasi
Toksin tanaman Toksin hewan Toksin mikroorganisme Toksin algae Enterotoksin
lain infeksi merupakan proses ketika mikroba yang patogen memasuki tubuh inangnya, mengadakan invasi, berkembang biak di dalam tubuh inang, dan menimbulkan penyakit. Dalam menghadapi adanya infeksi mikroba dan hasil-hasil metabolitnya, tubuh mengadakan suatu reaksi perlawanan, yang ditandai oleh adanya gejala-gejala demam yang dialami oleh penderita penyakit. Hal ini merupakan salah satu gejala penyakit yang membedakan antara intoksikasi dengan infeksi mikroba, dimana penderita intoksikasi biasanya tidak mengalami gejala demam.
Kemampuan suatu mikroba dalam menimbulkan penyakit biasanya dinyatakan dengan istilah virulen. Mikroba yang terdapat dalam jumlah yang sedikit tetapi sudah dapat menimbulkan gejala infeksi atau penyakit, dikatakan mempunyai daya virulensi yang tinggi. Sedangkan mikroba yang menyebabkan gejala penyakit yang ringan, atau yang harus terdapat dalam jumlah yang banyak untuk dapat memulai infeksi atau menimbulkan penyakit, dikatakan mempunyai daya virulensi yang rendah. Sifat virulensi juga dapat diartikan sebagai keseluruhan sifat patogenik suatu mikroba yang merupakan perpaduan dari tiga sifat kemampuan mikroba yaitu: (1) daya infeksi atau kemampuannya untuk memulai suatu infeksi di dalam tubuh inangnya, (2) daya invasif atau kemampuan suatu mikroba untuk menembus ke jaringan-jaringan yang lebih dalam, (3) daya patogenik atau kemampuan suatu mikroba untuk merusak sel-sel jaringan tubuh (Supardi dan Sukamto 1999).
Mikroba penyebab infeksi yang tumbuh pada pangan dapat dibedakan menjadi dua kelompok yaitu: (1) mikroba patogen yang pertumbuhannya tidak distimulir oleh pangan tempat mikroba tersebut hidup, dalam hal ini pangan hanya sebagai perantara (pembawa). Misalnya mikroba patogen yang menyebabkan hepatitis. (2) Mikroba patogen yang pertumbuhannya distimulir oleh pangan tempat tumbuhnya sehingga jumlahnya akan bertambah banyak. Misalnya Salmonella sp., EPEC, dan
Vibrio parahaemolyticus.
Mikroba lain yang menyebabkan infeksi antara lain: Clostridium perfringens, Bacillus cereus, Shigella sp., Yersinia enterocolica dan sebagainya. Clostridium perfringens dan Bacillus cereus
adalah mikroba yang juga dapat memproduksi enterotoksin (bersifat enterotoksigenik), sehingga kadang-kadang digolongkan ke dalam kelompok mikroba penyebab intoksikasi. Clostridium perfringens tipe A menghasilkan enterotoksin yang akan dilepaskan ke luar sel sewaktu terjadi sporulasi di dalam saluran pencernaan atau di dalam pangan. Bacillus cereus juga menghasilkan enterotoksin, tetapi toksin tersebut akan dilepaskan ke luar sel sewaktu mengalami lisis atau pecah di dalam saluran pencernaan atau di dalam pangan.
Jika toksin dan kedua mikroba itu dilepaskan oleh sel di dalam pangan dan pangan tersebut tertelan masuk ke dalam tubuh, gejala yang ditimbulkannya disebut sebagai gejala intoksikasi. Meskipun demikian, menunjukkan bahwa penyakit yang disebabkan oleh kedua mikroba tersebut baru timbul jika mikroba yang masih hidup tertelan dalam jumlah cukup tinggi. Hal ini menunjukkan bahwa kondisi yang terbaik bagi kedua mikroba tersebut untuk melepaskan toksinnya adalah di dalam saluran pencernaan. Oleh karena itu, kedua mikroba tersebut dapat digolongkan ke dalam kelompok mikroba penyebab infeksi.
Toksin mikroba dapat dibedakan menjadi dua kelompok diantaranya (1) eksotoksin yaitu toksin yang disintesis di dalam sel mikroba, kemudian dikeluarkan ke substrat di sekelilingnya, dan (2) endotoksin yaitu toksin yang disintesis di dalam sel mikroba, dan baru bersifat toksik bila sel mengalami lisis. Eksotoksin yang dihasilkan oleh mikroba biasanya bekerja secara spesifik terhadap tenunan-tenunan atau sel-sel tertentu. Misalnya sel-sel saraf, otot, sel-sel pada saluran pencernaan, dan sebagainya.
5 terhadap sistem pertahanan tubuh, dan melakukan invasi ke dalam tubuh inangnya, serta harus ada jalan keluar penyebab penyakit.
Mikroba yang dapat menginfeksi dan dapat menimbulkan penyakit adalah mikroba yang mempunyai daya patogenisitas yang tinggi, daya virulensi yang kuat, dan daya invasi yang tinggi, sehingga dapat berkembang biak dan menyebar ke dalam tubuh inang yang peka, serta mempunyai daya pertahanan dan daya hinder yang baik terhadap serangan sel-sel fagosit di dalam tubuh inang. Proses yang terjadi di setiap tahap sangat kompleks dan biasanya dipengaruhi oleh beberapa faktor. Suatu mikroba dapat kehilangan sifat patogeniknya jika salah satu dari faktor tersebut hilang. Substrat dan lingkungan tempat pertumbuhan mikroba mempengaruhi hilangnya atau terbentuknya faktor patogenik tersebut.
Mikroba patogen yang terdapat di dalam pangan biasanya masuk ke dalam tubuh melalui saluran pencernaan. Infeksi oleh mikroba tersebut dapat dimulai dari membran mukosa pada dinding saluran pencernaan, terutama usus halus. Meskipun demikian, tidak semua mikroba patogen masuk melalui saluran pencernaan dapat menyebabkan infeksi pada membran mukosa tersebut, karena sesungguhnya dinding saluran pencernaan dilindungi oleh lapisan lendir saluran mukus, pergerakan isi saluran pencernaan, dan mikroba komensal yang hidup berkembang biak tanpa merugikan di saluran pencernaan.
Berdasarkan cara penyebarannya dan daya penetrasinya di dalam tubuh, mikroba patogen yang dapat menyebabkan infeksi melalui saluran pencernaan dapat dibedakan atas tiga golongan yaitu: (1) mikroba yang berkembang biak pada permukaan dinding saluran pencernaan, dan tidak menembus terlalu jauh ke dalam sel-sel mukosa, (2) mikroba yang menembus sel-sel mukosa dan berkembang biak di dalam sel-sel tersebut tetapi tidak menyebar ke jaringan-jaringan yang lebih dalam, (3) mikroba yang menyebar ke jaringan-jaringan yang lebih dalam baik dengan cara menembus sel-sel mukosa atau diantara sel-sel mukosa misalnya Salmonella yang menyebabkan salmonellosis (Supardi dan Sukamto 1999).
Enam mikroba patogen yang menjadi penyebab utama KLB keracunan pangan di Amerika Serikat, yaitu Campylobacter jejuni, Clostridium perfringens, Escherichia coli O157:H7, Lysteria monocytogenes, Salmonella, dan Staphylococcus aureus (Doores 1999). Mikroba lain yang banyak menimbulkan KLB keracunan pangan di Indonesia adalah Escherichia coli, Bacillus cereus, Staphylococcus aureus, Clostridium perfringens, Salmonella, Vibrio cholera, dan Pseudomonas cocovenenans (Badan POM RI 2002, 2003a, 2004). Gejala dan akibat yang ditimbulkan, jumlah yang dapat menyebabkan keracunan pangan, dan pencegahan mikroba patogen tersebut dapat dilihat pada Tabel 1. berikut ini.
Tabel 1. Mikroba patogen penyebab keracunan pangan*
Mikroba Jumlah Akibat Gejala Pencegahan
C. jejuni 104 sel/ml Aborsi, infertilitas, enteridis
Sakit perut, demam kadang > 40oC, diare, muntah
Pemanasan (55-60oC)
C. perfringens 5x105 sel/ml
Diare akut Diare Pendinginan
Mikroba Jumlah Akibat Gejala Pencegahan E. coli EPEC ETEC EIEC EHEC
106-109 sel
108-109 sel
106 sel
10-100 sel
Diare akut sampai diare berdarah
Diare berair, muntah, dan demam
Diare berair, kejang perut, demam
Diare basiler (berlendir, berdarah)
Diare berdarah
Pemasakan (<70oC; 2 menit)
L. monocytogenes 103 sel Aborsi Demam, gangguan gastroenteritis, gejala mirip flu
Pemasakan medium atau pada suhu 70oC
Salmonella sp. 105-107 sel Salmonelosis Typhoid
Non-typhoid
Demam enterik (demam 39-40oC, kejang perut, sakit kepala, hilang nafsu makan), konstipasi. Sakit perut, diare, muntah dan demam
Pemanasan 70-75oC (3-7 menit); 66oC (12 menit); 60oC
(78-83 menit)
S. aureus 106 sel/ml Diare Mual, muntah, kejang perut, diare
Pendinginan (-10)-0oC Pemanasan 66oC; 10 menit
Vibrio sp. 105-107 sel Kolera Diare, diare disertai serpihan mukus
Pemasakan (>70oC)
P. cocovenenans 2 mg/100g Keracunan asam bongkrek; toksoflavin
Sakit perut, keringat berlebihan, lelah mual
pH < 5,5 atau penambahan garam NaCl (2,75-3,0%)
B. cereus 105 sel/g Diare Diare, kejang perut, muntah
Pendinginan (10oC), pemanasan (60oC)
Morganella sp. Keracunan histamin
Mual, muntah, diare, kejang perut, sakit kepala, rasa terbakar
Pembekuan, Pendinginan (1oC) *Badan POM RI (2003b)
B.
Salmonella
Typhimurium
Salmonella merupakan salah satu genus dari famili Enterobacteriaceae. Taksonomi Salmonella
sangatlah kompleks. Berdasarkan skema Kauffman-white dalam Brenner et al. (2000) genus
Salmonella terdiri dari dua spesies diantaranya adalah Salmonella bongori (V) dan Salmonella enterica. Spesies Salmonella enterica ini terdiri dari enam subspesies diantaranya adalah (1)
7
enterica subsp. arizonae (IIIa), (4) Salmonella enterica subsp. diarizonae (IIIb), (5) Salmonella enterica subsp. houtenae (IV), dan (6) Salmonella enterica subsp. indica (VI). Salmonella enterica
subsp. enterica terdiri dari 1.454 serotype/serovar dan salah satunya adalah serovar Typhimurium. Oleh karena itu, kata “Typhimurium” pada Salmonella Typhimurium atau biasa disingkat dengan
S. Typhimurim ditulis tidak dengan huruf miring dan juga ditulis dengan huruf kapital diawal katanya (Brenner et al. 2000).
Salmonella Typhimurium merupakan bakteri Gram negatif, fakultatif anaerob, berbentuk batang, tidak membentuk spora dan Salmonella Typhimurium juga memiliki flagella peritrikus sehingga bersifat motil. Salmonella Typhimuriumberdiameter 0,7 hingga 1,5 µm dan dengan panjang 2 hingga 5 µm. Suhu minimum untuk pertumbuhan Salmonella Typhimurium adalah 45oC, sedangkan suhu pertumbuhan optimumnya adalah 35-37oC (D’Aoust 2000).
Bakteri dari genus Salmonella merupakan bakteri penyebab infeksi yang jika tertelan masuk dan berkembang biak di dalam tubuh akan menimbulkan gejala yang disebut salmonellosis. Penyakit yang disebabkan oleh Salmonella dibagi menjadi dua grup besar yaitu nontyphoid salmonellosis (gastroenteritis) dan typhoid salmonellosis (demam typhoid). Gastroenteritis disebabkan karena infeksi Salmonella Typhimurium yang terbatas pada epithelium usus sedangkan demam typhoid disebabkan karena infeksi yang terjadi pada keseluruhan sistem.
Typhoid salmonellosis (demam typhoid) memiliki gejala awal yang agak berbeda dan jauh lebih lambat (24-48 jam setelah konsumsi) daripada nontyphoid salmonellosis (12 jam setelah konsumsi). Demam typhoid akan mengalami peningkatan secara bertahap setiap harinya dan sering kali muncul bintik merah pada kulit. Gejala yang timbul pada demam typhoid adalah demam dengan suhu 39-40oC, kejang perut, sakit kepala, hilang nafsu makan, dan konstipasi. Penderita mungkin mengalami perforsi dan pendarahan usus dan koma. Penderita yang telah sembuh seringkali menjadi
asymptomic carriers dimana organisme ini tetap berada dalam kantong empedu dan usus. Sedangkan gastroenteritis digejalai dengan mual, muntah, kejang perut, dan diare (Ziprin dan Hume 2001).
Pangan yang sering terkontaminasi Salmonella Typhimurium adalah telur dan hasil olahannya, ikan dan hasil olahannya, daging ayam, daging sapi, susu dan hasil olahannya. Keracunan pangan oleh
Salmonella disebabkan karena pangan mengandung Salmonella dalam jumlah yang signifikan yaitu 107 sel (Jay et al. 2005).
C.
Metode Identifikasi
Salmonella
Typhimurium Secara Konvensional
Analisis pangan terhadap kemungkinan adanya bakteri patogen atau bakteri pembusuk merupakan standar yang diharuskan untuk mengetahui kualitas pangan dan menjamin keamanan pangan (de Boer dan Baumer 1999). Analisis tersebut biasanya menggunakan metode konvensional yang telah distandarkan sehingga setiap negara di dunia memiliki standar yang sama dalam menilai kualitas pangan. Metode deteksi konvensional yang dimaksud ialah metode pengkulturan mikroba pada media spesifik dan menghitung sel mikroba yang hidup dalam pangan. Prinsipnya ialah mikroba bermultiplikasi pada media pertumbuhan sehingga dapat dideteksi. Metode ini sensitif, dapat memberi hasil uji kualitatif maupun kuantitatif, dan dapat menjadi standar bagi uji mikrobiologi terhadap produk pangan secara internasional.
Pada media Lactose Broth (LB), laktosa akan difermentasi oleh sebagian besar bakteri
non-Salmonella sehingga menyebabkan penurunan pH media. Penurunan pH media akan menghambat pertumbuhan bakteri non-Salmonella, sementara bakteri Salmonella dapat tetap tumbuh. Pada tahap ini inkubasi dilakukan pada suhu 37oC dan dianggap positif jika terjadi kekeruhan di media LB.
Metode pengayaan selektif Rappaport-Vassiliadis medium (RV) yang mengandung senyawa selektif seperti malachite green dan magnesium klorida yang dikombinasikan pada pH rendah (5,2 ± 2) akan menghambat pertumbuhan mikroba alami yang berasal dari saluran pencernaan selain
Salmonella. Media yang digunakan pada tahap isolasi terdiri dari tiga jenis agar selektif yaitu Hektoen Enteric Agar (HEA), Xylose Lysine Deoxycholate Agar (XLDA), dan Bismuth Sulfite Agar (BSA). Dari ketiga media yang berbeda ini akan ditemukan koloni spesifik yang diindikasikan Salmonella.
Pada media HEA dan XLDA yaitu ditandai dengan koloni dengan bintik hitam di tengah yang menghasilkan H2S, sedangkan pada media BSA koloni Salmonella akan menghasilkan warna coklat hingga hitam dengan kilau metalik (Difco dan BBL manual 2003).
Tahap akhir pada metode konvensional adalah tes biokimia, identifikasi, dan uji serologi. Salah satu langkahnya adalah dengan melihat pertumbuhan Salmonella pada media TSIA (Triple Sugar Iron Agar) dan LIA (Lysine Iron Agar). Pada media TSIA, jika hasil positif maka media akan berwarna kuning dengan adanya indikator pH fenol merah karena terfermentasinya laktosa oleh Salmonella.
Sedangkan pada media LIA akan terbentuk warna gelap pada agar miring yang menunjukkan terjadinya deaminasi lisin dan ammonia yang terbentuk akan bereaksi dengan ferric ammoniumcitrate
serta endapan hitam pada dasar tabung yang menunjukkan adanya produksi H2S (Difco dan BBL manual 2003).
Saat ini telah banyak teknik yang dikembangkan untuk mempermudah pelaksanaan metode konvensional, misalnya: gravimetric diluter, pulsifier dan stomacher yang mempermudah homogenisasi; spiral plater, dipslide, dan petrifilm untuk enumerisasi dan deteksi; colony counter dan kit uji untuk konfirmasi atau identifikasi (de Boer dan Beumer 1999). Meskipun demikian waktu pendeteksian tidak berkurang secara signifikan.
D.
Metode Isolasi dan Identifikasi
Salmonella
Typhimurium Secara Cepat
dengan
Real-Time
PCR
Metode deteksi cepat patogen dan kontaminan mikrobiologi lainnya dalam pangan merupakan hal yang sangat penting untuk menjamin keamanan pangan konsumen. Keuntungan dari metode cepat dibandingkan dengan metode konvensional adalah waktu lebih singkat (4-48 jam), tidak membutuhkan banyak tenaga, serta lebih sensitif dan akurat (Patel dan Williams 1994, Chen et al.
2009).
Metode cepat yang dalam dekade ini berkembang pesat adalah metode deteksi berbasiskan DNA. Metode berbasiskan DNA memiliki spesifikasi terbaik dan sesuai untuk mendeteksi patogen dalam pangan (de Boer dan Baumer 1999). Banyak uji yang berbasiskan DNA, tapi hanya metode probe, PCR dan bakteriofag yang telah dikembangkan secara komersial untuk mendeteksi patogen pencemar pangan (foodborne patogen).
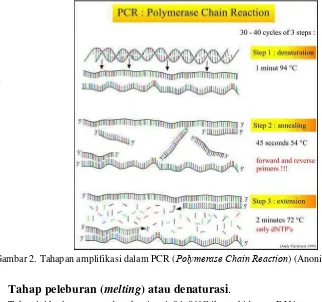
9 Secara prinsip, baik real-time PCR maupun PCR konvensional merupakan proses yang dilakukan berulang-ulang antara 20–30 kali siklus. Setiap siklus terdiri atas tiga tahap utama (Pestana et al.
2010). Berikut adalah tiga tahap utama kerja PCR dalam satu siklus, ketiga tahap utama dalam proses amplifikasi tersebut dapat dilihat lebih jelas pada Gambar 2 berikut ini.
Gambar 2. Tahapan amplifikasi dalam PCR (Polymerase Chain Reaction) (Anonim 2009).
1.
Tahap peleburan (
melting
) atau denaturasi
.
Tahap ini berlangsung pada suhu tinggi, 94–96°C ikatan hidrogen DNA terputus (denaturasi) dan DNA utas ganda menjadi utas tunggal. Biasanya pada siklus awal PCR, tahap ini dilakukan lebih lama (5 menit) untuk memastikan semua berkas DNA terpisah. Pemisahan ini menyebabkan DNA tidak stabil dan siap menjadi template (patokan) bagi primer. Umumnya, waktu/durasi yang dibutuhkan untuk tahap ini yaitu selama 1–2 menit.
2.
Tahap penempelan atau
annealing
.
Primer menempel pada bagian Template DNA yang komplementer urutan basanya. Tahap ini dilakukan pada suhu antara 45–60°C. Penempelan ini bersifat spesifik. Suhu yang tidak tepat menyebabkan tidak terjadinya penempelan atau primer menempel di sembarang tempat. Umumnya, waktu/durasi yang dibutuhkan untuk tahap ini yaitu selama 1–2 menit.
3.
Tahap pemanjangan (
extension
) atau elongasi.
Suhu untuk proses ini tergantung dari jenis enzim DNA polimerase yang dipakai. Karena suhu dinaikkan ke dalam kisaran suhu optimum kerja enzim DNA polimerase. Dengan Taq
ekstensi bergantung pada panjang daerah yang akan diamplifikasi, misalnya adalah 1 menit untuk setiap 1000 bp.
Selain ketiga proses tersebut umumnya tahap kerja PCR didahului dan diakhiri oleh tahapan berikut:
1.
Pra-denaturasi
Dilakukan selama 1-9 menit di awal reaksi untuk memastikan kesempurnaan denaturasi dan mengaktivasi DNA Polimerase (jenis hot-start/aktif jika dipanaskan terlebih dahulu).
2.
Final Elongasi
Umumnya dilakukan pada suhu optimum enzim (70-72oC). Tahap ini dilakukan selama 5-15 menit untuk memastikan bahwa setiap utas tunggal yang tersisa sudah diperpanjang secara sempurna. Proses ini dilakukan setelah siklus PCR terakhir.
Proses amplifikasi PCR dilakukan dengan menggunakan mesin thermal cycler yang dapat menaikkan dan menurunkan suhu dalam waktu cepat sesuai kebutuhan siklus PCR. Pada awalnya untuk melakukan proses tersebut digunakannya tiga penangas air (water bath) untuk melakukan
melting, annealing dan extention secara manual, berpindah dari satu suhu ke suhu lainnya menggunakan tangan. Tetapi sekarang mesin thermal cycler sudah terotomatisasi dan dapat diprogram sesuai kebutuhan.
Secara teoritis, PCR dapat mengamplifikasi satu salinan DNA menjadi satu juta kali dalam waktu kurang dari 2 jam (Gambar 3.) maka berpotensi untuk mengurangi bahkan menghilangkan kebutuhan untuk pengayaan kultur, namun kehadiran inhibitor dalam pangan dan dalam berbagai media kultur dapat mencegah terikatnya primer dan mengurangi efisiensi amplifikasi sehingga sangat sensitif dicapai dengan PCR dan kultur murni sering berkurang bila menguji pangan. Oleh karena itu, beberapa pengayaan kultur diperlukan sebelum dilakukannya analisis.
Gambar 3. Jumlah DNA yang dihasilkan dari proses amplifikasi (Anonim 2009)
Komponen lain yang dibutuhkan untuk pengujian ini selain template DNA yang akan digandakan dan enzim DNA polimerase, diantaranya adalah:
1.
Primer
11 menggandakan DNA pada daerah tertentu sepanjang maksimum 10000 bp, dan dengan teknik tertentu bisa sampai 40000 bp. Primer dirancang untuk memiliki sekuen yang komplemen dengan template DNA, sehingga dirancang agar menempel mengapit daerah tertentu yang diinginkan. Langkah pertama dalam perancangan primer adalah mencari/membuat urutan DNA/gen yang diinginkan, umumnya dapat menggunakan basis data umum seperti NCBI. Setelah urutan DNA/gen yang diinginkan diperoleh, kemudian software untuk merancang primer digunakan untuk mempermudah dan memaksimalkan keberhasilan dalam merancang primer. Langkah berikutnya adalah mempertimbangkan dengan hati-hati daerah gen yang akan digunakan sebagai template DNA. Gen yang memiliki struktur sekunder atau urutan nukleotida yang panjang harus dihindari dalam perancangan primer (Pestana et al. 2010).
Besarnya konsentrasi dari primer yang digunakan merupakan hal yang menentukan keberhasilan pengujian. Jika konsentrasi primer pada pengujian dengan real-time PCR yang digunakan terlalu tinggi, maka akan meningkatkan kesempatan untuk terjadinya mispriming yang artinya akan menghasilkan produk PCR yang tidak spesifik karena primer berikatan tidak hanya dengan gen target, tetapi dengan gen-gen lainnya pada template DNA yang ditambahkan (Pestana
et al. 2010), namun jika konsentrasi primer yang digunakan terlalu rendah, maka akan dihasilkan suatu metode pengujian yang tidak sensitif yang menyebabkan terjadinya false-negative dimana sampel yang positif (mengandung gen target) akan terdeteksi negatif (Lee et al. 2006). Oleh karena itu penentuan konsentrasi primer sangat dibutuhkan dalam pengujian dengan real-time
PCR.
2.
dNTP (
deoxynucleoside triphosphate
)
dNTP atau building blocks sebagai ‘batu bata’ penyusun DNA yang baru. dNTP terdiri atas empat macam sesuai dengan basa penyusun DNA, yaitu dATP, dCTP, dGTP dan dTTP.
3.
Buffer PCR
Buffer yang biasanya terdiri atas bahan-bahan kimia untuk mengkondisikan reaksi agar berjalan optimum dan menstabilkan enzim DNA polimerase.
4.
Kofakto
r
Kofaktor berfungsi untuk meningkatkan jumlah akhirdari reaksi. Kofaktor tersebut berupa Ion logam bivalen, umumnya magnesium klorida (Mg2+), sebagai kofaktor bagi enzim DNA polimerase. Tanpa ion ini enzim DNA polimerase tidak dapat bekerja (Pestana et al. 2010). Prinsip amplifikasi real-time PCR sama dengan PCR standar, hanya saja pada real-time PCR ditambahkan label berfluoresensi (fluoresence dye) sehingga penggunaan teknik real-time PCR lebih efisien dan efektif dibandingkan PCR standar (Edward et al. 2004). Terdapat dua jenis komponen label berfluoresensi yang umum digunakan dalam proses real-time PCR, diantaranya adalah label yang berikatan dengan DNA (DNA binding dyes) dan komponen kimia yang berbasiskan probe (Probe-based chemistries).
dari DNA polimerase untuk mendeteksi proses amplifikasi yang terjadi di dalam real-time PCR (Pestana et al. 2010).
1.
DNA
Binding Dyes
, SYBR
®green I
Keuntungan dari SYBR® green I adalah mengukur intensitas fluoresen secara proporsional sesuai dengan produk PCR yang dihasilkan. Sedangkan kekurangannya adalah SYBR® green I yang merupakan label fluoresen yang tidak spesifik sehingga dapat menghasilkan sinyal pengujian false-positive karena terjadinya primer-dimer atau terbentuknya produk yang tidak spesifik oleh karena itu setiap pengujian, penting dilakukannya penganalisisan formasi dari kurva pelelehan (melting curve) (Pestana et al. 2010).
SYBR® green I merupakan label fluoresen yang efektif dari segi biaya jika dibandingkan dengan komponen label kimia lainnya dan juga mudah digunakan. Penentuan konsentrasi primer yang tepat/optimum dapat dengan mudah dicapai dengan menggunakan SYBR® green I sebagai bahan fluoresen dibandingkan dengan menggunakan bahan yang lainnya seperti TaqMan dan Molecular Beacon. Ketika SYBR® green I berada dalam kondisi bebas dalam larutan, maka akan menunjukkan level fluoresen yang relatif rendah, tetapi ketika dsDNA ditambahkan, fluoresen yang berpendar akan meningkat lebih dari 1000 kali lipat. Akumulasi dari amplikon/produk amplifikasi PCR meningkat seiring dengan meningkatnya siklus reaksi, maka intensitas fluoresen juga akan meningkat karena molekul SYBR® green I berikatan dengan DNA, dan kemudian akumulasi dari produk yang dihasilkan dapat diukur dalam real-time PCR. SYBR® green I dapat berikatan dengan DNA utas ganda pada suhu 55oC, namun pada suhu suhu 95oC SYBR® green I akan terdisosiasi/terlepas dari DNA utas ganda. Hal tersebut dapat dilihat pada Gambar 4. berikut ini.
Gambar 4. DNA binding dyes dalam real-time PCR. Fluoresen secara dramatis meningkat ketika molekul dye berikatan dengan DNA (Pestana et al. 2010)
Sensitifitas deteksi dengan menggunakan SYBR® green I dipengaruhi oleh pembentukan primer-dimer, spesifisitas primer yang rendah, konsentrasi primer (dapat membatasi), dan terbentuknya struktur sekunder pada produk PCR. Semua faktor tersebut dapat mengarahkan pada terbentuknya produk DNA utas ganda yang tidak diharapkan yang dapat berikatan dengan SYBR® green I dan menghasilkan sinyal fluoresen. Telah dijelaskan sebelumnya bahwa SYBR® green I tidak dapat membedakan satu DNA dengan DNA yang lainnya, oleh karena itu penting untuk menganalisis kurva pelelehan pada akhir pengujian dengan real-time PCR (Pestana et al.
13
2.
Probe-Based Chemistry
Penggunaan berbasiskan probe merupakan pendeteksian yang lebih spesifik karena jenis fluoresen tersebut menggunakan pemeriksaan/penyelidikan internal disamping penggunaan sepasang primer yang digunakan untuk mengamplifikasi daerah tertentu. Penggunaan probe lebih mahal dan perlu dilakukannya perancangan sekuen nukleotida agar sekuen pada probe tersebut sesuai dengan sekuen pada gen target. Oleh karena itu deteksi dengan menggunakan probe nantinya dibutuhkan proses validasi dan optimasi yang terpisah khusus untuk probe, namun dapat menghasilkan spesifisitas yang lebih tinggi jika menggunakan Molecular Beacon (Pestana et al.
2010).
Real-time PCR melakukan amplifikasi dan deteksi dalam satu tahapan sebab akumulasi produk spesifik dicatat secara kontinyu selama siklus. Hal ini tidak dapat dilakukan pada PCR standar yang masih mengandalkan elektroforesis gel agarosa untuk mengkuantitasi amplikon. Selain itu, hasil dari elektroforesis gel agarosa dapat memperpanjang waktu deteksi dan timbulnya kontaminasi.
Pendeteksian produk real-time PCR secara kuantitatif berbeda dengan PCR standar yang mengkuantitasi amplikon di akhir fase amplifikasi (fase plato) dan juga berdasarkan panjang basa atau bobot molekul padahal molekul yang berbeda mungkin saja memiliki ukuran yang sama atau hampir sama. Kuantitasi produk real-time PCR terdeteksi pertama kali di setiap siklus (fase eksponensial) dan dihitung berdasarkan threshold cycle (Ct) yaitu waktu dimana intensitas fluoresen lebih besar dari pada fluoresen yang ditimbulkan oleh noise (background fluorescence). Noise dapat disebabkan oleh penempelan larutan isolat DNA beserta pereaksi-pereaksi PCR pada dinding tabung microwell.
Keunggulan real-time PCR lainnya ialah analisis dapat dilakukan tanpa membuka tabung sehingga mengurangi resiko kontaminasi amplikon PCR atau molekul target lainnya. Menurut Edward
et al. (2004), aplikasi teknologi real-time PCR mengurangi waktu penanganan atau pengujian dan meningkatkan keakuratan kuantifikasi metode PCR.
Sebelum tahap amplifikasi dengan real-time PCR, diperlukan tahap persisapan/pra-amplifikasi, kemudian setelah tahap amplifikasi diperlukan evaluasi produk real-time PCR. Tahap pra-amplifikasi PCR standard dan real-time PCR tidak berbeda, meliputi persiapan sampel bakteri dan isolasi DNA untuk memperoleh isolat DNA sebagai DNA target amplifikasi. Protokol yang baik dalam mempersiapkan isolat DNA adalah fokus pada tahap pengumpulan/pemanenan patogen, penghilangan inhibitor yang terdapat pada pangan atau pada media kultur, dan pengisolasian DNA (Lee et al. 2006). Prinsip dasar isolasi DNA adalah serangkaian proses untuk memisahkan DNA dari komponen-komponen sel lainnya. Hasil ekstraksi merupakan tahapan penting untuk langkah berikutnya. Oleh sebab itu, tahapan ini harus dilakukan dengan baik dan bebas kontaminasi. Terdapat enam tahap dalam mengisolasi DNA, yaitu (1) preparasi sampel, (2) pelisisan sampel yang umumnya menggunakan buffer yang mengandung Tris.HCl pH8,0; EDTA pH 8,0; CTAB, proteinase K, dan SDS (sodium dodecylsulphate), (3) proteksi dan stabilisasi DNA dengan menggunakan buffer TE yang mengandung Tris.HCl dan EDTA pH 8,0 dan juga ditambahkan NaCl, (4) pemisahan DNA dari debris sel dilakukan dengan cara sentrifugasi dan juga ditambahkan dengan fenol, kloroform, dan isoamil alkohol dengan perbandingan tertentu atau dengan dididihkan pada air suhu 100oC, (5) presipitasi DNA dengan menambahkan etanol 96% atau isopropanol, sedangkan (6) pemekatan DNA dilakukan dengan pencucian menggunakan etanol 70% dan ditambahkan dengan buffer TE (Tris.HCl dan EDTA). Berdasarkan prinsip tersebut, banyak metode untuk mengisolasi/mengekstraksi DNA dari sel mikroba salah satunya adalah metode pendidihan, dan dari berbagai metode tersebut, banyak industri yang mengembangkan suatu kit untuk memudahkan pengisolasian/pengekstraksian DNA.
Salmonella) dapat dengan mudah diisolasi/diekstraksi dengan menggunakan metode pendidihan/ mendidihkan sel bakteri di dalam air (Lee et al. 2006).
Tahap isolasi DNA dengan metode pendidihan dilakukan setelah tahap enrichment pada kultur murni maupun pada sampel pangan. Tahap enrichment ini berfungsi untuk menyembuhkan sel mikroba yang rusak maupun memperbanyak mikroba agar memberi keyakinan bahwa mikroba yang diisolasi adalah mikroba hidup. Selain itu juga untuk mengencerkan komponen inhibitor PCR yang terdapat pada matriks sampel (Rådström et al. 2004).
Bahan yang pertama kali digunakan pada metode pendidihan adalah buffer TE dimana mengandung Tris HCl 1 M pH 8,0 dan EDTA 0,5 M yang dicampur dengan pelet yang dihasilkan setelah sentrifugasi sampel. Proses sentrifugasi dan resuspensi pelet yang dihasilkan dengan buffer TE secara berulang berfungsi untuk mencuci sampel (kultur murni/sampel spike) dari inhibitor-inhibitor yang terkandung di dalamnya yang dapat mengganggu tahap amplifikasi DNA nantinya (Lee at al.
2006). Selain itu buffer TE juga berfungsi untuk menjaga kestabilan DNA agar tidak terdegradasi ketika pelisisan sel terjadi.
Kemudian bahan berikutnya adalah penambahan lisozim ke dalam pelet bersamaan dengan ditambahkannya buffer TE. Lisozim berfungsi untuk melisiskan dinding sel bakteri dimana lisozim dapat merusak membran luar sel bakteri. Aktivitas lisozim efektif pada suhu ruang. Kemudian pada metode pendidihan juga ditambahkan proteinase K. Proteinase K merupakan enzim proteolitik yang digunakan untuk menghilangkan protein. Protein merupakan salah satu inhibitor dalam proses amplifikasi DNA. Suhu optimum aktivitas enzim proteinase K adalah 55-60oC selama 1 jam. Semakin lama inkubasi, maka daya recovery DNA semakin baik (Sambrook et al. 1989).
Pendidihan di dalam air yang bersuhu 100oC berfungsi untuk menginaktivasi enzim proteinase K dan enzim nuklease atau DNAse dimana dapat mencerna asam nukleat dan menyebabkan penurunan kualitas maupun kuantitas DNA selama penyimpanan. Pendidihan dengan pemanasan 100oC juga dapat mempercepat lisis dinding sel bakteri sehingga DNA dapat diekstraksi sekaligus mempermudah proses denaturasi rantai DNA ketika dilakukan amplifikasi dengan PCR. Selain itu pendidihan juga dapat berfungsi mendenaturasi protein-protein inhibitor. Proses pendidihan tidak dapat mendenaturasi DNA plasmid karena secara topologi intertwined (Sambrook et al. 1989), namun dapat mendenaturasi DNA kromosomal.
Pendinginan segera dilakukan di dalam freezer setelah dilakukannya pendidihan agar DNA kromosomal dapat terenaturasi. Proses pendinginan ini juga menyebabkan debris sel, protein-protein inhibitor dan DNA plasmid mengendap. DNA kromosomal akan larut dalam supernatan sedangkan debris sel, protein inhibitor, dan DNA plasmid akan terendapkan pada pelet setelah proses sentrifugasi dimana dilakukan setelah proses pendinginan tadi. Dan supernatan yang dihasilkan tersebut diambil sebagai isolat DNA yang akan digunakan pada tahap amplifikasi selanjutnya.
Setelah persiapan isolat DNA selesai, maka dilakukan tahap amplifikasi yang dijalankan dalam
thermal cycler dan dihasilkan/ditampilkan dalam bentuk grafik pada layar komputer dengan software
yang aplikabel terhadap thermal cycler, contohnya adalah software IQ-5 (Bio-Rad). Grafik-grafik yang diperoleh berupa grafik amplifikasi, kurva standar, kurva pelelehan (melt curve) dan kurva puncak pelelehan (melt peak curve). Grafik tersebut digunakan untuk mengevaluasi kinerja amplifikasi real-time PCR.
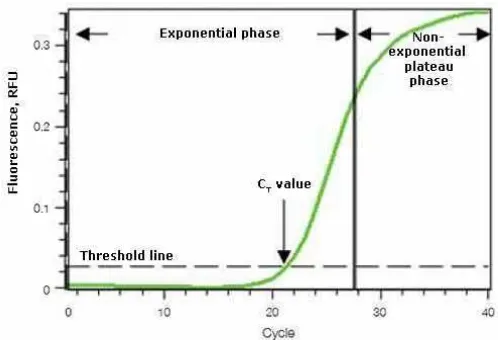
1.
Kurva Amplifikasi
15 Gambar 5. Grafik sigmoid proses amplifikasi dengan real-time PCR (Pestana et al. 2010)
Amplifikasi ditetapkan berdasarkan intensitas fuoresen, semakin banyak produk amplifikasi yang dihasilkan, semakin besar akumulasi fluoresen yang terbaca. Peningkatan fuoresen tersebut ditandai dengan terbentuknya grafik sigmoid, seperti yang tergambar pada Gambar 5. (garis hijau). Grafik sigmoid akan berpotongan dengan baseline threshold yang telah ditentukan secara otomatis oleh program. Titik perpotongan antara grafik sigmoid dan baseline threshold jika direfleksikan pada sumbu-x (Cycle) adalah threshold cycle (Ct) bagi sampel yang diamplifikasi.
Nilai Ct adalah siklus di atas noise dimana akumulasi produk (senilai 2n, n adalah jumlah pengulangan siklus amplifikasi) terbaca pertama kali pada fase eksponensial. Fase eksponensial berakhir menjadi fase plato saat pereaksi dalam campuran reaksi PCR sudah habis. Nilai Ct digunakan dalam kurva standarsebagai fungsi terhadap log konsentrasi bakteri.
2.
Melt Curve
dan
Melt Peak Curve
Setelah pengujian dengan real-time PCR selesai, thermo cycler dapat disesuaikan untuk menghasilkan kurva pelelehan (melt curve) dan kurva puncak pelelehan (melt peak curve) dengan meningkatkan suhunya sedikit demi sedikit hingga mencapai titik pelelehan dimana DNA untai ganda menjadi DNA untai tunggal dan kemudian mengukur perubahan fluoresen yang dihasilkan. Pada titik pelelehan, untai ganda DNA terpisah dan sinyal fluoresen pun turun. Kurva pelelehan menghubungkan laju perubahan Relative Fluorescence Unit (RFU) per waktu (T) (-d(RFU)/dT) pada sumbu-y dengan suhu pada sumbu-x, kurva tersebut akan menunjukkan suhu pelelehan (Tm). Suhu pelelehan merupakan suhu dimana 50% amplikon DNA untai ganda terpisah menjadi dua untai tunggal.
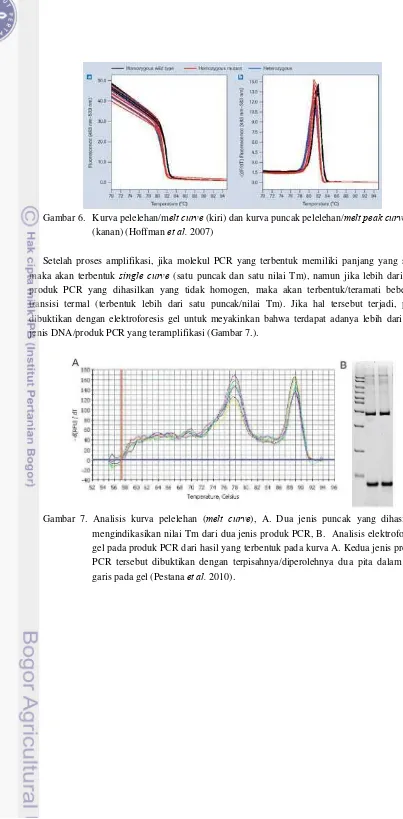
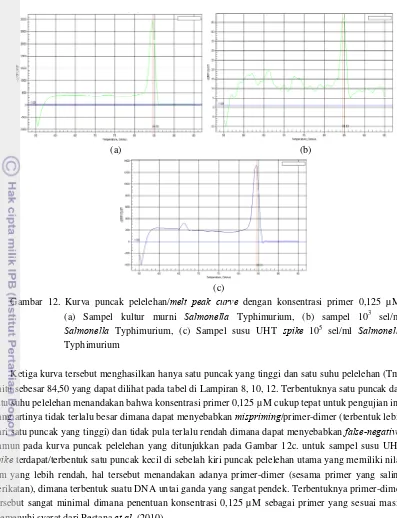
Gambar 6. Kurva pelelehan/melt curve (kiri) dan kurva puncak pelelehan/melt peak curve
(kanan) (Hoffman et al. 2007)
Setelah proses amplifikasi, jika molekul PCR yang terbentuk memiliki panjang yang sama maka akan terbentuk single curve (satu puncak dan satu nilai Tm), namun jika lebih dari satu produk PCR yang dihasilkan yang tidak homogen, maka akan terbentuk/teramati beberapa transisi termal (terbentuk lebih dari satu puncak/nilai Tm). Jika hal tersebut terjadi, perlu dibuktikan dengan elektroforesis gel untuk meyakinkan bahwa terdapat adanya lebih dari satu jenis DNA/produk PCR yang teramplifikasi (Gambar 7.).
17
II.
METODOLOGI PENELITIAN
A.
Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan di Pusat Riset Obat dan Makanan Badan POM RI pada bulan April 2011 hingga Juli 2011.
B.
Bahan
Bahan-bahan yang digunakan di dalam penelitian ini adalah sampel susu UHT, bakteri uji
Salmonella Typhimurium (ATCC 14028) dan bakteri kontrol negatif Shigella sonnei (ATCC 25931). Media yang digunakan untuk menumbuhkan dan menghitung jumlah bakteri Salmonella
Typhimurium dan Shigella sonnei yaitu larutan pengencer NaCl 0,85%, Heart Infusion Broth (Oxoid), dan Xylose Lysine Deoxycholate Agar (Oxoid). Bahan-bahan yang digunakan untuk mengisolasi DNA bakteri dengan menggunakan metode pendidihan yaitu buffer TE yang mengandung Tris.HCl 1 M pH 8,0 dan EDTA 0,5 M; lisozim (USB), dan proteinase K (USB). Bahan yang digunakan untuk mengisolasi DNA dengan menggunakan kit komersial QIAamp® DNA Blood Mini Kit (Qiagen) diantaranya adalah CTAB, Proteinase K, etanol 96%, buffer AL, buffer AW1, buffer AW2, dan buffer AE.
Bahan-bahan yang digunakan untuk mengamplifikasi DNA dengan menggunakan real-time PCR yaitu buffer TE pH 8,0; forward primer invA (5’-ATC AGT ACC AGT CGT CTT ATC TTG AT-3’),
reverse primer invA (5’-TCT GTT TAC CGG GCA TAC CAT-3’) (Shanmugasundaram et al. 2009),
template DNA yang diperoleh dari tahap isolasi DNA, SsoFastTM EvaGreen® Supermix yang di dalamnya terdiri dari 2x reaction buffer dengan dNTPs, Sso7d-fusion polymerase, MgCl2, EvaGreen dye (sama dengan SYBR® Green I), dan penstabil. Bahan yang digunakan untuk mengukur kemurnian DNA dengan spektrofotometer uv-vis DNA adalah milliQ.
C.
Alat
Alat-alat yang digunakan di dalam penelitian ini adalah cawan petri, mikropipet, tip mikropipet, gelas ukur, gelas piala, tabung reaksi, batang lup inokulasi, rak tabung reaksi, vortex, neraca analitik, erlenmeyer, hot plate, label, inkubator, bulp, bunsen, korek, petroff-hausser, mikroskop, masker dan sarung tangan latex. Alat yang digunakan untuk isolasi DNA dan amplifikasi real-time PCR diantaranya adalah IQTM5 real-time PCR (Bio Rad), plate 96 Well Reaction(BioRad), PCR Sealer TM Microseal® ‘B’ Film (BioRad), Refrigerator, mikrosentrifus (Hettich), Water Bath, tabung mikrosentrifus 2 ml, tip berfilter, MixMate PCR 96. Alat yang digunakan untuk mengukur kemurnian isolat DNA adalah UV-VIS Spektrofotometer DNA (Shimadzu) dan kuvet.
D.
Metode Penelitian
a.
Tahapan Pra-penelitian
1. Persiapan Bahan Kimia2. Persiapan Media Tumbuh Mikroba
b.
Tahapan Penelitian
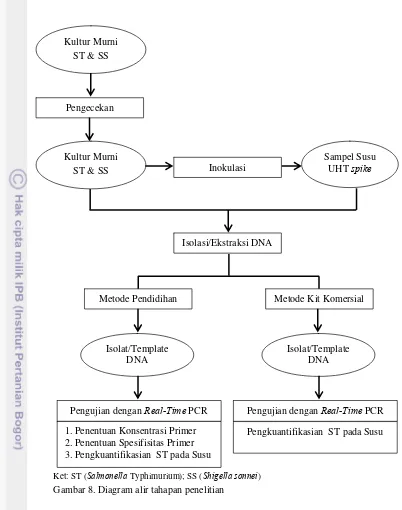
Ket: ST (Salmonella Typhimurium); SS (Shigella sonnei)
Gambar 8. Diagram alir tahapan penelitian
a.
Tahapan Pra-penelitian
1.
Persiapan Bahan Kimia
Bahan kimia yang disiapkan untuk penelitian ini diantaranya adalah larutan pengencer NaCl 0,85%, lisozim, proteinase K, CTAB, dan buffer TE pH 8,0. Pembuatan NaCl 0,85% dilakukan dengan melarutkan 4,25 gram kristal NaCl ke dalam 500 ml milliQ di dalam labu ukur dan diaduk hingga homogen. Lisozim dibuat dengan melarutkan 10 mg/ml lisozim dengan 25 ml larutan Tris.HCl 10 mM pH 8,0 dan dikocok sampai jernih. Pembuatan proteinase K dilakukan dengan melarutkan 10 mg/ml proteinase K dengan 25 ml Tris.HCl 10 mM pH 8,0 dan diaduk sampai jernih.
Cetyl trimethilammonium bromide (CTAB) dibuat dengan melarutkan 10 gram CTAB, 41 gram NaCl, dan 7,87 gram HCl ke dalam 400 ml akuades. Kemudian larutan tersebut ditambah dengan NaOH sampai larutan mencapai pH 8,0 dan ditambah dengan 20 ml EDTA
Pengujian dengan Real-Time PCR Pengkuantifikasian ST pada Susu Pengecekan
Isolasi/Ekstraksi DNA
Metode Pendidihan Metode Kit Komersial
Isolat/Template DNA
Isolat/Template DNA Kultur Murni
ST & SS Inokulasi
Sampel Susu UHT spike
Pengujian dengan Real-Time PCR 1. Penentuan Konsentrasi Primer 2. Penentuan Spesifisitas Primer 3. Pengkuantifikasian ST pada Susu Kultur Murni
19 0,5 M pH 8,0. Larutan tersebut dituang ke dalam labu ukur 500 ml dan ditera dengan akuades. Larutan dipindahkan ke dalam erlenmeyer, divortex sampai larut kemudian dilakukan degasing (penghilangan gas dari larutan). Larutan tersebut selanjutnya disaring dengan kertas saring dan disterilisasi dengan autoclave dan disimpan pada suhu ruang.
Bahan kimia lainnya yang disiapkan adalah buffer TE pH 8,0. Buffer TE pH 8,0 dibuat dengan mencampurkan 5 ml Tris.HCl 1 M pH 8,0 dengan 1 ml EDTA 0,5 M ke dalam labu ukur 500 ml dan ditambahkan milliQ sampai tanda tera. EDTA 0,5 M pH 8,0 diperoleh dengan menimbang 18,6 gram EDTA (Titriplex®; C10H14Na2O8.2H2O) dan dimasukkan ke dalam labu ukur 100 ml kemudian ditambahkan dengan 80 ml milliQ sedikit demi sedikit. Larutan tersebut ditambah dengan NaOH sampai larutan tersebut memiliki pH 8,0. Larutan kemudian ditera dan disterilisasi dengan autoclave. Seluruh proses pembuatan tersebut dapat dilihat pada Lampiran 1a, b, c, d, dan e.
2.
Persiapan Media Tumbuh Mikroba
Media tumbuh yang digunakan baik untuk Salmonella Typhimurium maupun Shigella sonnei adalah Heart Infusion Broth (HIB)dan Xylose Lysine Deoxycholate Agar (XLDA).
Media HIB dibuat dengan melarutkan 18,5 gram HIB dengan 500 ml akuades dan diaduk sampai homogen. Setiap 5 ml larutan HIB dipipet ke dalam tabung reaksi steril dan ditutup dengan kapas dan alumunium foil kemudian disterilisasi dalam autoclave. Media XLDA dibuat dengan melarutkan 27,75 gram XLDA ke dalam 500 ml akuades steril di dalam erlenmeyer steril. Kemudian larutan tersebut dipanaskan di atas hot plate sampai mendidih dan larut sempurna tetapi tidak sampai over heat. Setelah itu didinginkan sampai suhu 50oC dan masing-masing sebanyak 25 ml dituang ke dalam cawan petri steril. Proses tersebut dapat dilihat lebih jelas pada Lampiran 2a, b.
b.
Tahapan Penelitian
1.
Pengecekan Kemurnian
Salmonella
Typhimurium dan
Shigella
sonnei
Masing-masing bakteri Salmonella Typhimurium dan Shigella sonnei pada media Heart Infusion Broth (HIB) yang disimpan dalam refrigerator diinokulasi sebanyak 1 ose ke dalam HIB baru kemudian diinkubasi pada suhu 37oC selama 24 jam. Jika HIB yang telah diinkubasi tersebut menjadi keruh, selanjutnya HIB tersebut digores kuadran pada media
Xylose Lysine Deoxycholate Agar (XLDA) yang berbeda, kemudian diinkubasi selama 24 jam pada suhu 37oC. Satu koloni Salmonella Typhimurium dan Shigella sonnei spesifik yang tumbuh pada XLDA diinokulasikan kembali ke dalam media HIB baru yang berbeda pula dan diinkubasi pada suhu 37oC selama 24 jam.
2.
Inokulasi Sampel Pangan Susu UHT dengan Kultur Murni
Salmonella
Typhimurium dan
Shigella sonnei
105 sel/ml maka dilakukan pengenceran pada suspensi tersebut dengan menggunakan larutan pengencer NaCl 0,85% sampai didapatkan suspensi dengan konsentrasi 105 sel/ml.
Sebanyak 5 ml suspensi dengan konsentrasi 105 sel/ml tersebut dimasukkan ke dalam 5 ml susu UHT dan dikocok hingga homogen. Kemudian sampel susu UHT tersebut diinkubasi pada suhu 37oC selama 1 jam. Untuk mengetahui jumlah mikroba akhir
Salmonella Typhimurium yang terkandung pada susu UHT, maka sampel susu UHT ditumbuhkan dan dihitung dengan metode konvensional pada media Xylose Lysine Deoxycholate Agar (XLDA) yang diinkubasi pada suhu 37oC selama 24 jam. Sebelumnya untuk mengetahui jumlah mikroba (Salmonella Typhimurium) natural pada susu UHT, maka dilakukan penghitungan jumlah mikroba awal dengan menginokulasi 1 ml susu pada media XLDA dan diinkubasi pada suhu 37oC selama 24 jam.
3.
Isolasi/Ekstraksi DNA Mikroba
Proses isolasi/ekstraksi DNA mikroba baik dari kultur murni maupun dari sampel susu UHT spike dapat dilakukan dengan dua metode, yaitu metode pendidihan dan metode kit komersial. Masing-masing metode dijelaskan pada sub bab di bawah ini.
a)
Isolasi DNA
Salmonella
Typhimurium dengan Metode
Pendidihan (Rahayu
et al.
2009)
Satu ml suspensi kultur murni mikroba spesifik yang terdapat dalam media HIB atau sampel susu UHT spike dimasukkan ke dalam tabung mikrosentrifus. Suspensi tersebut disentrifus dalam mikrosentrifus selama 10 menit 14.000 rpm dengan suhu 20oC dengan tujuan untuk mengendapkan sel bakteri. Supernatan yang dihasilkan dibuang dan pelet diresuspensi dengan 500 µl buffer TE lalu dihomogenkan dengan cara divortex. Suspensi tersebut disentrifus kembali pada mikrosentrifus selama 3 menit 14.000 rpm pada suhu 20oC. Untuk sampel susu UHT spike, pencucian dengan buffer TE dan disentrifus dengan mikrosentrifus diulang kembali sebanyak satu kali. Pelet yang dihasilkan diresuspensi dengan 100 µl Lisozim dan dihomogenisasi dengan cara divortex. Kemudian suspensi tersebut diinkubasi pada suhu ruang selama 15 menit.
Setelah diinkubasi, sebanyak 100 µl proteinase K ditambahkan ke dalam suspensi dan divortex sampai homogen. Selanjutnya suspensi diinkubasi pada suhu 55oC selama 1 jam dan dinkubasi kembali pada suhu 100oC selama 15 menit. Proses tersebut bertujuan untuk melisiskan sel bakteri dan menghilangkan protein-protein yang terkandung di dalamnya. Penambahan buffer TE berfungsi untuk menjaga kestabilan DNA pada saat melisiskan sel. Suspensi yang telah diinkubasi didinginkan dalam es sampai membeku dan di-thawing ketika selanjutnya akan disentrifus selama 5 menit 14.000 rpm pada suhu 20oC. Supernatan yang dihasilkan dipindahkan pada tabung mikrosentrifus baru, disimpan sebagai isolat DNA pada suhu -20oC. Diagram alir proses ini dapat dilihat pada Lampiran 3.
21
b)
Isolasi DNA
Salmonella
Typhimurium dengan Kit Komersial
(Berdasarkan QIAamp
®DNA Blood Mini Kit
Handbook
dengan
modifikasi Bioteknologi PROM)
Tahapan proses dengan menggunakan kit komersial dilakukan sesuai dengan anjuran dari produsen yang bersangkutan, salah satunya berdasarkan protokol/handbook
yang dikeluarkan oleh Qiagen untuk penggunaan QIAamp® DNA Blood Mini Kit dalam mengisolasi/mengekstraksi DNA mikroba. Kit tersebut terdiri dari empat jenis buffer (AW1, AW2, AE, dan AL), collection tube, dan kolom mini dimana di dalamnya terdapat filter putih. Di bawah ini (Gambar 9.) merupakan gambar kit komersial yang digunakan pada penelitian ini.
Gambar 9. Kit komersial QIAamp® DNA Blood Mini Kit (Qiagen)
Proses isolasi/ekstraksi DNA dilakukan dengan penambahan buffer AL yang dilakukan setelah penambahan proteinase K dimana bersama-sama dengan proteinase K berfungsi untuk melisiskan sel bakteri sehingga dinding sel bakteri rusak. Supernatan yang dihasilkan dari proses sentrifugasi suspensi tersebut dimasukkan ke dalam kolom mini berfilter sehingga DNA tersangkut/terikat di dalamnya. Penambahan buffer AW1 dan buffer AW2 yang dilakukan setelahnya kedalam kolom mini berfilter yang berfungsi untuk pencucian dimana proses pencucian tersebut dapat meningkatkan kemurnian DNA nantinya serta meyakinkan penghilangan kontaminan secara keseluruhan dari proses tersebut tanpa mempengaruhi pengikatan DNA pada filter.
Penambahan buffer AE ke dalam kolom mini dan dilakukan proses sentrifugasi setelah proses pencucian berfungsi untuk mengelusi DNA dari filter dan juga berfungsi dalam penyimpanan purifikasi DNA. Supernatan yang dihasilkan merupakan isolat DNA yang kemudian disimpan di dalam freezer suhu -20oC. Buffer AE yang digunakan mengandung 10 mM Tris.Cl; 0,5 mM EDTA pH 9,0 dimana pH yang basa dapat menghindari DNA dari degradasi karena hidrolisis asam.
Teknis pengisolasian DNA dengan kit komersial pertama-tama adalah satu ml suspensi kultur murni mikroba spesifik yang terdapat dalam media HIB atau sampel susu UHT dimasukkan ke dalam tabung mikrosentrifus. Kemudian ditambahkan dengan 1 ml CTAB sebagai suatu modifikasi metode dari prosedur yang ditunjukkan pada handbook
dihomogenkan dengan vortex kemudian diinkubasi selama 20 menit pada suhu 65oC. Setelah itu, ditambahkan buffer AL sebanyak 300 µl dan dihomogenkan dengan vortex.
Suspensi diinkubasi selama 10 menit di dalam water bath pada suhu 65oC. Setelah itu, ditambahkan 500 µl etanol (96-100%), dihomogenkan dengan vortex dan disentrifus selama 2 menit dengan kecepatan 10000 rpm. Supernatan yang dihasilkan dipipet dan dimasukkan ke dalam kolom mini yang sudah dipasang pada collection tube, kemudian disentrifus 8000 rpm selama 1 menit. Sebanyak 500 µl buffer AW1 ditambahkan ke dalam kolom mini yang masih terpasang pada collection tube dan disentrifus 8000 rpm selama 1 menit.
Tahap selanjutnya adalah kolom mini dipindahkan ke dalam collection tube yang baru dan ditambahkan dengan 500 µl buffer AW2 ke dalam kolom mini, kemudian disentrifus 13000 rpm selama 3 menit. Kolom mini dipindahkan kembali ke dalam
collection tube dan disentrifus kembali selama 1 menit 13000 rpm. Kolom mini dipindahkan ke dalam tabung mikrosentrifus steril dan ditambahkan ke dalamnya sebanyak 80 µl buffer AE yang kemudian disentrifus 8000 rpm selama 1 menit. Supernatan yang diperoleh pada tabung mikrosentrifus merupakan isolat DNA yang akan digunakan pada tahap selanjutnya dan isolat tersebut disimpan pada freezer dengan sehu 20oC. Diagram alir proses ini dapat dilihat pada Lampiran 4. Kemudian kemurnian isolat DNA yang dihasilkan dapat diketahui dengan mengukurnya pada spektrofotometer. Gambar penggunaan kolom mini dan collection tube dapat dilihat pada gambar berikut ini (Gambar 10.).
(a) (b)
Gambar 10. Proses isolasi/ekstraksi DNA dengan menggunakan metode kit komersial, (a) penambahan buffer AW1 ke dalam kolom mini, (b) hasil sentrifugasi setelah penambahan AW1 dan supernatan yang terdapat di bawah dibuang untuk selanjutnya filter dicuci kembali dengan buffer AW2.
4.
Pengujian
Salmonella
Typhimurium dengan
Real-Time
PCR
Penggunaan real-time PCR dilakukan dengan membuat master mix terlebih dahulu kemudian diamplifikasi dengan memasukkan master mix tersebut ke dalam alat real-time
PCR.
a)
Pembuatan Master Mix
Master mix untuk pengujian dengan real-time PCR terdiri dari 7 µl buffer TE, 10 µl SsoFastTM EvaGreen® Supermix (terdiri dari 2x reaction buffer dengan dNTPs, Sso7d-fusion polymerase, MgCl2, EvaGreen dye, dan penstabil), dan masing-masing 0,5 µl
23
reverse dan forward primer InvA pada konsentrasi tertentu. Volume master mix keseluruhan untuk pengujian satu jenis template DNA adalah 18 µl. Bahan-bahan untuk membuat master mix tersebut dicampur dalam satu tabung mikrosentrifus 2 ml dan dihomogenkan dengan vortex. Untuk melakukan pengujian beberapa jenis template DNA, maka volume masing-masing bahan dikali dengan banyaknya jenis template DNA yang akan diuji pada real-time PCR dan dicampur di dalam tabung mikrosentrifus. Kemudian masing-masing 18 µl campuran master mix yang telah dibuat pada tabung mikrosentrifus tersebut diambil dan dimasukkan kedalam setiap well yang kemudian ditambahkan dengan 2 µl template DNA yang selanjutnya dihomogenkan pada MixMate PCR 96 dan dimasukkan pada alat real-time PCR.
b)
Amplifikasi
dengan
real-time
PCR
Masing-masing sebanyak 18 µl master mix dan 2 µl template/isolat DNA yang telah dibuat dimasukkan ke dalam setiap well pada plate 96 Well Reaction. Kemudian well
ditutup dengan PCR SealerTM Microseal® ‘B’ Film dan dihomogenkan dengan menggunakan MixMate PCR 96. Well dimasukkan ke dalam real-time PCR yang telah diatur dengan protokol PCR tertentu. Protokol PCR yang digunakan adalah pre-denaturasi pada suhu 95oC selama 1 menit, diikuti dengan 35 siklus denaturasi pada suhu 95oC selama 30 detik dan annealing pada suhu 58.1oC selama 1 menit. Primer elongasi dilakukan pada suhu 72oC selama 1 menit 30 detik dan final elongasi selama 10 menit pada suhu 72oC. Analisis melting curve dari produk akhir PCR dilakukan sebanyak 81 siklus pada suhu 55oC selama 10 detik. Kemudian dilakukan running dengan real-time
PCR.
5.
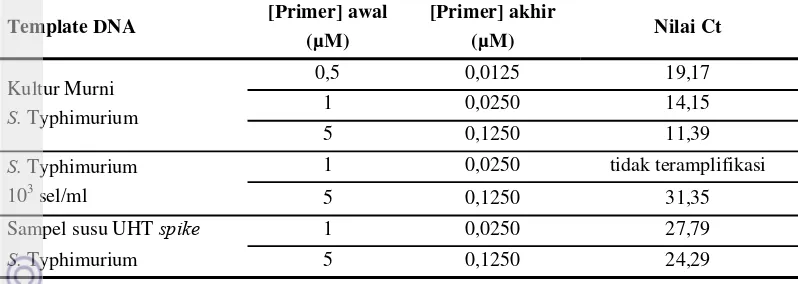
Penentuan Konsentrasi Primer
Penentuan konsentrasi primer pada penelitian ini dilakukan dengan cara menguji primer dengan konsetrasi 0,0125; 0,025; dan 0,125 µM pada sampel kultur murni Salmonella
Typhimurium, menguji primer dengan konsentrasi 0,025 dan 0,125 µM untuk sampel suspensi 103 sel/ml Salmonella Typhimurium dan sampel susu UHT spike Salmonella
Typhimurium ke dalam real-time PCR dengan melalui dua tahap yang telah dijelaskan pada sub bab sebelumnya. Kemudian dipilih konsentrasi primer yang sesuai/tepat. Konsentrasi primer yang sesuai/tepat adalah konsentrasi primer yang menghasilkan nilai Ct yang paling rendah dan tanpa menghasilkan atau seminimal mungkin menghasilkan primer-dimer pada kurva puncak pelelehan (Pestana et al. 2010).
6.
Penentuan Spesifisitas Primer (Ahmed
et al.
2009)
Isolat DNA kultur murni Salmonella Typhimurium sebagai kontrol positif dan isolat DNA Shigella sonnei sebagai kontrol negatif diuji dengan real-time PCR. Pengujian dengan
real-time PCR telah dijelaskan pada sub bab sebelumnya. Penentuan spesifisitas dilakukan dengan menggunakan primer InvA forward dan reverse yang telah dianalisis dengan Basic Local Alignment Search Tool (NCBI 2011) dimana sekuen oligonukleotida primer yang digunakan tersebut telah spesifik untuk Salmonella dan tidak sesuai dengan sekuen gen pada patogen tertentu yang biasa terdapat pada susu. Kemudian setelah pengujian dengan real-time PCR selesai, dilakukan analisis terhadap kurva puncak pelelehan (melt peak curve) dan kurva amplifikasi yang dihasilkan. Jika primer yang digunakan spesifik untuk Salmonella
nilai Tm pada melt peak curve sedangkan Salmonella Typhimurium akan teramplifikasi dan menghasilkan nilai Tm.
7.
Pengkuantifikasian
Salmonella
Typhimurium pada Sampel Pangan
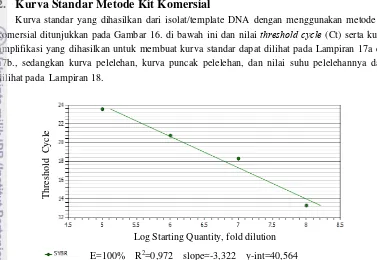
Proses pengkuantifikasian dilakukan dengan membuat kurva standar yang menghubungkan nilai Ct yang diperoleh dengan real-time PCR pada sumbu-y dan log konsentrasi mikroba pada sumbu-x. Kurva standar dibuat dengan mengencerkan 100 µl kultur murni Salmonella Typhimurium ke dalam 9 ml larutan pengencer NaCl 0,85%. Suspensi tersebut dihitung kandungan sel dalam setiap ml pengencer dengan hitungan mikroskopi pada petroff-hausser. Setelah diperoleh sejumlah 108 sel/ml dalam suspensi, maka dilakukan pengenceran hingga 103 sel/ml. Kemudian dilakukan isolasi DNA dan pengujian dengan real-time PCR sampai diperoleh nilai Ct dan kurva standar yang menghasilkan persamaan garis dengan efisiensi 96-110%; dan slope -3,1 hingga -3,6 (Pestana et al. 2010). Efisiensi merupakan faktor penting untuk setiap metode kuantitatif PCR yang reliable (Siebert 1999). Efisiensi amplifikasi dapat dihitung dengan menggunakan persamaan:% E = [(10-1/slope)-1] x 100%
Nilai Threshold Cycle (Ct) dari hasil pengujian sejumlah 105 sel/ml Salmonella
25
III.
HASIL DAN PEMBAHASAN
A.
Kultur Murni Hasil Pengecekan Kemurnian
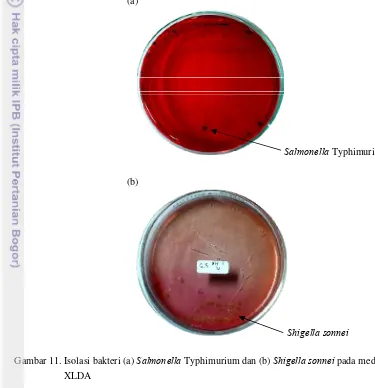
Hasil goresan kuadran kultur murni ke dalam media Xylose Lysine Deoxycholate Agar (XLDA) menghasilkan koloni Salmonella Typhimurium berwarna merah muda dengan inti hitam di tengahnya dimana hal tersebut sesuai dengan yang dijelaskan pada Bacteriological Analytical Manual mengenai
Salmonella (Andrews dan Hammack 2011). Sedangkan Shigella sonnei membentuk koloni merah muda tanpa ada inti hitam (Health Protection Agency 2007). Hasil pengisolasian Salmonella
Typhimurium dan Shigella sonnei dapat dilihat pada Gambar 11. di bawah ini.
(a)
Salmonella Typhimurium
(b)
Shigella sonnei
Gambar 11. Isolasi bakteri (a) Salmonella Typhimurium dan (b) Shigella sonnei pada media XLDA
Masing-masing satu koloni spesifik tersebut kemudian diisolasi dalam Heart Infusion Broth dan diinkubasi selama 24 jam pada suhu 37oC sebagai kultur murni mikroba spesifik Salmonella
B.
Inokulasi Susu dengan Kultur Murni
Salmonella
Typhimurium dan
Shigella
sonnei
(Sampel Susu UHT
spike
)
Hasil hitungan secara mikroskopi dengan menggunakan petroff-hausser terhadap suspensi
Salmonella Typhimurium pada larutan pengencer NaCl 0,85% yaitu sebesar 1,4 x 108 sel/ml
Salmonella Typhimurium untuk proses isolasi DNA dengan metode kit komersial dan 1,6 x 107 sel/ml
Salmonella Typhimurium untuk proses isolasi DNA dengan metode pendidihan, sedangkan penghitungan terhadap Shigella sonnei yaitu sebesar 2,5 x 107 sel/ml. Ketiga penghitungan tersebut dapat dilihat pada Lampiran 5. Karena konsentrasi suspensi lebih besar dari 105 sel/ml maka dilakukan pengenceran terhadap suspensi tersebut hingga diperoleh konsentrasi suspensi sekitar 105 sel/ml. Setelah dilakukan pengenceran, jumlah Salmonella Typhimurium pada susu UHT yang akan diisolasi/diekstraksi dengan metode kit komersial yaitu sebanyak 7,0 x 104 atau 0,7 x 105 sel/ml, pada susu UHT yang akan diisolasi/diekstraksi dengan metode pendidihan sebanyak 8,0 x 104 atau 0,8 x 105 sel/ml Salmonella Typhimurium, dan jumlah Shigella sonnei pada susu UHT yang akan diisolasi/diekstraksi dengan metode kit komersial sebanyak 1,2 x 105 sel/ml.
Hasil penghitungan konsentrasi akhir Salmonella Typhimurium pada sampel susu UHT spike dan penghitungan awal mikroba natural pada susu UHT dengan metode konvensional dapat dilihat pada Lampiran 6. Hasil tersebut menunjukkan bahwa sampel susu UHT tidak mengandung mikroba
Salmonella Typhimurium dan ketika diinokulasi dengan Salmonella Typhimurium maka sampel susu UHT spike tersebut mengandung 3,9 x 104 CFU/ml (penghitungan ke-1) dan 2,6 x 104 CFU/ml (penghitungan ke-2) Salmonella Typhimurium yang tumbuh dan hidup di dalamnya. Penghitungan ke-1 merupakan tahapan inokulasi Salmonella Typhimurium ke dalam susu yang akan disolasi/ diekstraksi dengan metode kit komersial dan penghitungan ke-2 merupakan tahapan inokulasi
Salmonella Typhimurium yang akan disolasi/diekstraksi dengan metode pendidihan.
Konsentrasi Salmonella Typhimurium yang terkandung di dalam sampel susu UHT spike (104 CFU/ml) tidak sama dengan konsentrasi Salmonella Typhimurium yang ditambahkan dimana diukur dengan menggunakan pengukuran mikroskopi pada petroff-hausser (105 sel/ml). Hal tersebut dikarenakan penghitungan dengan petroff-hausser tidak dapat membedakan mikroba yang hidup dan yang telah mati sehingga Salmonella Typhimurium yang telah mati juga ikut terhitung bersama dengan sel yang hidup. Sedangkan metode konvensional pada media XLDA, Salmonella
Typhimurium yang terhitung adalah mikroba yang hidup saja. Sehingga hitungan konsentrasi
Salmonella Typhimurium yang ditambahkan lebih besar dibandingkan konsentrasi akhir pada sampel susu UHT spike.
C.
Isolat/Template DNA yang Dihasilkan
Satu hal penting yang dibutuhkan untuk mendeteksi suatu mikroba dengan menggunakan real-time PCR adalah DNA mikroba tersebut yang murni tanpa pengotor. Cara memperolehnya adalah dengan mengisolasi/mengekstraksi DNA dari dalam sel. Berbagai teknik ekstraksi DNA salah satunya metode pendidihan telah dikembangkan dari prinsip dasar tersebut, sehingga saat ini muncul berbagai teknik ekstraksi dan purifikasi DNA dalam bentuk kit, dimana prosesnya cukup mudah, cepat, dan sederhana (Sulandari dan Zein 2003).
1.
Isolasi/Ekstraksi DNA dengan Metode Pendidihan
27
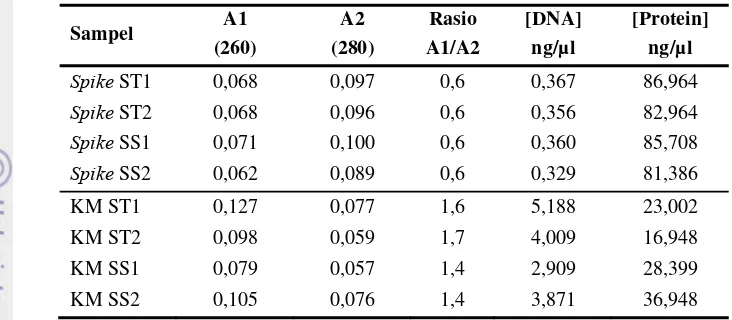
et al. 2007). Di bawah ini merupakan hasil pengukuran kemurnian isolat/template DNA yang diperoleh dengan metode pendidihan (Tabel 2.).
Tabel 2. Kemurnian isolat DNA Salmonella Typhimurium dan Shigella sonnei
dengan metode pendidihan
Sampel A1
(260) A2 (280) Rasio A1/A2 [DNA] ng/µl [Protein] ng/µl
Spike ST1 0,068 0,097 0,6 0,367 86,964
Spike ST2 0,068 0,096 0,6 0,356 82,964
Spike SS1 0,071 0,100 0,6 0,360 85,708
Spike SS2 0,062 0,089 0,6 0,329 81,386
KM ST1 0,127 0,077 1,6 5,188 23,002
KM ST2 0,098 0,059 1,7 4,009 16,948
KM SS1 0,079 0,057 1,4 2,909 28,399
KM SS2 0,105 0,076 1,4 3,871 36,948
Ket: Spike ST1 & 2 (Sampel susu UHT spikeSalmonella Typhimurium ulangan 1 dan 2); Spike SS1 & 2 (Sampel susu UHT spikeShigella sonnei ulangan 1 dan 2); KM ST1 & 2 (Kultur Murni Salmonella
Typhimurium ulangan 1 dan 2); KM SS1 & 2 (Kultur Murni Shigella sonnei ulangan 1 dan 2). A1 adalah nilai absorbansi pada panjang gelombang 260; A2 adalah nilai absorbansi pada panjang gelombang 280.
Berdasarkan hasil pengukuran spektrofotometri tersebut menunjukkan bahwa isolat DNA yang dihasilkan dari metode pendidihan belum murni yang artinya selain DNA, masih terdapat pengotor yang terkandung di dalam isolat tersebut salah satunya adalah protein. Hal ini ditunjukkan dengan nilai rasio yang tidak berada pada selang 1,8-2,0 baik pada sampel susu UHT
spike Salmonella Typhimurium dan Shigella sonnei maupun sampel kultur murni Salmonella
Typhimurium dan Shigella sonnei yang diambil dari media pengayaan HIB. Adanya pengotor pada isolat tersebut ditunjukkan pula dengan tingginya nilai konsentrasi protein yang dihasilkan.
Nilai konsentrasi protein yang dihasilkan pada sampel susu UHT spike jauh lebih tinggi dibandingkan dengan konsentrasi protein yang diperoleh dari sampel kultur murni mikroba spesifik. Hal tersebut menunjukkan bahwa perlu adanya modifikasi metode pendidihan khusus untuk sampel pangan yang mengandung protein tinggi seperti susu. Selain mengandung protein, susu juga mengandung lemak, kation (kalsium/Ca2+), dan pengkelat yang tinggi dimana komponen pangan tersebut menjadi inhibitor pada pengujian dengan menggunakan real-time
PCR. Inhibitor tersebut dapat mengikat dan menurunkan aktivitas enzim polimerase, menyebabkan perubahan konformasi dalam DNA target, atau bersaing dengan primer untuk menempati primer binding sites (Lee et al. 2006 dan Siebert 1999).
Hasil spektrofotometri tersebut membuktikan bahwa tahap pengisolasian DNA dari suatu matriks pangan merupakan hal yang sangat kritis dan kompleks dalam menjalankan pengujian dengan menggunakan real-time PCR. Keberadaan komponen gizi pada pangan dapat menjadi inhibitor PCR dimana dapat memberikan efek yang bervariasi, tetapi secara umum inhibitor tersebut dapat mempersulit pendeteksian DNA bakteri yang memiliki konsentrasi rendah (Lee et al. 2006).
A260/A280 pada UV-1700 spektrofotometer di dalam sampel pangan keju mozarela yang kaya akan lemak dan kalsium sebagai inhibitor dimana inhibitor tersebut juga terkandung pada susu UHT sebagai sampel pangan yang diuji pada penelitian ini. Penelitian Amagliani et al. (2006) tersebut menunjukkan bahwa metode pendidihan menghasilkan kemurnian isolat DNA yang kurang baik dimana nilai rasio A260/A280 lebih rendah dari 1,8.
Hal tersebut terbukti pada pengujian kuantifikasi Salmonella Typhimurium dengan real-time
PCR yang akan dibahas pada sub bab selanjutnya. Walaupun terdapat inhibitor di dalam isolat DNA yang diisolasi/diekstraksi dengan metode pendidihan, tetapi isolat tersebut masih dapat teramplifikasi contohnya pada pengujian penentuan spesifisitas primer InvA yang digunakan dan juga pada pengujian penentuan konsentrasi primer yang tepat dimana akan dibahas pada sub bab selanjutnya.
2.
Isolasi/Ekstraksi DNA dengan Kit Komersial
Keberadaan inhibitor baik dalam media kultur murni maupun dalam sampel pangan dapat menghambat proses amplifikasi dengan real-time PCR. Untuk mengatasinya, Chen et al. (1997) diacu dalam Lee et al. (2006) menggunakan metode kit komersial dalam proses isolasi/ekstraksi DNA Salmonella dari sampel susu nonpasteurisasi (raw milk), selain itu juga menggunakan proses pengayaan dan sentrifugasi untuk tahapan memanen/mengambil patogen. Begitu juga Omiccioli et al. (2009) dimana melakukan hal yang sama, namun menggunakan merk kit komersial yang berbeda. Isolat/template DNA yang dihasilkan diuji tingkat kemurniannya dengan menggunakan spektrofotometer dimana prinsipnya sama dengan pengukuran isolat DNA metode pendidihan. Hasil pengujiannya dapat dilihat pada Tabel 3. di bawah ini.
Tabel 3.