STUDI METAGENOMIK AKTINOMISET BERDASARKAN
GEN 16S rRNA DAN DETEKSI GEN
nif
H PADA TANAH DAN
AKAR EMPAT VARIETAS TANAMAN PADI DENGAN
TEKNIK DGGE
MAHYARUDIN
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Studi Metagenomik Aktinomiset Berdasarkan Gen 16S rRNA dan Deteksi Gen nifH Pada Tanah dan Akar Empat Varietas Tanaman Padi dengan Teknik DGGE adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Oktober 2014
Mahyarudin
RINGKASAN
MAHYARUDIN. Studi Metagenomik Aktinomiset Berdasarkan Gen 16S rRNA dan Deteksi Gen nifH Pada Tanah dan Akar Empat Varietas Tanaman Padi dengan Teknik DGGE. Dibimbing oleh YULIN LESTARI dan IMAN RUSMANA.
Aktinomiset merupakan salah satu mikrob yang sangat penting dikarenakan kemampuannya dalam menghasilkan senyawa metabolit seperti pemacu tumbuh tanaman. Aktinomiset endofit telah dilaporkan mampu memfiksasi nitrogen dan menghasilkan beberapa senyawa bioaktif seperti fitohormon, enzim, dan antimikrob pada tanaman padi yang mampu tumbuh pada berbagai tipe agroekosistem yang berbeda. Denaturing gradient gel electrophoresis (DGGE) digunakan untuk menganalisis keragaman mikrob. Analisis menggunakan DGGE mampu memisahkan gen yang memiliki ukuran DNA hasil amplifikasi dengan PCR yang sama tetapi komposisi sekuen basa DNA-nya berbeda. Deteksi keragaman aktinomiset dan bakteri yang memiliki kemampuan untuk memfiksasi nitrogen menggunakan penanda genetik seperti gen 16S rRNA dan nifH. Penelitian ini bertujuan mempelajari metagenom aktinomiset berdasarkan gen 16S rRNA dan deteksi gen nifH pada bakteri yang memiliki kemampuan memfiksasi nitrogen pada tanah dan akar empat varietas tanaman padi asal Indonesia dengan teknik DGGE.
DNA genom dari tanah dan akar empat varietas tanaman padi diisolasi menggunakan Soil DNA Isolation Kit dan Genomic DNA Mini Kit (Plant), selanjutnya gen 16S rRNA aktinomiset diamplifikasi menggunakan primer spesifik aktinomiset dengan teknik nested PCR. Amplifikasi PCR gen 16S rRNA aktinomiset menghasilkan produk berukuran 1087 pb dan 180 pb. Deteksi keragaman bakteri dianalisis menggunakan primer universal gen nifH untuk mendeteksi gen nifH pada bakteri pemfiksasi nitrogen dengan teknik direct PCR. Amplifikasi PCR gen nifH menghasilkan produk berukuran 360 pb. Analisis keragaman aktinomiset dan bakteri pemfiksasi nitrogen dengan DGGE pada gel poliakrilamida. Produk PCR hasil pemotongan pita pada gel poliakrilamida disekuensing dan dianalisis kekerabatannya dengan menggunakan software
bioinformatika MEGA 5.05.
Hasil analisis karakter fisika dan kimia tanah menunjukkan bahwa tanah sawah, tanah kering, dan tanah rawa (tiga tipe agroekosistem) merupakan tanah asam dan memiliki kandungan total nitrogen organik yang tergolong sedang. Profil DGGE berdasarkan gen 16S rRNA menunjukkan keragaman komunitas aktinomiset pada sampel akar (20-23 pita) lebih beragam dibandingkan dengan sampel tanah (12-18 pita). Keragaman komunitas aktinomiset memiliki kemiripan pada empat varietas tanaman padi dan tiga tipe agroekosistem. Profil pita DGGE dipotong dan di re-PCR menggunakan primer tanpa GC clamps untuk disekuensing. Sekuen DNA dibandingkan dengan database pada Genbank yang mengindikasikan hubungan kekerabatannya dengan mikrob lain. Hasil BLAST.N menunjukkan 17 pita memiliki hubungan kekerabatan dengan identitas maksimum berkisar 93-100% terhadap 5 genera dari aktinomiset seperti Geodermatophilus, Actinokineospora, Actinoplanes, Streptomyces and Kocuria. Genus
yaitu Actinokineospora diospyrosa NRRL B-24047 dan Actinoplanes friuliensis
HAG 010964. Geodermatophilus terdiri atas dua spesies yaitu G. terrae PB261 dan G. normandii CF 5/3, sedangkan Kocuria terdiri atas tiga spesies yaitu K. rhizophila DC2201, K. aegyptia YIM 70003 dan K. himachalensis K07-05.
Streptomyces merupakan genus yang paling dominan yang terdiri atas empat spesies yaitu S. alboniger DSM 40043, S. chiangmaiensis TA4-1, S. acidiscabies
RL-110, dan S. rapamycinicus ATCC 29253. Hasil penelitian ini menemukan bahwa spesies dari genus Streptomyces pada sampel tanah dan akar tanaman padi lebih bervariasi dibandingkan dengan genus dari aktinomiset lainnya, dengan dominansi S. alboniger dan S. acidiscabies pada hampir seluruh sampel. Pita 1 dan 6 memiliki indikasi sebagai novel species karena kedua sekuens DNA ini memiliki identitas maksimum kemiripan <97% dengan K. rhizophila DC2201 (96%) dan G. terrae PB261 (93%).
Analisis keragaman komunitas bakteri berdasarkan gen nifH dengan DGGE menunjukkan bahwa keragaman komunitas bakteri tanah (12-14 pita) yang memiliki gen nifH lebih tinggi dibandingkan dengan keragaman komunitas bakteri yang memiliki gen nifH pada akar tanaman padi (7-11 pita). Keragaman komunitas bakteri pemfiksasi nitrogen memiliki kemiripan pada empat varietas tanaman padi dan tiga tipe agroekosistem. Analisis sekuen DNA menggunakan BLAST.N menunjukkan bahwa lima pita memiliki hubungan kekerabatan pada gen nifH dari uncultured bacterium clone J50, uncultured bacterium clone clod-38, dan uncultured bacterium clone BG2.37 dengan identitas maksimum masing-masing yaitu 99%, 98%, dan 92%. Analisis dengan BLAST.X menunjukkan bahwa lima pita berkerabat dekat dengan sekuen protein dinitrogenase reduktase dari uncultured bacterium, nitrogenase iron protein dari uncultured bacterium dan
nifH Bradyrhizobium japonicum dengan identitas maksimum masing-masing yaitu 97%, 99%, dan 85%.
SUMMARY
MAHYARUDIN. Metagenomic Study of Actinomycetes Based on 16S rRNA Genes and Detection of nifH Genes in Soil and Roots of Four Rice Cultivars Using DGGE. Supervised by YULIN LESTARI and IMAN RUSMANA.
Actinomycetes are one of important microbes which have capability to produce various metabolite compounds such as plant growth promoter. Endophytic actinomycetes have been reported to fix nitrogen and produce several bioactive compounds such as phytohormone, enzyme, and antimicrobes in rice plant which can grow at several different types of agro-ecosystems. The denaturing gradient gel electrophoresis (DGGE) was used to analyse the microbial diversity. DGGE analysis can separate genes which have the same size of PCR amplified DNA but have different base sequences composition. Detection of actinomycetes diversity and bacteria which have capability to fix nitrogen using genetic marker i.e. 16S rRNA and nifH genes. The research was conducted to study the metagenomic of actinomycetes based on 16S rRNA gene and nifH gene in soil and roots of four rice cultivars using DGGE technique.
DNA genome from soil and roots of four rice cultivars were isolated using
Soil DNA Isolation Kit and Genomic DNA Mini Kit (Plant), followed by PCR amplification of actinomycetes 16S rRNA genes using specific primer of actinomycetes with nested PCR technique. The PCR amplification of actinomycetes 16S rRNA genes produced a product size of 1087 bp and 180 bp. Detection of bacterial diversity was analysed using bacterial universal primer of
nifH gene to detect nifH gene in nitrogen fixing bacteria with direct PCR technique. The PCR amplification of nifH gene produced a product size of 360 bp. Diversity analysis of actinomycetes and nitrogen fixing bacteria were conducted using DGGE on polyacrylamide gel. PCR product of excised bands resulted from polyacrylamide gel were sequenced and analyzed by bioinformatics software (MEGA 5.05).
The analysis of soil physical and chemical characteristics showed that irrigated, dry and swampy tidal soils (three type of agroecosystem) were acid soil and have a moderate of organic nitrogen content. The DGGE profile based on 16S rRNA showed that the diversity of actinomycetes in soil sample (12-18 bands) was lower than rice plant roots (20-23 bands). The diversity of actinomycetes community were similar in four variety of rice plant and three type of agroecosystem. The bands profile were excised, followed by re-PCR using primer without GC clamps and partially sequenced. The DNA sequences result were compared to GenBank database which indicating their identity with closely related microbes. The BLAST.N results showed that 17 bands were closely related ranging from 93% - 100% of maximum identity with 5 genera of actinomycetes, such as Geodermatophilus, Actinokineospora, Actinoplanes, Streptomyces and Kocuria. Actinokineospora and Actinoplanes only cosisted of one species i.e. Actinokineospora diospyrosa NRRL B-24047 and Actinoplanes friuliensis HAG 010964, respectively. Geodermatophilus consisted of two species i.e. G. terrae PB261 and G. normandii CF 5/3. Kocuria consisted of three species i.e. K. rhizophila DC2201, K. aegyptia YIM 70003 and K. himachalensis K07-05.
S. alboniger DSM 40043, S. chiangmaiensis TA4-1, S. acidiscabies RL-110, and
S. rapamycinicus ATCC 29253. The study found that Streptomyces species in soil and roots of rice plant were more varied than other genera, with a dominance of
S. alboniger and S. acidiscabies in almost all of samples. Band 1 and 6 have an indication as novel species with <97% maximum identity, and clustered with
K. rhizophila DC2201 (96%) and G. terrae PB261 (93%), respectively.
Diversity of bacterial community analysis based on nifH gene using DGGE showed that diversity of soil bacterial community (12-14 bands) which have nifH genes was higher than diversity of bacterial community in rice plant roots (7-11 bands). The diversity of bacteria which have nifH genes was similar in four variety of rice plant and three type of agroecosystem. Analysis using BLAST.N showed that five bands were closely related with nifH gene from uncultured bacterium clone J50, uncultured bacterium clone clod-38, and uncultured bacterium clone BG2.37 with maximum identity 99%, 98%, and 92%, respectively. Analysis using BLAST.X showed that five bands were closely related with protein sequence of dinitrogenase reductase from uncultured bacterium, nitrogenase iron protein from uncultured bacterium and nifH of
Bradyrhizobium japonicum with maximum identity 97%, 99%, and 85%, respectively.
Keywords: Actinomycetes, Bacteria, DGGE, 16S rRNA gene, nifH gene, Rice Plant.
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
STUDI METAGENOMIK AKTINOMISET BERDASARKAN
GEN 16S rRNA DAN DETEKSI GEN
nif
H PADA TANAH DAN
AKAR EMPAT VARIETAS TANAMAN PADI DENGAN
TEKNIK DGGE
MAHYARUDIN
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Mikrobiologi
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Studi Metagenomik Aktinomiset Berdasarkan Gen 16S rRNA dan Deteksi Gen nifH Pada Tanah dan Akar Empat Varietas Tanaman Padi Asal Indonesia dengan Teknik DGGE
Nama : Mahyarudin NIM : G351120131
Disetujui oleh Komisi Pembimbing
Dr Ir Yulin Lestari Ketua
Dr Ir Iman Rusmana, MSi Anggota
Diketahui oleh
Ketua Program Studi Mikrobiologi
Prof Dr Anja Meryandini, MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan November 2013 hingga Mei 2014 ini ialah keragaman aktinomiset dan bakteri pemfiksasi nitrogen, dengan judul Studi Metagenomik Aktinomiset Berdasarkan Gen 16S rRNA dan Deteksi Gen nifH Pada Tanah dan Akar Empat Varietas Tanaman Padi Asal Indonesia dengan Teknik DGGE.
Terima kasih penulis ucapkan kepada Ibu Dr Ir Yulin Lestari dan Bapak Dr Ir Iman Rusmana, MSi selaku pembimbing yang telah memberikan nasehat, saran, motivasi, waktu luang untuk konsultasi, serta solusi dari setiap permasalahan yang dihadapi penulis selama melaksanakan penelitian dan penyusunan karya ilmiah ini. Selain itu penulis ucapkan terima kasih kepada penguji luar komisi Dr Munti Yuhana, SPi MSi dan Dr Nisa Rachmania Mubarik, MSi selaku Wakil Ketua Program Studi Mikrobiologi IPB, yang telah memberikan masukan pada saat ujian sidang tesis untuk membuat karya ilmiah ini menjadi lebih baik. Kepada DIKTI melalui Beasiswa Unggulan 2012/2013 terima kasih atas kepercayaannya untuk memberikan beasiswa kuliah selama menempuh pendidikan pascasarjana di IPB, dan terima kasih atas proyek penelitian dari Pusat Studi Biofarmaka a.n. Dr Ir Yulin Lestari sehingga penelitian yang penulis lakukan dapat terlaksana dengan baik.
Penulis juga ingin menyampaikan terima kasih kepada staf laboratorium Mikrobiologi, yaitu Bapak Jaka dan Mbak Heni. Ungkapan terima kasih juga penulis sampaikan kepada Kak Sari, Kak Sipri, Randi, Mbak Dina, Mbak Anja, Mbak Gegek, Asril, Hendri, Yeni, Mei, Eja, Vita dan Bu Sumarsih atas bantuan dan motivasinya selama penelitian. Kepada teman-teman Pascasarjana Mikrobiologi IPB 2012 terima kasih atas kebersamaan yang singkat dan sangat indah. Ungkapan terima kasih yang tak terhingga juga penulis ucapkan kepada orang tua tercinta Bapak Ashari Maidin, ibu Nurmawati, dan kedua saudara tersayang Feri Junaidi, Artika Mabrurah, serta seluruh keluarga besar, atas segala doa, dukungan, motivasi dan kasih sayangnya selama ini. Semoga penelitian ini dapat bermanfaat bagi kemajuan ilmu pengetahuan selanjutnya.
Bogor, Oktober 2014
DAFTAR ISI
DAFTAR TABEL xi
DAFTAR GAMBAR xi
DAFTAR LAMPIRAN xi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 3
Tujuan Penelitian 3
Manfaat Penelitian 3
Ruang Lingkup Penelitian 4
TINJAUAN PUSTAKA 4
Tanaman Padi 4
Fiksasi Nitrogen 4
Mikrob Endofit 8
Metagenom 10
METODE 12
Kerangka Penelitian 12
Waktu dan Tempat Penelitian 13
Pengambilan dan Sterilisasi Sampel 13
Isolasi DNA Genom Tanah dan Akar Tanaman Padi 13 Amplifikasi Gen 16S rRNA Aktinomiset Tanah dan Akar Tanaman Padi 14 Amplifikasi Gen nifH Bakteri Tanah dan Akar Tanaman Padi 15
Analisis DGGE 15
Sekuensing Gen 16 S rRNA dan Gen nifH 16
Konstruksi Pohon Filogeni 16
HASIL DAN PEMBAHASAN 16
Hasil 16
Pembahasan 27
SIMPULAN DAN SARAN 32
Simpulan 32
Saran 32
DAFTAR PUSTAKA 33
LAMPIRAN 40
DAFTAR TABEL
1 Keragaman aktinomiset pada berbagai tanaman 6
2 Keragaman aktinomiset pada berbagai tanaman 9
3 Konsentrasi dan kemurnian DNA hasil ekstraksi dari sampel tanah
rizosfer dan akar tanaman padi 17
4 Persentase kemiripan sekuen gen 16S rRNA aktinomiset endofit pada
sampel tanah rizosfer dan akar tanaman padi 21
5 Persentase kemiripan sekuen gen nifH pada sampel tanah rizosfer dan
akar tanaman padi dengan analisis BLAST.N 26
6 Persentase kemiripan sekuen gen nifH pada sampel tanah rizosfer dan
akar tanaman padi dengan analisis BLAST.X 26
DAFTAR GAMBAR
1 Tahapan kerja penelitian 12
2 Amplifikasi PCR gen 16S rRNA pada sampel tanah dan akar tanaman
padi. 18
3 (A) Profil pita DGGE hasil produk PCR gen 16S rRNA pada sampel tanah dan akar tanaman padi (kiri). Ilustrasi pita DGGE menggunakan piranti lunak Phoretix 1D (kanan), 1-17 merupakan pita yang dipotong
untuk analisis sekuen DNA 19
3 (B) Analisis pengelompokan kemiripan komunitas aktinomiset pada sampel tanah dan akar tanaman padi menggunakan data biner 19 4 Hasil amplifikasi ulang pita hasil DGGE dengan primer non GC clamps 20 5 Pohon filogenetik dari 17 sekuen gen 16S rRNA aktinomiset yang
diperoleh dari analisis DGGE 22
6 Amplifikasi PCR gen nifH pada sampel tanah dan akar tanaman padi (~360 pb) menggunakan primer polF (dengan GC clamps) dan polR 23 7 (A) Profil pita DGGE hasil produk PCR gen nifH pada sampel tanah dan
akar tanaman padi (kiri). Ilustrasi pita DGGE menggunakan piranti lunak Phoretix 1D (kanan), 1-5 merupakan pita yang dipotong untuk analisis
sekuen DNA 24
7 (B) Analisis pengelompokan kemiripan komunitas berdasarkan gen nifH pada sampel tanah dan akar tanaman padi menggunakan data biner 24 8 Hasil amplifikasi ulang pita hasil DGGE dengan primer non GC clamps 25 9 Pohon filogenetik dari 5 sekuen gen nifH yang diperoleh dari analisis
DGGE 27
DAFTAR LAMPIRAN
1 Deskripsi dan gambar varietas tanaman padi 40
2 Karakteristik fisik dan kimia tanah 48
PENDAHULUAN
Latar Belakang
Padi merupakan bahan makanan pokok penduduk Indonesia. Kebutuhan terhadap padi semakin meningkat seiring dengan pertambahan jumlah penduduk Indonesia yang mencapai 1.3% setiap tahunnya. Peningkatan kebutuhan terhadap padi tidak sebanding dengan angka produktivitas padi di Indonesia. Angka produksi padi di Indonesia pada awal tahun 2012 yaitu sebesar 68.96 juta ton gabah kering giling (GKG), sedangkan pada akhir tahun 2012 mengalami penurunan sebanyak 4.73% (0.66 juta ton) (BPS 2012). Penurunan angka produksi padi tersebut dapat disebabkan oleh penyakit, cekaman lingkungan, atau defisiensi salah satu unsur hara seperti nitrogen. Duan et al. (2012) menyatakan bahwa kebutuhan tanaman akan nitrogen lebih tinggi dibandingkan dengan unsur hara lainnya. Kekurangan suplai nitrogen dapat mengakibatkan pertumbuhan tanaman tidak optimum karena nitrogen merupakan faktor pembatas bagi produktivitas tanaman. Upaya alternatif yang dapat dilakukan untuk meningkatkan produktivitas tanaman padi yaitu dengan memanfaatkan mikrob endofit.
Mikrob endofit merupakan mikrob yang hidup didalam jaringan internal tumbuhan tanpa menyebabkan efek negatif terhadap inangnya (Hasegawa et al. 2006). Setiap tanaman dapat mengandung beberapa mikrob endofit penghasil senyawa metabolit sekunder yang diduga sebagai akibat koevolusi atau transfer genetik dari tanaman inangnya (Tan dan Zao 2001). Salah satu mikrob endofit yang berperan penting sebagai penghasil senyawa metabolit sekunder yaitu aktinomiset. Aktinomiset diketahui memiliki kemampuan menghasilkan beragam senyawa bioaktif. Sebanyak 22.500 senyawa bioaktif yang dihasilkan oleh mikrob, 45% dihasilkan oleh aktinomiset, 38% oleh fungi dan 17% oleh bakteri uniselular (Berdy 2005). Sifat aktinomiset endofit yang tidak berdampak negatif pada jaringan tumbuhan menunjukkan adanya hubungan simbiosis mutualisme antara aktinomiset endofit dan inangnya. Aktinomiset dapat memberikan keuntungan pada tanaman dengan memproduksi fitohormon, pelarut fosfat, pendegradasi selulosa, dan fiksasi nitrogen serta dapat mengontrol patogen dengan memproduksi metabolit sekunder (Tian et al. 2004; Nurkanto 2007, Khamna et al. 2010; Soe et al. 2010).
2
Penelitian terdahulu telah berhasil membuktikan bahwa aktinomiset endofit memiliki kemampuan untuk menstimulasi pertumbuhan tanaman padi. Kemampuan yang dimiliki aktinomiset tersebut yaitu mampu menambat N2 bebas
(Pratyasto 2012), mampu menghasilkan IAA (Yusepi 2011), melarutkan fosfat, siderofor, HCN, kitinase, serta mampu menghambat mikrob patogen seperti
Xanthomonas oryzae pv. oryzae yang merupakan penyebab penyakit hawar daun pada tanaman padi (Hastuti et al. 2012a).
Berbagai macam kemampuan aktinomiset endofit pada tanaman padi yang telah berhasil diketahui sangat terkait dengan keberadaan dan keragamannya di dalam jaringan tanaman inang. Tanaman padi di Indonesia memiliki beberapa tipe agroekosistem berbeda yang digunakan sebagai lahan pertanian, seperti lahan persawahan irigasi sangat baik ditanami padi varietas Ciherang (CHR) dan IR 64, lahan kering yang subur sangat baik ditanami padi varietas Situ Patenggang (STP), sementara itu dilahan pasang surut atau lebak sangat baik ditanami padi varietas Inpara (INR) (Suprihatno et al. 2009). Keragaman aktinomiset sangat bervariasi pada tanah yang berbeda (George et al. 2012). Tian et al. (2007) menyatakan bahwa Streptomyces spp. dan Nocardiodies sp. berhasil diisolasi dari tanaman padi dan Streptomyces merupakan genus yang paling banyak diisolasi dari batang serta akar tanaman padi asal provinsi Guandong, Cina Selatan. Penelitian terdahulu mengenai aktinomiset endofit yang dapat dikulturkan melaporkan bahwa jumlah populasi aktinomiset tertinggi pada tanaman padi yaitu pada saat masa vegetatif. Populasi tertinggi diperoleh dari bagian akar dengan dominansi pada genus Streptomyces berdasarkan identifikasi secara morfologi (Jelita 2012). Isolat Streptomyces endofit memiliki kemampuan untuk tumbuh pada media tanpa nitrogen dan memproduksi amonia yang mengindikasikan bahwa isolat tersebut mampu memfiksasi nitrogen pada tanaman padi (Sari 2014). Informasi mengenai pengaruh berbagai tipe agroekosistem terhadap komunitas aktinomiset pada tanah dan akar tanaman padi Indonesia masih belum diketahui. Selanjutnya, informasi mengenai bakteri yang berasosiasi dengan tanaman padi yang memiliki gen fungsional seperti gen nifH juga belum banyak diketahui. Masalah utama untuk mengetahui keragaman aktinomiset secara keseluruhan baik di tanah maupun di dalam jaringan tanaman padi yaitu hanya 1% mikrob yang dapat dikulturkan, sedangkan 99% lainya masih tidak bisa dikulturkan pada media buatan (Sekiguchi 2006). Solusi untuk mengatasi kesulitan dan keterbatasan yang terkait dengan teknik kultivasi melaluipendekatan secara metagenomik.
Prinsip analisis keragaman secara metagenomik dilakukan berdasarkan analisis DNA yang diambil langsung dari lingkungan, sehingga dapat diperoleh gambaran menyeluruh untuk suatu komunitas. Pendekatan ini sangat memungkinkan untuk menganalisis spesies-spesies yang tidak dapat dikulturkan di laboratorium. Penelitian keragaman aktinomiset pada batang dan akar tanaman padi asal Cina menunjukkan populasi aktinomiset lebih beragam menggunakan pendekatan molekuler dibandingkan dengan pendekatan kultivasi secara konvensional (Tian et al. 2007). DNA total yang telah berhasil diisolasi dapat diidentifikasi menggunakan penanda filogenetik seperti gen 16S rRNA dan recA (Stein et al. 1996). Beberapa teknik berbasis molekuler yang digunakan untuk menganalisis keanekaragaman bakteri adalah Denaturing Gradient Gel Electrophoresis (DGGE), Terminal Restriction Fragment Length Polymorphism
3 2000) dan Pyrosequencing (Fakruddin et al. 2013). Analisis keragaman genetika yang cepat dan sederhana untuk menelaah profil DNA pengkode 16S rRNA maupungen nifH hasil amplifikasi dapat dilakukan dengan teknik DGGE. Prinsip penggunakan teknik ini yaitu pemisahan fragmen DNA yang mempunyai panjang sama, tetapi komposisi sekuens pasangan basa yang berbeda (Fischer dan Lerman 1983). Pemisahan ini didasarkan pada pengurangan mobilitas elektroforesis dari molekul DNA yang terdenaturasi secara parsial di dalam gel poliakrilamida yang mengandung gradien DNA denaturant yang bertambah secara linear. Penggunaan teknik DGGE telah berhasil digunakan untuk menentukan keragaman komunitas mikrob yang kompleks (Muyzer dan Smalla 1998). Analisis keragaman menggunakan pendekatan metagenomik dengan teknik DGGE memungkinkan untuk mengetahui keragaman aktinomiset endofit secara keseluruhan serta mempunyai kemampuan untuk memfiksasi nitrogen pada tanah dan jaringan akar tanaman padi.
Perumusan Masalah
Aktinomiset endofit telah terbukti berperan penting bagi pertumbuhan tanaman padi. Salah satu faktor yang mempengaruhi hal tersebut yaitu keragaman aktinomiset endofit pada tanaman. Sampai saat ini penelitian mengenai keragaman aktinomiset lebih mengarah pada aktinomiset yang bersifat culturable
saja. Sekitar 99% mikrob termasuk aktinomiset tidak dapat dikulturkan secara in vitro sehingga data mengenai keragaman aktinomiset endofit secara keseluruhan dan keragaman mikrob atau aktinomiset endofit yang mampu memfiksasi nitrogen pada tanaman padi asal Indonesia baik yang bersifat culturable ataupun
unculturable belum tersedia.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengetahui keragaman genetik aktinomiset berdasarkan gen 16S rRNA dan keragaman bakteri yang mampu memfiksasi nitrogen berdasarkan gen nifH pada tanah dan akar empat varietas tanaman padi dengan menggunakan pendekatan metagenomik.
Manfaat Penelitian
Analisis keragaman genetik aktinomiset pada tanaman padi diharapkan mampu memberikan informasi ilmiah dasar tentang keragaman aktinomiset yang berkolonisasi pada tanaman padi, baik yang bersifat culturable ataupun
4
Ruang Lingkup Penelitian
Penelitian meliputi pengambilan dan sterilisasi sampel, isolasi DNA genom pada jaringan tanaman padi, amplifikasi gen 16S rRNA dan gen nifH, analisis keragaman menggunakan teknik DGGE dan konstruksi pohon filogenetik.
TINJAUAN PUSTAKA
Tanaman Padi
Padi merupakan tanaman yang memiliki karakteristik tipe rumput dengan umpun kuat, termasuk tumbuhan annual (berumur 1 tahun), tinggi tanaman bervariasi antara 1,5-2 m, helaian daun berbentuk garis (linearis) dengan tepi yang kasar dan panjangnya 15-80 cm. Tanaman padi memiliki malai dengan panjang 15-40 cm yang tumbuh keatas dengan ujung menggantung. Buah tanaman padi tersusun atas bulir yang memiliki bentuk beragam, panjang bulir 7-10 mm dan memiliki lebar ± 3 mm. Buah tanaman padi ini mengandung pati (van Steenis et al. 2008).
Pengelompokan tanaman padi secara taksonomi adalah sebagai berikut Dasuki (1991):
Dunia : Plantae
Kelas : Monocotyledoneae
Sub-Kelas : Commelinidae
Ordo : Cyperales
Famili : Poaceae
Genus : Oryza
Spesies : Oryzasativa
Fiksasi Nitrogen
Fiksasi nitrogen merupakan proses pengubahan dinitrogen (N2 atau N N)
menjadi amonia (NH3) yang berguna untuk pertumbuhan tanaman. Reaksi yang
terjadi pada semua mikrob pemfiksasi nitrogen dalam proses fiksasi gas nitrogen dikatalisis oleh enzim nitrogenase (Kaminski et al. 1998). Nitrogenase terdiri atas 2 jenis komponen protein yang sensitif terhadap oksigen. Komponen 1 (dinitrogenase) terdiri atas protein besi-molibdenum yang mengandung 2 subunit. Komponen 2 (dinitrogenase reduktase) terdiri atas protein besi-sulfur yang mentransferkan elektron pada dinitrogenase. Protein-protein ini beserta ATP, Mg2+, dan sumber elektron merupakan komponen-komponen yang esensial dalam aktivitas fiksasi N2 (Moat et al. 2002).
Nitrogenase memerlukan donor elektron dari adenosin-3-fosfat (ATP) untuk memulai proses fiksasi nitrogen. Elektron dihasilkan secara in vivo, baik secara oksidatif maupun fotosintetik. Siklus oksidasi-reduksi dimulai ketika
5 elektron carrier yang mentransfer elektron ke protein Fe dari nitrogenase. Dua molekul MgATP terikat pada Fe protein yang tereduksi dan dihidrolisis untuk menghasilkan elektron yang akan ditransfer ke protein FeMo. Proses reduksi molekul N2 terjadi pada protein FeMo melalui beberapa tahapan reaksi. Transfer
elektron harus terjadi sebanyak enam kali pada setiap molekul N2 yang difiksasi.
Oleh karena itu total ATP yang dibutuhkan untuk memfiksasi molekul N2 yaitu 12
ATP, akan tetapi nitrogenase juga mereduksi proton menjadi H2. Reaksi tersebut
menggunakan dua elektron, sehingga total energi yang digunakan untuk mereduksi molekul N2 menjadi NH3 yaitu 8 elektron yang ditransfer dan 16
MgATPs yang dihidrolisis (Cheng 2008).
Enzim nitrogenase merupakan enzim yang diekspresikan ketika N2
merupakan sumber nitrogen satu-satunya, karena gen yang menyandikan fiksasi nitrogen dihambat oleh sumber nitrogen yang disuplai secara eksogen (contoh: amonia) (White 2000). Kompleks nitrogenase mengandung dua protein yang terpisah. Komponen 1 (dinitrogenase) merupakan komponen yang terdiri atas empat sub unit 240 kDa yang disandikan oleh gen nifD dan nifK. Kofaktor besi-molibdenum (FeMo-co) dari dinitrogenase disintesis oleh beberapa gen nif (nif Q, B, V, N, E). Komponen 2 (dinitrogenase reduktase, disandikan oleh gen nifH) merupakan protein yang terdiri atas 2 subunit 60 kDa. Protein ini mengandung kluster besi-belerang (Fe4S4) pada bagian tengahnya (Moat et al. 2002). Gen yang
beperan mengekspresikan kemampuan fiksasi nitrogen (nif) telah dikaji dengan baik pada Klebsiella pneumonia (Madigan et al. 2012), sehingga regulasi gen-gen
nif pada bakteri ini dijadikan model yang sangat baik untuk bakteri yang memiliki kemampuan memfiksasi nitrogen. Regulasi gen-gen nif pada K. pneumoniae
diatur oleh operon nifLA. Protein NtrC mengaktivasi transkripsi operon nifLA dibawah kondisi nitrogen yang terbatas. Protein nifA merupakan regulator positif yang berperan sebagai faktor yang dibutuhkan untuk transkripsi gen nif. Produk gen nifL berpengaruh dengan protein nifA untuk mencegah aktivasi nifA ketika tersedianya produk fiksasi nitrogen (amonia atau asam amino) atau oksigen. Fungsi nifZ, W, U, S, X, dan T masih belum diketahui secara jelas. Akan tetapi, ada bukti yang menyatakan bahwa produk gen nifW dan nifZ terlibat dalam proses salah satu komponen nitrogenase dan produk gen nifX merupakan regulator positif pada regulasi nif ketika merespon adanya asam amino dan oksigen (Moat
et al. 2002).
Fragmen spesifik gen nifD dan nifH dari K. pneumoniae merupakan gen-gen yang memiliki sekuens yang konservatif pada semua mikrob pemfiksasi nitrogen (Kaminski et al. 1998). Gen nifH sering digunakan sebagai penanda molekuler suatu mikrob yang dapat memfiksasi nitrogen karena memiliki basis data sekuen non-ribosomal terbesar dari berbagai mikrob. Selain itu gen ini telah digunakan pada berbagai jenis primer dan mampu mengamplifikasi dengan baik sampel dari lingkungan maupun mikrooganisme secara langsung (Zehr et al.
6
juga memiliki situs variatif yang lebih bervariasi dibandingkan dengan gen nifD, sehingga penanda gen ini sangat baik digunakan untuk analisis keragaman bakteri pemfiksasi nitrogen.Analisis keragaman mikrob pemfiksasi nitrogen pada rizosfer dan akar rumput endemik (Lasiurus sandicus) yang tahan terhadap kekeringan asal gurun Thar, India telah dilakukan dengan menggunakan primer spesifik gen
nifH. Hasil penelitian tersebut berhasil menemukan dominasi suatu sekuens nifH yang berkerabat dekat dengan Pseudomonas pseudoalcaligens. Selain itu, beberapa sekuen nifH memiliki kesamaan dengan diazotrof yang dapat dikultivasi seperti Azospirillum brasilense, Rhizobium spp. dan berbagai macam bakteri pemfiksasi nitrogen yang tidak dapat dikulturkan (Chowdury et al. 2009). Dua aktinomiset endofit pada akar tanaman Casuarina equisetifolia asal Meksiko telah berhasil diisolasi dan termasuk ke dalam anggota genus Thermomonospora dan
Micromonospora. Kedua aktinomiset terebut memiliki kemampuan menambat nitrogen karena dapat tumbuh pada media tanpa nitrogen. Selain itu hasil amplifikasi dengan menggunakan primer spesifik gen nifH menunjukkan adanya kemiripan dengan gen nifHpada genus Frankia (Valdes et al. 2005).
Penelitian mengenai bakteri pemfiksasi nitrogen telah banyak dilakukan termasuk dengan teknik DGGE. Analisis keragaman dengan teknik DGGE telah mampu menggambarkan keragaman bakteri pemfiksasi nitrogen yang ada pada lingkungan. Penelitian yang ada saat ini lebih kearah bakteri pemfiksasi nitrogen yang ada pada tanah. Penelitian mengenai bakteri pemfiksasi nitrogen yang ada pada tanah rizosfer dan jaringan akar tanaman padi serta pengaruh tipe agroekosistem masih belum ada kajian lebih lanjutnya. Sehingga sangat penting untuk mengetahui keragaman bakteri pemfiksasi nitrogen yang ada pada tanah rizosfer dan jaringan akar tanaman padi agar dapat melengkapi informasi yang ada. Beberapa penelitian mengenai keragaman bakteri pemfiksasi nitrogen yang dianalisis dengan teknik DGGE dapat dilihat pada Tabel 1.
Tabel 1 Keragaman bakteri pemfiksasi nitrogen pada tanah rizosfer
Sampel Diazotrof Referensi
Tanah Rizosfer tanaman padi di Provinsi Fujian,
Cina
Uncultured nitrogen-fixing bacterium
Wartiainen et al. 2008 Uncultured bacterium clone nifH
gene. Rhizosphere soil Uncultured bacterium clone
Diazotrophic rhizosphere community, Bt-transgenic white spruce
Endophytic Azoarcus wild rice
Azoarcus kallar grass endophyte Diazotrophs associated with rice roots
Uncultured endophytic bacterium clone associated with maize
Azonexus caeni
Methylococcus capsulatus Bath
7 Tabel 1 Keragaman bakteri pemfiksasi nitrogen pada tanah rizosfer (Lanjutan)
Sampel Diazotrof Referensi
Tanah rizosfer tanaman
gandum (Sorghum bicolor) di
Brazil
Uncultured bacterium clone IS110 EU048166.1
Uncultured bacterium clone IPA74 EU048016.1
Klebsiella pneumoniae AY242355.1
Azohydromonas lata AB188122.1
Uncultured bacterium clone IS98 EU048155.1
Klebsiella pneumoniae AY242355.1
Uncultured bacterium clone IS92 EU048149.1
Uncultured bacterium clone IS41 EU048099.1
Uncultured bacterium clone DQ995905.1 Uncultured bacterium clone IPA69 EU048011.1
Coelho et al.
2009
Tanah pertanian hortikultura di Shanghai, Cina
Rhizobium sp. (GQ241353.1)
Bradyrhizobium japonicum (GQ289565.1)
Azospirillum brasilense (GQ161239.1)
Azospirillum amazonense (GU256445.1)
Azospirillum lipoferum (FQ311868.1)
Sphingomonas azotifigens (AB217474.1)
Burkholderia xenovorans (EF158805.1)
Azohydromonas lata (AB188122.1)
Azoarcus sp. BH72 (AM406670.1)
Dechloromonas sp. (AJ563286.1)
Zoogloea oryzae (AB201045.1)
Azonexus hydrophilus (EF626685.1)
Rhodocyclales Azospira oryzae (U97115.2)
Azoarcus indigens (U97118.2)
Sideroxydans lithotrophicus (CP001965.1)
Methylocaldum szegediense (DQ002937.1)
Acinetobacter sp. (EU693341)
Ectothiorhodospira sp. (HM149325.1)
γ-Proteobacterium BAL281 nifH isolate (AY972874.1)
Anabaena sp. (HQ836199.1)
Uncultured bacterium (EU048119.1) Uncultured bacterium (HQ335575.1 Uncultured bacterium (GU097336.1)
8
Mikrob Endofit
Mikrob endofit merupakan mikrob yang hidup di dalam jaringan tanaman, akar, batang, daun, dan buah selama periode tertentu dari siklus hidupnya (Tan dan Zou 2001). Mikrob endofit membentuk koloni dalam jaringan tanaman tanpa membahayakan inangnya. Pada satu jaringan tanaman kemungkinan ditemukan beberapa jenis mikrob endofit (Strobel dan Daisy 2003). Hampir setiap tanaman tingkat tinggi memiliki beberapa mikrob endofit yang mampu menghasilkan senyawa bioaktif atau metabolit sekunder. Salah satu mikrob endofit yang mulai dikembangkan terutama dalam bidang kesehatan dan pertanian yaitu aktinomiset. Aktinomiset merupakan kelompok bakteri gram-positif yang memiliki filamen dan tergolong bakteri yang memiliki komposisi basa nitrogen GC yang tinggi yaitu >55% (Miyadoh 1997). Sebagian besar aktinomiset membentuk spora dan hal ini dapat digunakan sebagai dasar identifikasi. Aktinomiset secara taksonomi termasuk dalam Kelas Actinobacteria, Sub-kelas Actinobacteridae, Ordo Actinomycetales, dan berdasarkan klasifikasi molekuler terbagi dalam 10 Sub-ordo (Stackebrandt et al. 1997).
Aktinomiset telah diketahui mampu menghasilkan beragam metabolit sekunder dengan beragam fungsi biologi seperti antimikrob dan enzim pendegradasi bahan organik. Oleh karena itu aktinomiset digunakan sebagai agen biokontrol dan mampu menginduksi tanaman menjadi tahan terhadap penyakit (Hasegawa et al. 2006). Beberapa penelitian telah menunjukkan hasil yang mendukung pendapat tersebut. Streptomyces spp. yang diisolasi dari rizosfer mampu menghasilkan senyawa metabolit yang dapat menghambat pertumbuhan mikrob patogen tular tanah (Lestari 2006). Streptomyces sp. juga dilaporkan mampu menekan perkembangan penyakit hawar daun bakteri pada tanaman padi yang disebabkan oleh X. oryzae pv. oryzae. Kemampuan isolat yang diperoleh ini juga menunjukkan spektrum penghambatan yang cukup luas karena mampu menghambat mikrob kompetitor lainnya, seperti bakteri Gram positif dan Gram negatif serta fungi patogen (Hastuti et al. 2012b). Aktinomiset endofit PM5 telah diketahui mampu menghasilkan senyawa antifungi alifatik dengan unit lakton dan keton karbonil yang dapat menghambat pertumbuhan miselia Pyricularia oryzae
9 Tabel 2 Keragaman aktinomiset pada berbagai tanaman
Tanaman inang Aktinomiset endofit Referensi
Culturable Unculturable
Akar dan daun tanaman padi
asal Provinsi Guangdong,
Cina
S. cyaneus S. cyaneus Tian et al. 2007
S. exfoliates S. lanatus S. aurantiacus S. galilaeus S. paresii Streptomyces sp.
S. glauciniger S. turgidiscabies S. kathirae S. thermocarboxydus. S. caviscabies S. laceyi
S. scabies Janibacter sp.
Actinomycete NK951 N. simplex
N. thermolilacinus Mycobacterium sp.
Actinomycetales bacterium M. anthracenicum M. chitae
M. rhodesiae M. fortuitum
Micromonospora sp.
Corynebacterium sp.
Rhodococcus sp.
Actinoplanes sp.
Frankia sp. Amycolatopsis sp.
Dactylosporangium sp. Tanaman tropis
asal Papua New Guinea
dan Pulau Solomon
10
Tabel 2 Keragaman aktinomiset pada berbagai tanaman (Lanjutan)
Tanaman inang Aktinomiset endofit Referensi
Culturable Unculturable
Akar tanaman tomat (Physalis
ixocarpa) asal Meksiko
Microbacterium sp. - Patrick et al. 2010
M. foliorum Cellulomonas sp.
C. xylanilitica
20 Tanaman padi asal India
Streptomyces albosporus - Gangwar
et al. 2012 S. cinereus S. flavus S. globisporus S. roseosporus S. hygroscopicus S. lavendulae S. viridis S. griseorubroviolaceus Actinopolyspora sp.
Micromonospora sp.
Nocardia sp.
Saccharopolyspora sp.
Akar tanaman dataran pasang surut asal Korea
Micrococcus - Bibi et al. 2012 Mycobacterium Microbacterium Daun tanaman Spermacoce verticillata (Rubiaceae)
Microbispora sp. - Conti et al. 2012
Streptomyces sp. Nocardia sp.
Metagenom
11 berkembang karena adanya perancangan (construction) vektor kloning gen seperti kromosom artifisial bakteri (Bacterial Artificials chromosomes (BACs) atau kosmid (Xu 2006; Babcock et al. 2007) yang memungkinkan untuk mengkloning dan mengekspresikan segmen DNA yang besar dan kompleks (Singh et al. 2008).
Prinsip analisis keragaman secara metagenomik berdasarkan analisis DNA yang diambil secara langsung dari lingkungan. Saat ini beberapa gen telah digunakan sebagai penanda molekuler bagi kelompok prokariot, dan yang paling banyak digunakan adalah gen penyandi 16S rRNA. Gen ini bersifat ubikuitus hanya pada kelompok prokariot sehingga dapat menjadi penanda universal yang spesifik untuk kelompok prokariot. Gen 16S rRNA dapat digunakan sebagai penanda molekuler karena molekul ini bersifat ubikuitus dengan fungsi yang identik pada seluruh organisme. Molekul ini juga dapat berubah sesuai jarak evolusinya, sehingga dapat digunakan sebagai kronometer evolusi yang baik (Stackebrandt dan Goebel 1994). Molekul 16S rRNA memiliki beberapa daerah yang memiliki urutan basa yang relatif konservatif dan beberapa daerah urutan basanya variatif. Perbandingan urutan basa yang konservatif berguna untuk mengkonstruksi pohon filogenetik universal karena mengalami perubahan relatif lambat dan mencerminkan kronologi evolusi bumi. Sebaliknya, urutan basa yang bersifat variatif dapat digunakan untuk melacak keragaman dan menempatkan galur-galur dalam satu spesies. Selain gen 16S rRNA, gen nif juga memiliki sekuens yang konservatif pada semua mikrob pemfiksasi nitrogen, sehingga dapat digunakan untuk mengetahui keragaman mikrob pada suatu sampel lingkungan (Kaminski et al. 1998).
Beberapa teknik berbasis molekuler yang digunakan untuk menganalisis keanekaragaman mikrob adalah Denaturing Gradient Gel Electrophoresis
(DGGE), Fluorescent in-situ Hybridization (FISH), Terminal Restriction Fragment Length Polymorphism (TRFLP) dan Amplified Ribosomal DNA Restriction Analysis (ARDRA) (Marsh et al. 2000). Analisis keragaman genetika yang sederhana untuk menelaah keanekaragaman dapat dilakukan dengan teknik ARDRA. ARDRA dan TRFLP merupakan metode yang paling sering digunakan, akan tetapi metode ini memiliki kelemahan yaitu teknik ARDRA memiliki keterbatasan dalam hal kepraktisan, dibutuhkan pustaka gen 16S rRNA dalam ukuran yang besar untuk menggambarkan keragaman mendekati kergaman yang sebenarnya di alam. TRFLP memiliki keterbatasan yaitu data yang kurang representatif karena fragmen yang dianalisis hanya pada bagian ujung 5’ dari produk amplifikasi saja, sehingga ada kemungkinan berbagai kelompok besar bakteri yang berbeda hanya memunculkan satu macam TRF (Pangastuti 2008). Oleh karena itu, dalam penelitian ini menggunakan teknik DGGE dikarenakan oleh kesederhanaan dan kepraktisan untuk menganalisis komunitas mikrob secara langsung. Selain itu penggunaan teknik ini dapat memisahkan fragmen yang memiliki panjang sama tetapi urutan basa berbeda. Dengan demikian, sekuen DNA yang berbeda, bahkan perbedaan hanya satu pasang basa nukleotida, akan muncul sebagai pita pada posisi yang berbeda di dalam gel poliakrilamida (Muyzer dan Smalla 1998).
12
(Sutiknowati 2011). Analisis DGGE juga digunakan untuk menganalisis keragam aktinomiset yang mengkolonisasi residu jerami padi pada tanah yang mengalami berbagai sistem rotasi tanaman di Mekong Delta, Vietnam (Dung 2010).
METODE
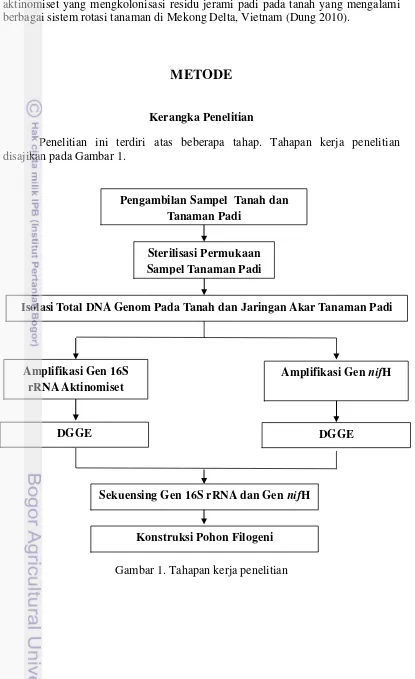
Kerangka Penelitian
Penelitian ini terdiri atas beberapa tahap. Tahapan kerja penelitian disajikan pada Gambar 1.
Gambar 1. Tahapan kerja penelitian Sterilisasi Permukaan Sampel Tanaman Padi
Isolasi Total DNA Genom Pada Tanah dan Jaringan Akar Tanaman Padi
Amplifikasi Gen 16S rRNA Aktinomiset
Amplifikasi Gen nifH
DGGE DGGE
Sekuensing Gen 16S rRNA dan Gen nifH
Konstruksi Pohon Filogeni Pengambilan Sampel Tanah dan
13 Waktu dan Tempat Penelitian
Penelitian dilakukan mulai Januari 2014 sampai Juni 2014. Pengambilan sampel tanah dan tanaman padi dilakukan di Bogor dan Cianjur, Jawa Barat. Isolasi DNA genom aktinomiset endofit, amplifikasi gen 16S rRNA dan gen nifH, analisis menggunakan teknik DGGE dilakukan di Laboratorium Mikrobiologi dan Laboratorium Biologi Terpadu, Departemen Biologi FMIPA Institut Pertanian Bogor (IPB).
Pengambilan dan Sterilisasi Sampel
Sampel tanah yang digunakan adalah tanah rizosfer pada masing-masing tanaman padi yang sehat. Bagian tanaman padi yang digunakan yaitu akar berumur ±4 minggu. Berdasarkan penelitian Jelita (2012) dinamika populasi aktinomiset tertinggi terdapat pada tanaman padi yaitu pada umur 4 minggu. Padi yang digunakan pada penelitian ini terdiri atas beberapa varietas yaitu Ciherang (CHR), IR 64, Situ Patenggang (STP) dan Inpara 2 (INR) (Lampiran 1). Sebanyak ±1 kg tanah pada rizosfer dari tiap varietas tanaman padi dikirim dan dianalisis di Balai Penelitian Tanah, Bogor untuk mengetahui karakteristik fisik dan kimiawi dari tanah tersebut.
Sterilisasi permukaan sampel untuk akar tanaman padi dilakukan berdasarkan metode Coombs dan Franco (2003) yang telah dimodifikasi oleh Jelita (2012). Tanaman yang telah dicuci bersih, disterilkan dengan merendam potongan tanaman dalam larutan etanol 70% selama 1 menit, natrium hipoklorit (NaOCl) 1% selama 5 menit, etanol 70% selama 1 menit dan dibilas menggunakan akuades steril sebanyak 3 kali.
Isolasi DNA Genom Tanah dan Akar Tanaman Padi
Isolasi DNA pada sampel tanah dilakukan sesuai dengan protocol Power
14
disentrifugasi pada kecepatan 13000 rpm selama 1 menit. Bead filter dipindahkan ke tabung mikro baru dan ditambahkan 50 µL larutan S5 lalu disentrifugasi pada kecepatan 13000 rpm selama 1 menit untuk elusi DNA yang telah murni.
Isolasi DNA pada jaringan akar tanaman padi dilakukan sesuai dengan protokol Genomic DNA Mini Kit, Plant (Geneaid, Shijr, TPE, TW). Sebanyak 0.1-0.5 g sampel batang atau daun yang telah disterilisasi, dihaluskan dengan penambahan nitrogen cair dan digerus sampai halus. Sampel yang telah halus ditransfer ke dalam tabung mikro 2 mL dan ditambahkan 700 µL larutan buffer
GP1 dan 5 µL RNAse, setelah itu dikocok dengan vorteks selama 1-2 menit. Campuran tersebut kemudian diinkubasi pada suhu 60 oC selama 15 menit bersama dengan buffer elusi. Selama inkubasi, tabung mikro harus dibolak-balik setiap 5 menit. Setelah proses inkubasi, larutan buffer GP2ditambahkan ke dalam campuran dan dikocok dengan vorteks. Setelah itu diinkubasi selama 5 menit di dalam es. Campuran yang telah diinkubasi kemudian dipindahkan ke dalam kolom filter yang telah diletakkan di atas tabung koleksi dan disentrifugasi dengan kecepatan 1000 rpm selama 1 menit. Supernatan dalam tabung koleksi dipindahkan ke dalam tabung mikro1.5 mL dan ditambahkan larutan buffer GP3 isopropanol 1.5x dari volume supernatan. Setelah itu campuran dikocok dengan vorteks selama 30 detik. Sebanyak 700 µL campuran dipindahkan ke dalam kolom GD yang telah diletakkan di atas tabung koleksi, kemudian disentrifugasi dengan kecepatan 13000 rpm selama 2 menit. Supernatan yang ada didalam tabung koleksi dibuang. Proses tersebut diulangi hingga campuran dalam tabung mikro habis. Sebanyak 400 µL larutan buffer W1 kemudian ditambahkan ke dalam kolom GD dan disentrifugasi dengan kecepatan 13000 rpm selama 30 detik. Supernatan di dalam tabung koleksidi buang. Setelah itu sebanyak 600 µ L
buffer penyuci ditambahkan ke dalam kolom GD dan disentrifugasi dengan kecepatan 13000 rpm selama 30 detik. Kemudian kolom GD disentrifugasi kembali dengan kecepatan 13000 rpm selama 3 menit untuk mengeringkan volume matriks. Kolom GD yang telah dikeringkan diletakkan di atas tabung mikro 1.5 mL. Sebanyak 30 µL buffer elusi ditambahkan ke dalam kolom GD dan dibiarkan hingga 20 menit kemudian disentrifugasi selama 30 detik dengan kecepatan 13000 rpm untuk elusi DNA yang telah murni. Visualisasi DNA dilihat dengan elektroforesis pada gel agarosa 1%. Kuantifikasi DNA dengan menggunakan NanoDrop 2000 (Thermo Scientific, Wilmington, DE, USA).
Amplifikasi Gen 16S rRNA Aktinomiset Tanah dan Akar Tanaman Padi
Amplifikasi DNA menggunakan teknik polymerase chain reaction (PCR) dengan T1-thermocycler (Biometra, Goettingen, Germany). Analisis gen 16S rRNA aktinomiset dilakukan dengan menggunakan dua tahap PCR, proses PCR
tahap pertama menggunakan primer spesifik aktinomiset 27F (5’ -AGAGTTTGATCCTGGCTCAG-3’) (Bruce et al. 1992) dan 16Sact1114R (5’ -GAGTTGACCCCGGCRGT-3’) (Martina et al. 2008). Reaksi PCR berlangsung dengan total volume 25 µL yang mengandung 12.5 µL GoTaq Green Master Mix
15 siklus ke 20, 55 oC untuk 10 siklus berikutnya), 2 menit proses ekstensi pada suhu
72 oC, dan 7 menit proses ekstensi akhir pada suhu 72 oC. Proses PCR berlangsung sebanyak 30 siklus (Zhang et al. 2013). PCR tahap kedua menggunakan primer bakteri P338F-GC (5’-CGCCCGCCGCGCG CGGCGGGCGGGGCGGGGGCACGGGGGGACTCCTACGGGAGGCAGCAG -3’) dan P518R (5’-ATTACCGCGGCTGCTGG-3’) (Overeas et al. 1997). Reaksi PCR berlangsung dengan total volume 50 µL yang mengandung 25 µL
GoTaq Green Master Mix 2X (Promega, Madison, WI, USA), 0.5 µL masing-masing primer (100 pmol), 1 µL produk PCR tahap pertama, dan 23.5 µL
nuclease free water. Proses PCR gen 16S rRNA yaitu pre-denaturasi (suhu 94 oC, 5 menit), denaturasi pada (suhu 92 oC, 30 detik), annealing (suhu 55 oC, 30
detik), proses ekstensi (suhu 72 oC, 30 detik), dan ekstensi akhir (suhu 72 oC, 7 menit). Proses PCR berlangsung sebanyak 30 siklus (Edenborn dan Sexstone 2007). Produk PCR dielektroforesis pada gel agarosa 1%, dan diamati pita tunggal DNA di G:BOX gel documentation (Syngene, Frederick, MD, USA) untuk memastikan fragmen DNA yang diamplifikasi pada ukuran ~1087 pb (tahap pertama) dan ~180 pb (tahap kedua).
Amplifikasi Gen nifH Bakteri Tanah dan Akar Tanaman Padi
Proses amplifikasi gen nifH bertujuan untuk mengamplifikasi fragmen internal yang berukuran 360 pb menggunakan primer PolF-GC (5’-CGCCCGC CGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGTGCGAYCCSARRGC BGGYATCGG-3’) dan primer PolR (5’-ATSGCCATCATYTCRCCGGA-3’) (Poly et al. 2001). Reaksi PCR berlangsung dengan total volume 25 µL yang mengandung 12,5 µL GoTaq Green Master Mix 2X (Promega, Madison, WI, USA), 2.5 µL masing-masing primer (10 pmol), 4 µL cetakan DNA (~100 ng µL
-1), dan 16 µL nuclease free water. Proses PCR gen 16S rRNA yaitu
pre-denaturasi (suhu 94 oC, 3 menit), denaturasi pada (suhu 94 oC, 1 menit),
annealing (suhu 55 oC, 1 menit), proses ekstensi (suhu 72 oC, 45 detik), dan ekstensi akhir (suhu 72 oC, 5 menit). Proses PCR berlangsung sebanyak 30 siklus. Visualisasi produk PCR dilakukan dengan elektroforesis gel agarosa 1% (w/v), dan diamati pita tunggal DNA di G:BOX gel documentation (Syngene, Frederick, MD, USA).
Analisis DGGE
Produk PCR gen 16S rRNA dan gen nifH (sekitar 30 µL) dimuat pada gel vertikal 1 mm yang mengandung 8% (w/v) gel poliakrilamida (akrilamida-bisakrilamida, 37.5:1) di Tris-asetat-EDTA (TAE) 1X. Gradien linier denaturan dari 30-70% untuk gen 16S rRNA (Edenborn dan Sexstone 2007) dan 40-65% untuk gen nifH (100% denaturant sesuai dengan 7 M urea dan 40% formamida deionisasi). Elektroforesis dilakukan pada suhu 60 oC dan tegangan 150 V, selama
5-6 jam dengan menggunakan D Code Universal Mutation Detection System (Bio-Rad, Hercules, CA, USA). Gel diwarnai selama 1 jam dengan 30 mL pewarna
SYBR Safe (Molecular Probes, Invitrogen, Carlsbad, CA, USA) dalam 270 mL larutan buffer TAE 1X dalam kondisi gelap. Gel dibilas dengan 500 mL buffer
16
G:BOX gel documentation (Syngene, Frederick, MD, USA). Hasil DGGE dianalisis menggunakan 1D Phoretix software (Total Lab) untuk mengestimasi jumlah total pita yang tampak pada gel poliakrilamida. Pita yang terlihat pada gel poliakrilamida dipotong menggunakan pisau bedah (scalpel) steril dan dimasukkan ke dalam tabung mikro yang berisi 100 µL ddH2O. Selanjutnya
tabung mikro tersebut diinkubasi pada suhu 4 oC selama overnight dan 60 oC selama 2 jam (Coelho et al. 2009; Perez et al. 2014). Sebanyak 10 µL (~50 ng µL-1) cetakan DNA di amplifikasi dengan menggunakan primer gen 16S rRNA
dan gen nifH yang sama, akan tetapi primer ini tidak mengandung GC clamps. Kondisi PCR sama seperti kondisi PCR sebelumnya.
Sekuensing Gen 16S rRNA dan Gen nifH
Produk PCR hasil analisis DGGE disekuensing sesuai dengan standar protokol menggunakan DNA sekuenser (ABI PRISM 3100) di Perusahaan Jasa Sekuensing First Base. Hasil sekuen nukleotida dibandingkan dengan GenBank database melalui NCBI BLAST.N melalui website (http://www.ncbi.nlm.nih.gov/BLAST). BLAST (Basic Local Alignment Search Tool) merupakan salah satu program dan basis data yang digunakan untuk pensejajaran sekuen. BLAST yang digunakan untuk analisis sekuen gen 16S rRNA dan gen nifH yaitu BLAST.N (BLAST Nukleotida). Analisis Protein yang disandikan gen nifH menggunakan BLAST.X.
Konstruksi Pohon Filogeni
Analisis filogenetik atau kekerabatan antar isolat aktinomiset endofit dilakukan berdasarkan pohon filogenetik menggunakan neighbor-joining method
serta software MEGA 5.0 (Tamura et al. 2011). Topologi dari konstruksi pohon filogenetik dievaluasi menggunakan analisis bootstrap dengan 1000 replikasi (Felsenstein 1985).
HASIL DAN PEMBAHASAN
Hasil
Karakteristik Tanah Rizosfer Tanaman Padi
17 ialah 2.03%, 11.82%, dan 4.83%. Kandungan total organik nitrogen pada tanah sawah, tanah gogo dan tanah rawa ialah 0.21%, 0.44% dan 0.35%. Rasio C/N pada tanah sawah, tanah gogo dan tanah rawa yaitu 10, 27, 14 (Lampiran 2). Efektivitas Sterilisasi Permukaan
Efektivitas sterilisasi permukaan menggunakan air rendaman terakhir hasil setrilisasi permukaan menunjukkan tidak adanya pertumbuhan aktinomiset pada media spesifik aktinomiset (Humic Acid-Vitamin B Agar) setelah inkubasi selama empat minggu, dengan demikian aktinomiset yang ada pada tanaman padi tersebut merupakan aktinomiset yang hidup di jaringan tanaman (endofit).
DNA Genom Tanah Rizosfer dan Akar Tanaman Padi
Hasil isolasi genom total sampel tanah rizosfer dan akar tanaman padi menunjukkan bahwa proses isolasi berhasil dilakukan. Hasil isolasi DNA menunjukkan bahwa konsentrasi DNA pada sampel tanah lebih tinggi dibandingkan dengan sampel akar tanaman padi. Konsentrasi DNA pada sampel tanah berkisar 32.1-37.7 µg/µ L sedangkan pada sampel tanaman berkisar 13.4-18.2 µg/µ L (Tabel 2). Kemurnian DNA genom sampel tanah dan akar tanaman padi berkisar 1.75-1.99.
Tabel 3 Konsentrasi dan kemurnian DNA hasil ekstraksi dari sampel tanah rizosfer dan akar tanaman padi
No Sampel Konsentrasi DNA (ng µL-1) A260/A280
1 S-CHR 37.3 1.98
2 S-IR64 37.7 1.99
3 S-STP 32.1 1.95
4 S-INR 37.1 1.93
5 R-CHR 18.2 1.92
6 R-IR64 13.4 1.75
7 R-STP 16.4 1.82
8 R-INR 17.6 1.91
Keterangan: S-CHR : DNA Genom Tanah Rizosfer Padi Varietas Ciherang
S-IR64 : DNA Genom Tanah Rizosfer Padi Varietas IR64
S-STP : DNA Genom Tanah Rizosfer Padi Varietas Situ Patenggang S-INR : DNA Genom Tanah Rizosfer Padi Varietas INPARA 2 R-CHR : DNA Genom Akar Tanaman Padi Varietas Ciherang R-IR64 : DNA Genom Akar Tanaman Padi Varietas IR64
R-STP : DNA Genom Akar Tanaman Padi Varietas Situ Patenggang R-INR : DNA Genom Akar Tanaman Padi Varietas INPARA 2
18
Komunitas Aktinomiset pada Tanah dan Akar Tanaman Padi
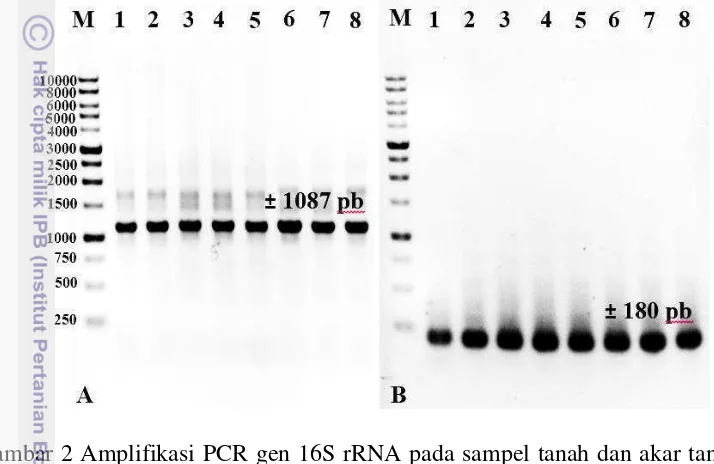
[image:34.595.97.454.194.426.2]Produk PCR gen 16S rRNA aktinomiset pada tanah dan akar tanaman padi dianalisis menggunakan teknik nested PCR. Teknik ini menggunakan dua kali PCR, proses PCR tahap pertama menghasilkan produk berukuran 1087 pb (Gambar 2A), sedangkan tahap kedua menghasilkan produk berukuran 180 pb (Gambar 2B).
Gambar 2 Amplifikasi PCR gen 16S rRNA pada sampel tanah dan akar tanaman padi. A) Produk PCR tahap pertama berukuran 1087 pb menggunakan primer 27F dan 16Sact1114R, B) Produk PCR tahap kedua 180 pb menggunakan primer p338F (dengan GC clamps) dan p518R. Marker 1 Kb; Sumur 1-8, S-CHR, R-CHR, S-IR64, R-IR64, S-STP, R-STP, S-INR, dan R-INR
Pada Gambar 2A terlihat pita berukuran diatas 1000 pb pada sampel tanah dan jaringan akar tanaman padi. Hasil tersebut mengindikasikan bahwa pada semua sampel terdapat aktinomiset. Hal ini karena primer yang digunakan merupakan primer spesifik aktinomiset, sehingga bakteri selain aktinomiset tidak dapat teramplifikasi. Hasil dari produk PCR tahap pertama dijadikan cetakan DNA untuk proses PCR tahap kedua. Pada semua sampel digunakan 1 µL produk PCR tahap pertama sebagai cetakan DNA untuk tahap kedua. Proses PCR tahap kedua ini menggunakan primer universal bakteri, yang menghasilkan fragmen berukuran 180 pb (Gambar 2B).
19
(A)
(B)
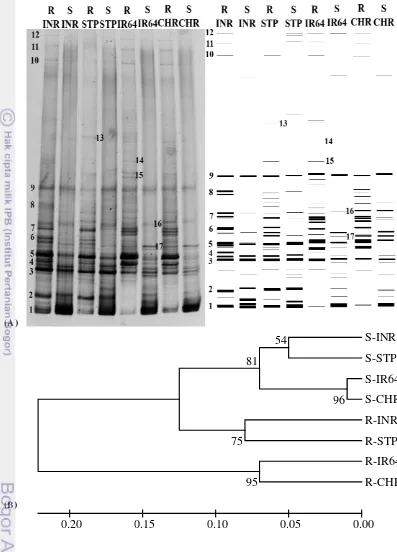
Gambar 3 (A) Profil pita DGGE hasil produk PCR gen 16S rRNA pada sampel tanah dan akar tanaman padi (kiri). Ilustrasi pita DGGE menggunakan piranti lunak Phoretix 1D (kanan), 1-17 merupakan pita yang dipotong untuk analisis sekuen DNA. (B) Analisis pengelompokan kemiripan komunitas aktinomiset pada sampel tanah dan akar tanaman padi menggunakan data biner
S-INR S-STP S-IR64 S-CHR R-INR R-STP R-IR64 R-CHR 96
54 81
95 75
0.00 0.05
0.10 0.15
[image:35.595.109.506.93.645.2]20
Analisis pola kemiripan komunitas aktinomiset dengan data biner (Gambar 3B) menunjukkan komunitas aktinomiset pada sampel tanah CHR memiliki kemiripan sebesar ±99% dengan sampel tanah IR64. Sementara itu, pola komunitas sampel tanah STP dan INR memiliki kemiripan sebesar ±95% tetapi klusternya terpisah dengan sampel tanah CHR dan IR64. Hal tersebut mengindikasikan terdapatnya perbedaan pola komunitas dengan sampel tanah CHR dan IR64. Pola yang sama juga ditunjukkan oleh sampel akar, komunitas aktinomiset pada sampel akar CHR memiliki kemiripan sebesar ±93% dengan sampel akar IR64. Sampel akar STP dan INR memiliki kemiripan pola komunitas yaitu sebesar ±92%, tetapi klusternya terpisah dengan sampel CHR dan IR64 yang mengindikasikan bahwa terdapat perbedaan pola kominitas dengan sampel akar CHR dan IR64. Pola kemiripan komunitas aktinomiset pada berbagai tipe agroekosistem menunjukkan bahwa masing-masing sampel tanah lebih mirip (95%-99%) jika dibandingkan dengan masing-masing sampel akar (92%-93%).
Hasil pemotongan pita pada gel DGGE didapatkan 17 pita yang dominan dan pita tersebut dipurifikasi dengan penambahan akuabides steril dan diinkubasi selama 24 jam, kemudian diinkubasi pada suhu 60 oC selama 30 menit. Hasil purifikasi diamplifikasi dengan primer yang sama akan tetapi tidak menggunakan
GC clamps dengan produk berukuran ±180 pb (Gambar 4). Hasil PCR tanpa GC clamps ini kemudian disekuensing untuk mengetahui runutan basa sehingga dapat dianalisis dan dibuat pohon filogeninya.
Gambar 4 Hasil amplifikasi ulang pita hasil DGGE dengan primer non GC clamps. Marker 1 Kb; Sumur 1-17, pita 1-17
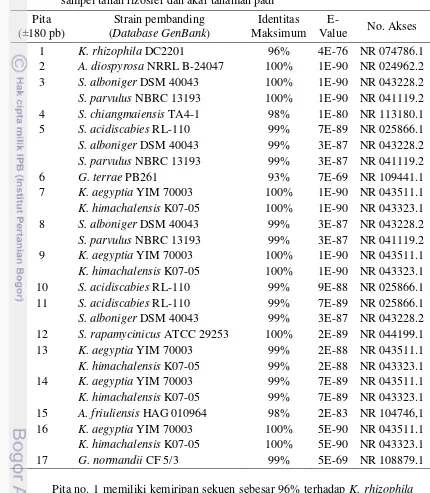
Sekuen DNA dari 17 pita hasil separasi menggunakan DGGE memiliki kekerabatan dengan 5 genera dari aktinomiset, seperti Geodermatophilus, Actinokineospora, Actinoplanes, Streptomyces dan Kocuria (Tabel 3). Genus
Actinokineospora dan Actinoplanes hanya ditemukan masing-masing satu spesies yaitu Actinokineospora diospyrosa NRRL B-24047 dan Actinoplanes friuliensis
HAG 010964. Genus Geodermatophilus terdiri atas dua spesies yaitu G. terrae
PB261 dan G. normandii CF 5/3. Genus Kocuria terdiri atas tiga spesies yaitu K. rhizophila DC2201, K. aegyptia YIM 70003 dan K. himachalensis K07-05. Genus
21
S. chiangmaiensis TA4-1, S. acidiscabies RL-110, dan S. rapamycinicus ATCC 29253.
Tabel 4 Persentase kemiripan sekuen gen 16S rRNA aktinomiset endofit pada sampel tanah rizosfer dan akar tanaman padi
Pita (±180 pb)
Strain pembanding (DatabaseGenBank)
Identitas Maksimum
E-Value No. Akses 1 K. rhizophila DC2201 96% 4E-76 NR 074786.1 2 A. diospyrosa NRRL B-24047 100% 1E-90 NR 024962.2 3 S. alboniger DSM 40043 100% 1E-90 NR 043228.2
S. parvulus NBRC 13193 100% 1E-90 NR 041119.2 4 S. chiangmaiensis TA4-1 98% 1E-80 NR 113180.1 5 S. acidiscabies RL-110 99% 7E-89 NR 025866.1
S. alboniger DSM 40043 99% 3E-87 NR 043228.2
S. parvulus NBRC 13193 99% 3E-87 NR 041119.2 6 G. terrae PB261 93% 7E-69 NR 109441.1 7 K. aegyptia YIM 70003 100% 1E-90 NR 043511.1
K. himachalensis K07-05 100% 1E-90 NR 043323.1 8 S. alboniger DSM 40043 99% 3E-87 NR 043228.2
S. parvulus NBRC 13193 99% 3E-87 NR 041119.2 9 K. aegyptia YIM 70003 100% 1E-90 NR 043511.1
K. himachalensis K07-05 100% 1E-90 NR 043323.1 10 S. acidiscabies RL-110 99% 9E-88 NR 025866.1 11 S. acidiscabies RL-110 99% 7E-89 NR 025866.1
S. alboniger DSM 40043 99% 3E-87 NR 043228.2 12 S. rapamycinicus ATCC 29253 100% 2E-89 NR 044199.1 13 K. aegyptia YIM 70003 99% 2E-88 NR 043511.1
K. himachalensis K07-05 99% 2E-88 NR 043323.1 14 K. aegyptia YIM 70003 99% 7E-89 NR 043511.1
K. himachalensis K07-05 99% 7E-89 NR 043323.1 15 A. friuliensis HAG 010964 98% 2E-83 NR 104746,1 16 K. aegyptia YIM 70003 100% 5E-90 NR 043511.1
K. himachalensis K07-05 100% 5E-90 NR 043323.1 17 G. normandii CF 5/3 99% 5E-69 NR 108879.1
Pita no. 1 memiliki kemiripan sekuen sebesar 96% terhadap K. rhizophila
DC2201. Pita no. 2 memiliki kemiripan sekuen sebesar 100% terhadap A. diospyrosa NRRL B-24047. Pita no. 3 dan 8 memiliki kemiripan sekuen sebesar 100% dan 99% dengan S. alboniger DSM 40043 dan S. parvulus NBRC 13193. Pita no. 4 memiliki kemiripan sekuen sebesar 98% dengan S. chiangmaiensis
TA4-1. Pita no. 5 memiliki kemiripan sekuen sebesar 99% dengan S. acidiscabies
[image:37.595.97.527.147.640.2]22
K. aegyptia YIM 70003 dan K. himachalensis K07-05. Pita n0. 10 dan 11 memiliki kesamaan 99% dengan S. acidiscabies RL-110. Pita no. 12 memiliki kesamaan 99% dengan S. rapamycinicus ATCC 29253. Pita no. 15 memiliki kesamaan 98% dengan A. friuliensis HAG 010964. Pita no. 17 memiliki kesamaan dengan G. normandii CF 5/3 sebesar 99%.
Gambar 5 Pohon filogenetik dari 17 sekuen gen 16S rRNA aktinomiset yang diperoleh dari analisis DGGE
Hasil analisis berdasarkan konstruksi filogenetik (Gambar 5) menunjukkan konsistensi bahwa pita no. 1 memiliki hubungan kekerabatan dengan K. rhizophila DC2201, pita no. 2 memiliki hubungan kekerabatan dengan A. diospyrosa NRRL B-24047, pita no. 3 dan 8 memiliki hubungan kekerabatan dengan S. alboniger DSM 40043, pita no. 4 memiliki hubungan kekerabatan dengan S. chiangmaiensis TA4-1, pita no. 5, 10, dan 11 memiliki hubungan kekerabatan dengan S. acidiscabies RL-110, pita no. 6 memiliki hubungan kekerabatan dengan G. terrae PB261, pita no. 7, 9, 13, 14, dan 16 memiliki hubungan kekerabatan dengan K. aegyptia YIM 70003 dan K. himachalensis
[image:38.595.82.495.175.665.2]23 ATCC 29253, pita no. 15 memiliki hubungan kekerabatan dengan A. friuliensis
HAG 010964, dan pita no. 17 memiliki hubungan kekerabatan dengan G. normandii CF 5/3.
Sekuen DNA dari pita yang memiliki kemiripan A. friuliensis HAG 010964 ditemukan pada sampel akar IR64 dan STP. Sekuen DNA dari pita yang memiliki kemiripan G. terrae PB261 hanya ditemukan pada seluruh sampel akar. Sekuen DNA dari pita yang memiliki kemiripan S. rapamycinicus ATCC 29253 ditemukan pada sampel akar CHR, IR64, INR, sampel tanah IR64 dan STP. Sekuen DNA dari pita yang memiliki kemiripan K. rhizophila DC2201, S. alboniger DSM 40043, S. acidiscabies RL-110, K. aegyptia YIM 70003 dan K. himachalensis K07-05 ditemukan di seluruh sampel tanah dan akar tanaman padi. Hasil ini mengindikasikan bahwa tipe agroekosistem (tanah) dan varietas tanaman padi (akar) yang berbeda tidak memberikan pengaruh terhadap keragaman aktinomiset, tetapi hanya memberikan pengaruh terhadap kelimpahan masing-masing aktinomiset pada setiap sampel. Kelimpahan spesies aktinomiset dapat dilihat dari ketebalan pita yang muncul pada gel poliakrilamida. Hasil ini juga menunjukkan bahwa komunitas aktinomiset yang termasuk dalam genus
Streptomyces pada sampel tanah dan akar empat varietas tanaman padi di Jawa Barat, Indonesia lebih bervariasi dibandingkan genus aktinomiset lainnya (Gambar 5).
Komunitas Bakteri Pemfiksasi Nitrogen pada Tanah dan Akar Tanaman Padi
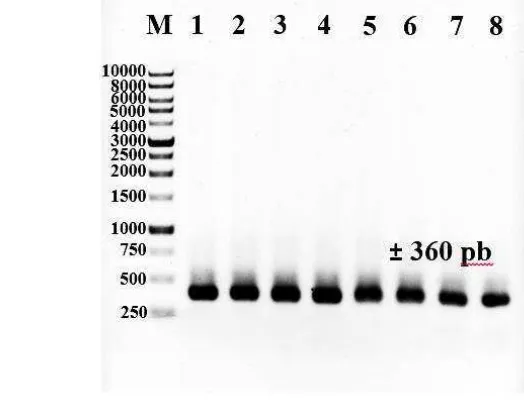
Produk PCR gen nifH pada sampel tanah dan akar tanaman menghasilkan fragmen DNA berukuran ~360 pb (Gambar 6) diseparasi menggunakan gel poliakrilamida yang mengandung denaturan berkisar 40-65%.
[image:39.595.168.430.474.674.2]24
(A)
(B)
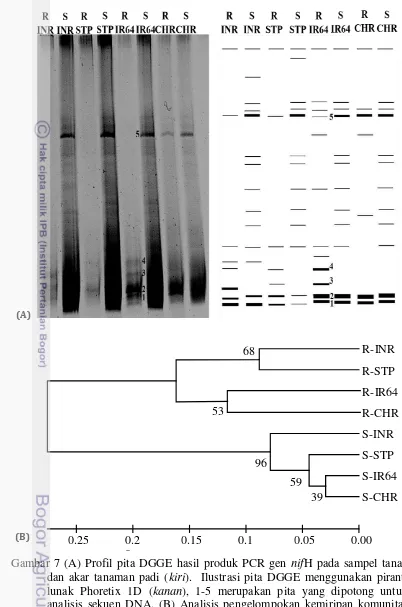
Gambar 7 (A) Profil pita DGGE hasil produk PCR gen nifH pada sampel tanah dan akar tanaman padi (kiri). Ilustrasi pita DGGE menggunakan piranti lunak Phoretix 1D (kanan), 1-5 merupakan pita yang dipotong untuk analisis sekuen DNA. (B) Analisis pengelompokan kemiripan komunitas berdasarkan gen nifH pada sampel tanah dan akar tanaman padi menggunakan data biner
R-INR R-STP R-IR64 R-CHR S-INR S-STP S-IR64 S-CHR 39
59 96
68
53
0.00 0.05
0.1 0.15
[image:40.595.74.476.81.688.2]25 Berdasarkan hasil analisis DGGE menunjukkan struktur komunitas bakteri pemfiksasi nitrogen pada sampel tanah (12-14 pita) lebih bervariasi dibandingkan sampel akar tanaman padi (7-11 pita) (Gambar 7A). Analisis pengelompokan kemiripan komunitas dengan data biner menunjukkan komunitas bakteri pemfiksasi nitrogen pada sampel tanah memiliki perbedaan dengan sampel akar. Hal ini dapat dilihat dari semua sampel tanah yang membentuk kluster terpisah dengan sampel akar, akan tetapi dapat dilihat pada (Gambar 7B) bahwa pola kemiripan komunitas bakteri pemfiksasi nitrogen pada berbagai tipe agroekosistem menunjukkan bahwa masing-masing sampel tanah lebih mirip (92%-97%) jika dibandingkan dengan masing-masing sampel akar (87%-91%). Komunitas bakteri pemfiksasi nitrogen pada sampel tanah CHR memiliki kemiripan sebesar ±97% dengan sampel tanah IR64. Sementara itu, pola komunitas sampel tanah STP memiliki kemiripan sebesar ±96% terhadap sampel tanah CHR dan IR64. Pola komunitas sampel tanah INR memiliki kemiripan sebesar ±92% pada sampel tanah STP, CHR dan IR64. Pola yang sama juga ditunjukkan oleh sampel akar, komunitas aktinomiset pada sampel akar CHR memiliki kemiripan sebesar ±91% dengan sampel akar IR64. Sampel akar STP dan INR memiliki kemiripan pola komunitas yaitu sebesar ±87%, tetapi klustern