i
PEMANFAATAN MARKA MOLEKULER DALAM
PEMULIAAN TANAMAN UNTUK MENDUKUNG
PENYEDIAAN KULTIVAR UNGGUL BARU
JARAK PAGAR (Jatropha curcas L.)
DARMAWAN SAPTADI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
ii
SURAT PERNYATAAN MENGENAI DISERTASI DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa disertasi Pemanfaatan Marka Molekuler dalam Pemuliaan Tanaman untuk Mendukung Penyediaan Kultivar Unggul Baru Jarak Pagar (Jatropha curcas L.) adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Bogor, Januari 2012
iii
ABSTRACT
DARMAWAN SAPTADI. Utilization of Molecular Marker in Plant Breeding to Support the Provision of New High Yielding Cultivar of Physic Nut (Jatropha curcas L.). Under supervised by SUDARSONO, ASEP SETIAWAN, BAMBANG HELIYANTO.
The main obstacle in the cultivation and commercialization of physic nut (Jatropha curcas L.) as a biodiesel plant is the unavailability of superior cultivars or hybrids with high yield and oil content. Assessment of molecular genetic diversity is important because it is a pre-requisite for plant breeding programs.This research was conducted to develop and exploit molecular marker in order to support genetic improvement of physic nut. Development of Simple Sequence Repeat (SSR) primer was conducted based on DNA sequences available in the GenBank DNA database and 28 primer pairs were yielded. Twenty four accessions of physic nut from Kebun Induk Jarak Pagar (KIJP) Pakuwon germplasm collection were assessed by SRR marker designed. The results show that there was no variability among accession tested. In addition, Random Amplified Polymorphic DNA (RAPD), Inter Simple Sequence Repeat (ISSR) marker was employed to asses genetic variability. Out of 31 RAPD and ISSR primers evaluated 8 RAPD and 4 ISSR primers produced scorable DNA markers and four primers (UBC 873, OPG 17, OPP 03 and OPQ 11) produced polymorphic bands. Genetic similarity coefficients ranged from the highest of 1.0 (between 3189-2 / PT13-2; MT7-1 / PT15-1; PT3-1, 2555-1 / SP8-1; 2555-1 / PT3-1) to the lowest of 0.6 (between 554-1/HS49-2) with a mean 0.9. SSR primer then tested for cross species amplification ability to the relative species of J. curcas i.e. J. integerrima, J. multifida, J. gossypifolia, J. podagrica) and study the relationship between these species was conducted. Out of 28 primers checked, 11 primers showed cross species amplification in all the species tested. Overall percentage of polymorphism (PP) among all species tested was 95% and the mean genetic similarity (GS) was found to be 0.34. Dendrogram showed that J. integerrima have the closest distance from J. curcas. Interspecific crossing between J. curcas x J. integerrima have been conducted and morphological characters were recorded. Variation between hybrids was not too large and generally intermediate between the parents. Molecular analysis using SSR markers, RAPD and ISSR conducted on 8 hybrids and their parents. Two markers (EU099522 and OPC 10) were polymorphic in both parents and co-inherited to all hybrids. EF612741 and EU099524 were specific to J. integerrima and J. curcas respectively and could be used for identification of hybrids through multiplex PCR.
Keywords: primer development, genetic variability, cross specific amplification, With such a low genetic diversity among physic nut accessions, breeding program using the analyzed accessions may not useful. Introduction of new accessions of physic nut and or interspecific crossing with its relative may be necessary to increase genetic diversity and improve genetic gain through breeding program.
iv
RINGKASAN
DARMAWAN SAPTADI. Pemanfaatan Marka Molekuler dalam Pemuliaan Tanaman untuk Mendukung Penyediaan Kultivar Unggul Baru Jarak Pagar (Jatropha curcas L.). Dibimbing oleh: SUDARSONO, ASEP SETIAWAN, BAMBANG HELIYANTO.
Hambatan utama dalam budidaya dan komersialisasi jarak pagar (Jatropha curcas L.) sebagai tanaman penghasil bahan bakar nabati adalah belum tersedianya kultivar unggul yang berdaya hasil dan berkadar minyak tinggi. Modal utama untuk perbaikan genetik adalah tersedianya keragaman genetik dari spesies yang bersangkutan. Informasi keragaman genetik jarak pagar belum memadai karena sebagian besar masih berdasar pengamatan secara morfologis. Penelitian ini dimaksudkan untuk mengembangkan dan memanfaatkan marka molekuler untuk mendukung pemuliaan jarak pagar.
Primer SSR telah dikembangkan berdasarkan informasi sekuen DNA yang tersedia pada basis data Genbank. Dua puluh delapan pasang primer spesifik SSR telah berhasil didesain menggunakan aksesi DNA asal jarak pagar yang ada di GenBank DNA database. Ekstraksi DNA pada penelitian ini menggunakan protokol standar CTAB yang dimodifikasi. Separasi dan visualisasi hasil amplifikasi menggunakan marka SSR dilakukan dengan polyacrilamide gel electrophoresis (PAGE) dan pewarnaan perak. Separasi dan visualisasi hasil amlifikasi menggunakan marka RAPD, ISSR dan SCAR dilakukan dengan elektroforesis gel agarosa dan ethidium bromida.
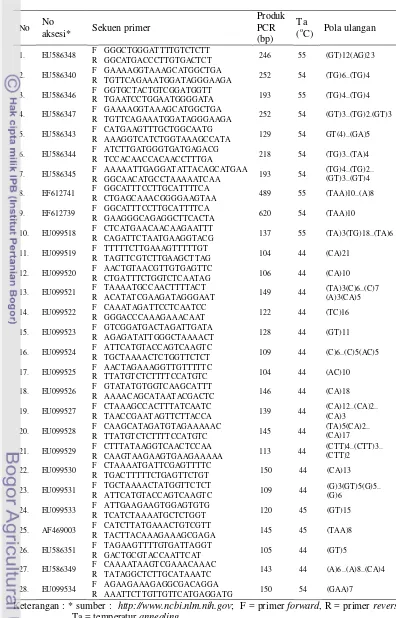
Dua puluh delapan pasang primer yang dikembangkan mampu menghasilkan marker SSR dari genom jarak pagar dan 14 pasang di antaranya menghasilkan marker SSR dari genom J. multifida. Sembilan belas pasangan primer yang dievaluasi menghasilkan 44 alel dengan ukuran produk amplifikasi berkisar antara 100 – 360 bp. Sebanyak 35 alel (79.5%) yang diamati merupakan alel yang polimorfik. Marker SSR yang didapatkan tidak polimorfik intra-aksesi jarak pagar atau intra-aksesi J. multifida tetapi polimorfik untuk inter-aksesi kedua spesies. MarkaSSR yang dihasilkan bersifat polimorfik untuk aksesi jarak pagar dengan aksesi J. multifida sehingga dapat digunakan sebagai marka untuk mendeteksi hasil persilangan F1 inter-spesies J. curcas x J. multifida.
v
Persentase polimorfisme paling rendah (0%) yaitu antara 3189-2/PT13-2; MT7-1/ PT15-1; PT3-1, 2555-1/ SP8-1; 2555-1/ PT3-1 dan paling tinggi (55.26%) yaitu antara 554-1/HS49-2 dengan rerata 15.87%. Pada batas kesamaan genetik di atas 80%, dendrogram dapat dibagi menjadi dua klaster di mana satu klaster terdiri satu aksesi yaitu HS 49-2 sedangkan klaster yang lainnya berangotakan semua aksesi yang lain. Berdasarkan marka SCAR, semua aksesi yang diuji termasuk dalam jarak pagar tipe Meksiko.
Penelitian berikutnya dilakukan untuk mengetahui kemampuan amplifikasi lintas spesies marka SSR yang telah dikembangkan dari J. curcas kepada spesies kerabatnya (J. integerrima, J. multifida, J. gossypifolia, J. podagrica) dan mengetahui derajat hubungan antara spesies-spesies tersebut. Dari 28 primer yang digunakan, 11 primer dapat diamplifikasi pada semua spesies yang diuji. Pasangan primer EU099519, EU099528 dan EU099525 tidak dapat diamplifikasi selain pada J. curcas. Persentase polimorfisme keseluruhan di antara semua spesies yang diuji adalah 95% dengan rerata kesamaan genetik sebesar 0.34. Persentase polimorfisme paling rendah (17.35) dengan kesamaan genetik paling tinggi (0.60) didapatkan antara J. podagrica vs J. multifida. Korelasi antara persentase polimorfisme dengan kesamaan genetik cukup baik yaitu bernilai 0.75. Analisis dendrogram menunjukkan bahwa jarak paling jauh ditemukan antara J. curcas dengan J. gossypifolia dan jarak paling dekat ditemukan antara J. podagrica dan J. multifida. Spesies yang mempunyai hubungan paling dkat dengan J. curcas adalah J. integerrima. Marka yang digunakan dapat diandalkan untuk membedakan antar spesies dan dapat digunakan untuk identifikasi hasil persilangan interspesifik secara molekuler.
Persilangan interspesifik antara J. curcas x J. integerrima telah berhasil dilakukan dan mendapatkan tanaman F1 yang telah memasuki fase generatif. Pengamatan terhadap beberapa karakter morfologis telah dilakukan terhadap individu-individu F1. Hasil pengamatan menunjukkan bahwa keragaman antar tanaman F1 tidak terlalu besar dan secara umum mempunyai karakter pertengahan (intermediate) antara kedua tetuanya. Individu F1 cenderung mengikuti sifat J. integerrima pada karakter bentuk percabangan, batang, bentuk karangan bunga serta pigmentasi pada daun, tangkai daun dan tangkai bunga. Karakter pada F1 yang mengikuti tetua J. curcas adalah bentuk daun. Analisis molekuler menggunakan marka SSR, RAPD dan ISSR dilakukan terhadap 8 indifidu F1 dan kedua tanaman tetua. Marka EU099522 dan OPC10 polimorf pada kedua tetua dan dapat diturunkan secara bersama (co-inherited) pada semua individu F1. Marka EF612741 dan EU099524 dapat digunakan untuk identifikasi hasil persilangan melalui multiplex PCR karena masing-masing spesifik untuk J. integerrima dan J. curcas. Keragaman antar kedua tetua tinggi (66%) sementara antar F1 keragamannya rendah (rerata 18%). Berdasarkan dendrogram, tetua J. integerrima berada pada klaster tersendiri (out grouped) sementara tetua J. curcas dan semua individu F1 berada pada satu klaster yang lain.
vi
sangat berguna dalam pemuliaan tanaman untuk mendapatkan kemajuan genetik yang signifikan. Marka molekuler yang dikembangkan pada penelitian ini dapat digunakan untuk mempercepat proses seleksi yang merupakan bagian utama dari kegiatan pemuliaan untuk perbaikan genetik jarak pagar.
vii
© Hak cipta milik Institut Pertanian Bogor, tahun 2012
Hak cipta dilindungi
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik atau tinjauan suatu masalah
b. Pengutipan tidak merugikan kepentingan yang wajar IPB
ix
PEMANFAATAN MARKA MOLEKULER DALAM
PEMULIAAN TANAMAN UNTUK MENDUKUNG
PENYEDIAAN KULTIVAR UNGGUL BARU
JARAK PAGAR (Jatropha curcas L.)
DARMAWAN SAPTADI
Disertasi
Sebagai salah satu syarat untuk memperoleh Gelar Doktor pada
Program Studi Pemuliaan dan Bioteknologi Tanaman
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
x
Penguji pada Ujian Tertutup: Dr. Ir. Hajrial Aswidinnoor, M.Sc.
Dr. Ir. Ni Made Armini Wiendi, MSc.
Penguji pada Ujian Terbuka : Dr. Ir. Maya Melati, M.Sc., MS
xi
Judu l Disertasi : Pemanfaatan Marka Molekuler dalam Pemuliaan Tanaman untuk Mendukung Penyediaan Kultivar Unggul Baru Jarak Pagar (Jatropha curcas L.)
Nama Mahasiswa : Darmawan Saptadi No. Pokok : A263070021
Program Studi : Pemuliaan dan Bioteknologi Tanaman
Disetujui
Komisi Pembimbing
Ketua
Prof. Dr. Ir. Sudarsono, M.Sc.
Dr. Ir. Asep Setiawan, MS
Anggota I Anggota II
Dr. Ir. Bambang Heliyanto, M.Sc.
Mengetahui
Ketua Program Studi Dekan Sekolah Pascasarjana
Pemuliaan dan Bioteknologi Tanaman
xii
PRAKATA
Alhamdulillahirabbil’alamin. Segala puji penulis panjatkan kepada Allah SWT karena dengan segala nikmat dan rahmatNya penulis berhasil menyelesaikan penelitian dan disertasi yang berjudul: Pemanfaatan Marka Molekuler dalam Pemuliaan Tanaman untuk Mendukung Penyediaan Kultivar Unggul Baru Jarak Pagar (Jatropha curcas L.). Hasil penelitian yang dilaksanakan mulai bulan Mei 2008 hingga Juni 2011 ini diharapkan dapat menjadi tambahan informasi bagi penelitian pemuliaan jarak pagar berikutnya.
Atas selesainya penelitian dan disertasi ini penulis menyampaikan penghargaan dan terima kasih kepada Prof. Dr. Ir. Sudarsono, M.Sc. selaku ketua komisi pembimbing, Dr. Ir. Asep Setiawan, MS serta Dr. Ir. Bambang Heliyanto, M.Sc. selaku pembimbing pendamping yang telah banyak memberi bimbingan, arahan dan saran sejak persiapan hingga selesainya disertasi ini. Kepada Ditjen Dikti disampaikan terima kasih atas bantaun biaya studi melalui program BPPS. Terima kasih juga penulis sampaikan kepada Badan Litbang Pertanian, Kementrian Pertanian yang telah memberi dukungan biaya penelitian melalui program KKP3T. Kepada pihak Universitas Brawija disampaikan terima kasih atas ijin dan dukungannya dalam studi yang penulis tempuh.
Terima kasih dan penghargaan penulis sampaikan kepada rekan-rekan Program Studi Pemuliaan dan Bioteknologi Tanaman, rekan-rekan Laboratorium Biologi Molekuler Tanaman beserta teknisi atas segala persahabatan, kerja sama bantuan moril maupun mareriil serta diskusi-diskusinya. Terima kasih tak terhingga kepada istri, anak-anak, orang tua serta saudara-saudara semua atas bantuan, do’a dan pengorbanannya selama penulis menempuh studi. Kepada Bapak Nur Asbani S.P., M.Si penulis sampaikan terima kasih atas ijin untuk menggunakan materi penelitiannya. Kepada sejawat di Universitas Brawijaya disampaikan terima kasih atas dukungannya. Semua fihak yang tidak dapat penulis sebutkan satu persatu, terima kasih atas dukungan dan bantuannya. Semoga semua kebaikan yang telah diberikan kepada penulis dicatat sebagai amal ibadah dan mendapat balasan yang setimpal dari Allah SWT. Akhirnya, permohonan maaf yang sebesar-besarnya penulis sampaikan kepada semua fihak apabila ada kesalahan penulis selama berinteraksi dalam penelitian maupun dalam penulisan disertasi ini.
xiii
RIWAYAT HIDUP
xiv
DAFTAR ISI
Halaman
DAFTAR TABEL ………... xiii
DAFTAR GAMBAR ……….. xv
DAFTAR LAMPIRAN ………... xvii
BAB I PENDAHULUAN Latar Belakang ……….……… 1
Tujuan Penelitian ……….……… 3
Strategi Penelitian ……….…...………… 4
BAB II TINJAUAN PUSTAKA Biologi, Botani, Asal Usul dan Penyebaran Jarak Pagar ………. 7
Pemuliaan Jarak Pagar ……….. 10
Marka Molekuler untuk Evaluasi Genetik ……… 12
Keragaman Jarak Pagar Berbasis Marka Morfologi dan Biokimia ………. 16
Keragaman Jarak Pagar Berbasis Marka Molekuler … 20 BAB III PENGEMBANGAN MARKA SIMPLE SEQUENCE REPEAT (SSR) UNTUK Jatropha spp. Abstrak ………. 25
Abstract ……….... 26
Pendahuluan …..……… 27
Bahan dan Metode ……… 28
Hasil ……….. 31
Pembahasan ……….. 37
Kesimpulan ……….. 41
Daftar Pustaka ……….. 41
BAB IV EVALUASI KERAGAMAN GENETIK JARAK PAGAR (Jatropha curcas L.) BERDASARKAN MARKA MOLEKULER Abstrak ………. 45
Abstract ……….... 46
Pendahuluan …..……… 47
Bahan dan Metode ……… 49
Hasil ……….. 53
Pembahasan ……….. 62
Kesimpulan ……….. 67
Daftar Pustaka ……….. 67
xv
JARAK PAGAR (Jatropha curcas L.) DALAM
GENUS: POLIMORFISME ALEL DAN DERAJAT HUBUNGAN
Abstrak ………. 71
Abstract ……….... 72
Pendahuluan …..……… 73
Bahan dan Metode ……… 75
Hasil ……….. 77
Pembahasan ……….. 81
Kesimpulan ……….. 84
Daftar Pustaka ……….. 84
BAB VI PENGEMBANGAN MARKA DAN KARAKTERISASI MOLEKULER HASIL PERSILANGAN INTERSPESIFIK J. curcas x J. integerrima Abstrak ………. 89
Abstract ……….... 90
Pendahuluan …..……… 91
Bahan dan Metode ……… 93
Hasil ……….. 96
Pembahasan ……….. 101
Kesimpulan ……….. 104
Daftar Pustaka ……….. 105
BAB VII PEMBAHASAN UMUM ……….……. 109
BAB VIII KESIMPULAN DAN SARAN Kesimpulan ………... 115
Saran ………. 115
DAFTAR PUSTAKA ……….……….. 117
xvi
DAFTAR TABEL
Halaman
1. Aksesi jarak pagar dan kerabatnya yang digunakan untuk evaluasi primer SSR yang dikembangkan dalam penelitian ………...
32 2. Daftar 28 pasang primer spesifik untuk mengamplifikasi SSR yang
didesain menggunakan aksesi DNA jarak pagar dari GenBank
DNA database ……… 33
3. Konstitusi genetik berdasarkan hasil skoring alel marker SSR yang diamplifikasi dari templat lima aksesi J. curcas dan dua aksesi J. multifida dengan menggunakan 19 pasangan primer SSR yang
didesain dalam penelitian ………... 36
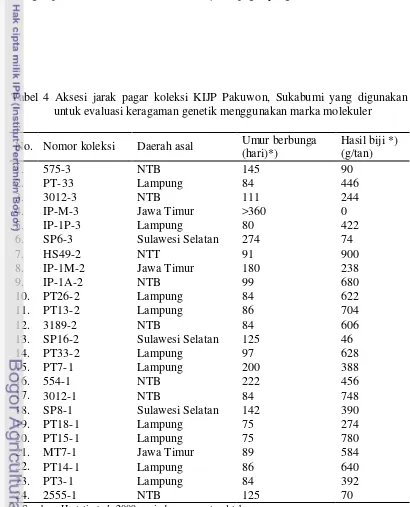
4. Aksesi jarak pagar koleksi KIJP Pakuwon, Sukabumi yang digunakan untuk evaluasi keragaman genetik menggunakan marka
molekuler ………... 53
5. Daftar primer spesifik untuk mengamplifikasi SSR yang didesain menggunakan aksesi DNA jarak pagar dari basis data GenBank
DNA ……….. 54
6. Daftar 10 pasang primer SSR yang dikembangkan dari M. esculenta yang digunakan untuk mengakses keragaman pada jarak
pagar ………. 55
7. Primer RAPD dan ISSR yang digunakan untuk evaluasi plasma
nutfah jarak pagar Indonesia ………. 57 8. Primer SCAR yang digunakan untuk evaluasi plasma nutfah jarak
pagar Indonesia ………. 58
9. Daftar primer RAPD dan ISSR yan teramplifikasi, jumlah produk
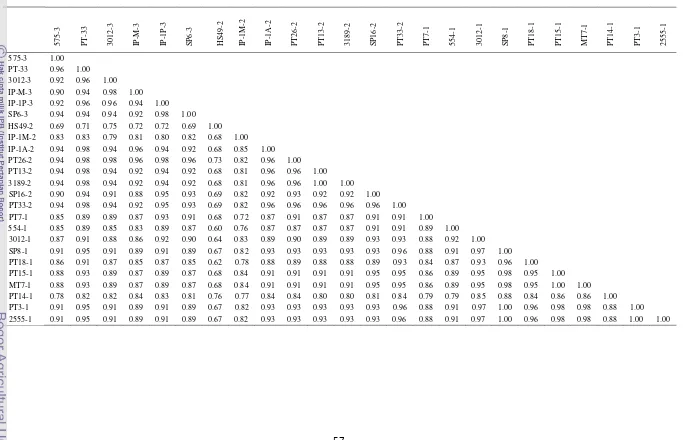
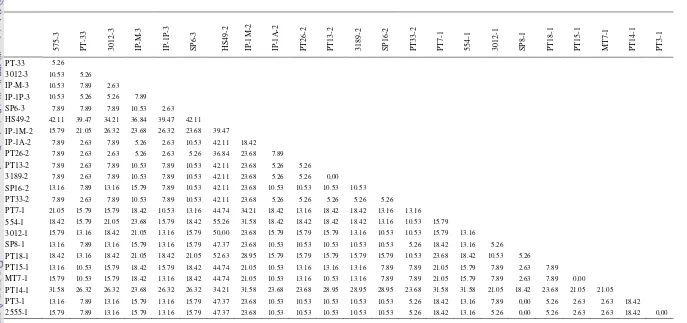
amplifikasi, pita polimorf dan persentase polimorfisme ………….. 58 10. Koefisien kesamaan genetik menurut Dice pada 24 aksesi jarak
pagar berdasarkan marka RAPD dan ISSR ………. 60 11. Persentase polimorfisme 24 aksesi jarak pagar berdasarkan marka
RAPD dan ISSR …………...……… 61
xvii
13. Daftar 28 pasang primer spesifik untuk mengamplifikasi SSR yang didesain menggunakan aksesi DNA jarak pagar dari GenBank
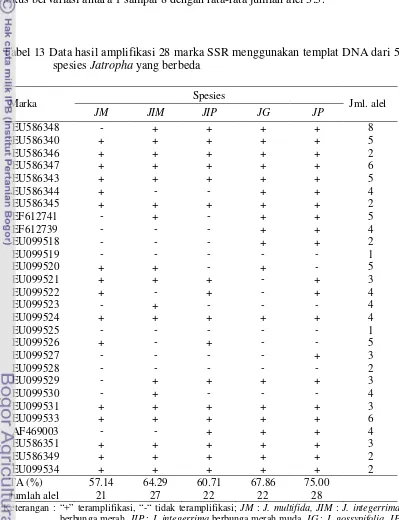
DNA Database untuk amplifikasi lintas spesies Jatropha ………… 78 14. Data hasil amplifikasi 28 marka SSR menggunakan templat DNA
dari 5 spesies Jatropha yang berbeda ……….. 80 15. Perbedaan karakter morfologi yang teramati pada tanaman P1 (J.
curcas), P2 (J. integerrima) dan F1 ……… 96 16. Hasil skoring terhadap analisis molekuler menggunakan marka
SSR, RAPD dan ISSR pada individu-individu tetua dan F1 hasil
persilangan keduanya ………. 98
17. Koefisien kesamaan genetik antara 8 individu F1 (1-8) dan 2 tanaman tetuanya (P1 = J. curcas; P2 = J. integerrima) berdasarkan hasil analisis molekuler dengan 9 marka SSR, 5 marka RAPD dan 3 marka ISSR ………..
xviii
DAFTAR GAMBAR
Halaman
1. Bagan alir kegiatan penelitian ...………... 5
2. Bagian-bagian penting jarak pagar :a-cabang generative, b-batang, c-arsitektur vena daun, d-bunga betina, f-irisan melintang buah
muda, g-buah, h-irisan membujur buah, i-biji (Heller, 1996) …….. 8
3. High molecular weight DNA hasil ekstraksi dengan protokol (A) CTAB standar dan (B) protokol modifikasi. Kolom (1-4) - aksesi
J. curcas dan kolom (5-6) – aksesi J. multifida ……… 32 4. Elektroferogram hasil evaluasi produk amplifikasi PCR dengan
menggunakan elektroforesis gel agarosa. Pewarnaan gel agarosa dilakukan dengan ethidium bromide dan foto gel diambil di bawah pencahayaan sinar UV. Hasil amplifikasi PCR dengan menggunakan templat genom jarak pagar dan pasangan primer (1) EF612741, (2) EU586345, (3) EF612739, (4) EU586340, (5) EU586343, (6) EU586344, (7) EU586346, (8) EU586347, dan (9)
EU586348 ……… 34
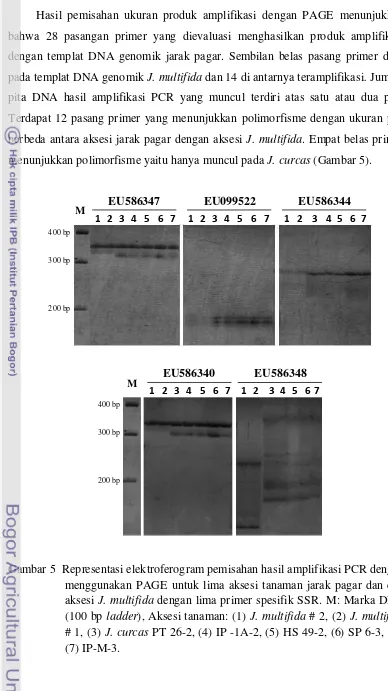
5. Representasi elektroferogram pemisahan hasil amplifikasi PCR dengan menggunakan poliakrilamid gel elektroforesis (PAGE) untuk lima aksesi tanaman jarak pagar dan dua aksesi J. multifida dengan menggunakan lima primer spesifik SSR. M: Marka DNA (100 bp ladder), Aksesi tanaman: (1) J. multifida # 2, (2) J. multifida # 1, (3) J. curcas PT 26-2, (4) IP -1A-2, (5) HS 49-2, (6)
SP 6-3, dan (7) IP-M-3 ……….…… 35
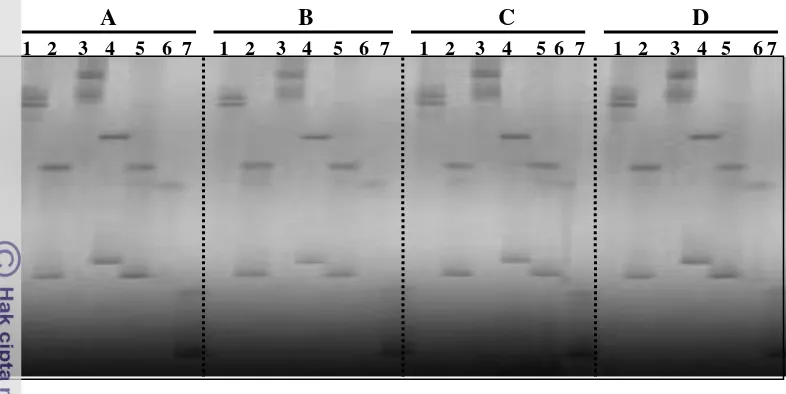
6. Representasi elektroferogram pemisahan hasil amplifikasi PCR dengan menggunakan poliakrilamid gel elektroforesis (PAGE) untuk empat individu tanaman jarak pagar F1 (A-D), dengan menggunakan pasangan primer (1) EU586345, (2) EU586343, (3) EF612741, (4) EU586347, (5) EF612739, (6) EU586346, dan (7)
EU586340 ………...……….. 40
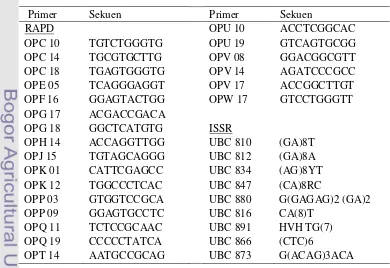
7. Pola pita hasil PAGE pada 24 nomor koleksi (1 – 24) dengan
primer EU586340 (►) dan EU586347 (→) …….……… 56 8. Elektroferogram hasil amplifikasi DNA pada 24 nomor koleksi
jarak pagar (1-24) dengan primer OPV 17, M = marka DNA 1
xix
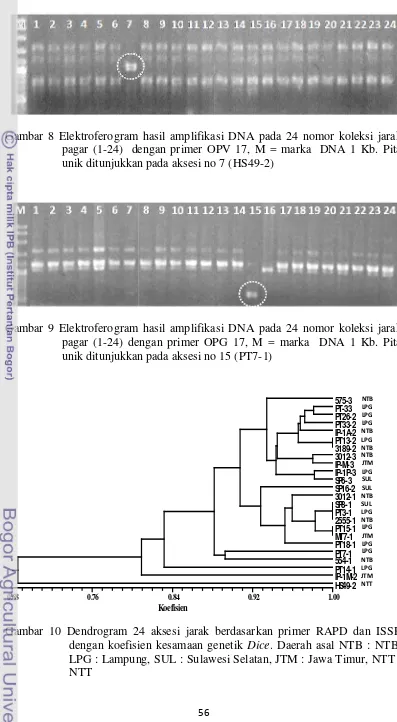
9. Elektroferogram hasil amplifikasi DNA pada 24 nomor koleksi jarak pagar (1-24) dengan primer OPG 17, M = marka DNA 1
Kb. Pita unik ditunjukkan pada aksesi no 15 (PT7-1)………... 59 10. Dendrogram 24 aksesi jarak berdasarkan primer RAPD dan ISSR
dengan koefisien kesamaan genetik Dice. Daerah asal B : NTB, L :
Lampung, S : Sulawesi Selatan, J : Jawa Timur, T : NTT ...……… 59
11. Elektroferogram hasil amplifikasi lintas spesies 5 marka SSR (EU586348, EU586347, EU586346, EU586344, EU586340) pada 5 spesies Jatropha yang berbeda (1 = J. curcas, 2 = J. integerrima bunga merah muda, 3 = J. integerrima bunga merah, 4 = J. gossypifolia, 5 = J. multifida, 6 = J. podagrica) M = marka DNA
100 bp……… 79
12. Dendrogram dibuat berdasarkan data analisis amplifikasi marka SSR lintas spesies pada 5 spesies Jatropha (JC = J. curcas, JIP = J. integerrima bunga merah muda, JIM = J. integerrima bunga merah, JM = J. multifida, JP = J. podagrica dan JG = J.
gossypifolia) ….………..………… 80
13. Hasil pengamatan secara morfologis karakter daun (A), bunga (B), buah (C), biji (D) pada tanaman tetua dan F1; variasi bentuk biji (E) dan percabangan (F) pada tanaman F1; Variasi pigmentasi pada tangkai karangan bunga tanaman F1: berpigmen (G) dan
tidak berpigmen (H)
…….………..
97 14. Elektroferogram hasil amplifikasi DNA 8 individu F1 (1-8) hasil
persilangan J. curcas (P1) x J. integerrima (P2) dengan 4 marka
SSR. M = marka DNA 100 bp ..………. 99
15. Dendrogram hasil analisis molekuler menggunakan marka SSR, RAPD dan ISSR pada individu-individu tetua (P1 = J. curcas; P2
= J. integerrima) dan F1 hasil persilangan keduanya (1-8) ……. 101 16. Skema perbaikan genetik jarak pagar melalui persilangan
xx
DAFTAR LAMPIRAN
Halaman
1. Gambar peta penyebaran jarak pagar (J. curcas) dari pusat
penyebaran di Amerika Tengah (Heller, 1996) sampai ke Asia ... 131 2. Sistematika genus Jatropha .……… 132
3. Dendrogram 24 aksesi jarak berdasarkan analisis molekuler menggunakan marka RAPD dan ISSR dengan koefisien kesamaan
genetik Dice. Karakter daya hasil (g/tan) diplot pada aksesi ……... 133 4. Dendrogram 24 aksesi jarak berdasarkan analisis molekuler
menggunakan marka RAPD dan ISSR dengan koefisien kesamaan
genetik Dice. Karakter umur berbunga (hari) diplot pada aksesi …. 133 5. Gambar morfologi tanaman, daun, bunga dan buah beberapa
spesies kerabat J. curcas ……….. 134
6. Gambar morfologi daun, bunga buah dan pigmentasi pada
1
BAB I
PENDAHULUAN
Latar Belakang
Kebutuhan energi dunia hingga saat ini sebagian besar dipenuhi dengan mengandalkan bahan bakar fosil (minyak bumi, gas dan batubara). Telah disadari bahwa bahan bakar fosil adalah sumber energi yang tidak terbarukan dan saat ini cadangannya di dalam bumi semakin menipis. Bahan bakar tak terbarukan ini dari hari ke hari harganya semakin meningkat dan perannya dalam kerusakan lingkungan (baik dari proses eksploitasi maupun sisa penggunaannya) semakin terasa. Saat ini dunia tengah dihadapkan pada krisis energi jika alternatif pengganti bahan bakar fosil tidak ditemukan.
Antisipasi terhadap krisis energi harus segera dilakukan. Cara yang dapat ditempuh adalah dengan melakukan penghematan penggunaan bahan bakar fosil dan mencari alternatif baru sumber bahan bakar terbarukan yang lebih ramah lingkungan. Beberapa sumber minyak yang berasal dari bahan nabati telah dieksplorasi seperti kelapa sawit, kelapa, sorgum, jagung dan ubi kayu yang kemudian dikenal sebagai bahan bakar nabati (BBN). Hampir semua komoditi yang disebutkan tersebut adalah komoditi pangan, sehingga pemanfatannya untuk sumber energi akan mengganggu keamanan ketersediaan pangan. Diperlukan produksi yang cukup, bahkan berlebih untuk dapat menggunakan komoditi-komoditi tersebut sebagai sumber bahan bakar agar tidak bersaing dengan kebutuhan pangan (Djazuli dan Prastowo, 2008).
2
Selain hal-hal tersebut, keunggulan lain dari jarak pagar adalah bahwa hampir semua produk sampingnya seperti bungkil, kulit biji, gliserol dll. masih dapat dimanfaatkan (Anonim, 2006).
Pemerintah Indonesia telah menanggapi perkembangan kelangkaan sumber energi dengan keluarnya Perpres No 5 tahun 2006 tentang ‘Kebijakan Energi Nasional’ yang salah satu poinnya menyebutkan bahwa proyeksi penggunaan energi nasional pada tahun 2025 sebesar 5% berasal dari bio-fuel. Jarak pagar merupakan salah satu komoditi yang ditetapkan sebagai sumber bio-fuel. Kebijakan lain yang dikeluarkan oleh pemerintah adalah Inpres No 1 tahun 2006 tentang ‘Penyediaan dan Pemanfaatan Bahan Bakar Nabati (bio-fuel) sebagai Bahan Bakar Lain’. Di antara poin yang tertuang dalam Inpres ini menyebutkan tugas Menteri Pertanian di antaranya adalah: memfasilitasi penyediaan benih dan bibit tanaman bahan baku bahan bakar nabati. Selanjutnya Badan Penelitian dan Pengembangan Pertanian telah menyusun langkah-langkah strategis yang salah satu di antaranya adalah mengembangkan bibit ungul tanaman jarak pagar.
Pusat Penelitian dan Pengembangan Tanaman Perkebunan telah mengawali langkah untuk dapat menyediakan bahan tanam unggul jarak pagar dengan melakukan eksplorasi terhadap plasma nutfah jarak pagar ke hampir seluruh wilayah Indonesia. Seleksi tahap pertama terhadap provenan-provenan tersebut mendapatkan IP-1 (Improved Population -1) yang memiliki produksi 1 ton/ha/tahun dengan kadar minyak 36.06 – 37.64%. Siklus ke dua mendapatkan IP-2 dengan peningkatan pada hasilnya yaitu menjadi 2 ton/ha/tahun (Hasnam et al., 2007). Hasil ini menurut kajian ekonomi yang dilakukan Kemala (2006) belum menguntungkan karena dengan teknologi budidaya sederhana, keuntungan baru akan diperoleh jika hasil jarak pagar mencapai lebih dari 2 ton/ha/tahun sejak tahun pertama dengan patokan harga Rp. 700 sampai dengan Rp. 1000 per kilogram biji kering. Bahkan, Martono (2009) mengemukakan bahwa jika dibudidayakan dengan metode standar, harga jual biji Rp. 1.000 per kg dan asumsi produksi 5 ton/ha/th maka petani masih akan merugi.
3
massa tetapi harus diiringi dengan kegiatan persilangan memanfaatkan keragaman genetik yang dimiliki. Beberapa pengamatan terhadap keragaman genetik plasma nutfah Indonesia berdasarkan marka morfologis menunjukkan adanya keragaman yang cukup tinggi (Hasnam, 2006a; Sudarmo et al., 2007; Hartati et al., 2009). Pemilihan tetua untuk persilangan jarak pagar dengan tujuan meningkatkan daya hasil mengandalkan pengamatan pada karakter morfologis cukup riskan karena penampilan fenotipik tidak sepenuhnya menggambarkan genotip. Kesalahan penentuan tetua akan sangat merugikan baik dari segi biaya, tenaga, waktu maupun kesempatan. Jarak pagar adalah tanaman tahunan sehingga perencanaan program pemuliaan harus dilakukan dengan lebih matang khususnya yang berkaitan dengan informasi keragaman genetik.
Teknologi marka molekuler telah berkembang pesat dan telah dimanfaatkan untuk mendukung program pemuliaan pada banyak spesies tanaman terutama dalam identifikasi genetik. Marka molekuler memiliki presisi yang lebih tinggi dibandingkan dengan marka morfologi dan dapat diaplikasikan pada fase sangat awal dari pertumbuhan tanaman. Seleksi tetua maupun keturunan dengan demikian dapat dilakukan dengan lebih akurat dan lebih awal menggunakan marka molekuler. Marka molekuler belum banyak dieksplorasi pada komoditi jarak pagar khususnya di Indonesia. Pengembangan marka molekuler untuk jarak pagar diperlukan untuk dapat mengonfirmasi keragaman morfologis yang telah terdokumentasi sebelumnya maupun untuk eksplorasi keragaman pada materi genetik yang lebih luas bahkan pada kerabat jarak pagar yang lain (Jatropha spp.).
Tujuan Penelitian
Secara umum penelitian ini bertujuan untuk mendapatkan informasi berbasis kajian molekuler untuk mendukung pelaksanaan program pemuliaan dalam rangka mendapatkan kultivar jarak pagar unggul yang memiliki daya hasil dan kadar minyak tinggi. Informasi yang dimaksud di antaranya ialah:
4
• Keragaman genetik plasma nutfah jarak pagar Indonesia berdasarkan marka molekuler yang dikembangkan.
Strategi Penelitian
Marka molekuler akan dikembangkan berdasarkan pertimbangan ketersediaan sarana pendukung penelitian, ketersediaan informasi awal yang memadai, ketersediaan dana serta manfaat yang akan diperoleh. Marka SSR (Simple Sequence Repeat) dipilih karena keunggulannya untuk evaluasi keragaman genetik. Pengembangan marka SSR dilakukan dengan memanfaatkan basis data sekuen DNA yang tersedia pada bank gen sehingga biaya tinggi dalam pembuatan pustaka genom, pengurutan DNA dan penapisan SSR dapat dihindari. Berdasarkan informasi awal yang telah diperoleh maka marka RAPD (Random Amplified Polymorphic DNA), ISSR (Inter Simple Sequence Repeat) dan SCAR (Sequence Characterized Amplified Region) dipilih karena kemudahan dan kesederhanaan teknik serta ketersediaan informasi sekuen primer yang telah ada.
Primer-primer yang telah dibuat kemudian diuji coba dan dimanfaatkan untuk mengevaluasi keragaman genetik plasma nutfah jarak pagar. Identitas genetik yang diperoleh berdasarkan evaluasi dengan marka molekuler tersebut apabila mencukupi maka selanjutnya dapat digunakan untuk pemilihan tetua persilangan, evaluasi heterosis pada keturunan serta pembuatan peta pautan genetik pada jarak pagar. Adapun apabila informasi yang didapatkan dari marka tersebut tidak memadai untuk melakukan hal tersebut di atas maka marka dapat digunakan untuk mengakses keragaman genetik dari spesies kerabat jarak pagar. Marka juga dapat dimanfaatkan untuk mengevaluasi hasil persilangan interspesifik pada Jatropha spp.
Secara skematis keseluruhan kegiatan penelitian digambarkan pada Gambar 1. Adapun tahapan kegiatan untuk mencapai tujuan penelitian dilakukan sebagai berikut:
I. Pengembangan marka simple sequence repeat (SSR) untuk Jatropha spp. Kegiatan 1 : Optimasi ekstraksi DNA jarak pagar
5
II. Evaluasi keragaman genetik jarak pagar (Jatropha curcas L.) berdasarkan marka molekuler.
Kegiatan 1 : Evaluasi keragaman genetik dengan marka SSR
Kegiatan 2 : Evaluasi keragaman genetik dengan marka RAPD, ISSR dan SCAR
III. Amplifikasi lintas spesies marka SSR jarak pagar (Jatropha curcas L.) dalam genus: polimorfisme alel dan derajat hubungan
IV. Pengembangan marka dan karakterisasi molekuler hasil persilangan interspesifik J. curcas x J. integerrima
Kegiatan 1 : Pengamatan karakter morfologis pada F1 dan tetua
Kegiatan 2 : Evaluasi genetik berdasar marka molekuler pada F1 dan tetua
6
BAB II
TINJAUAN PUSTAKA
Biologi, Botani, Asal usul dan Penyebaran Jatropha spp.
Jarak pagar (Jatropha curcas L.) termasuk dalam famili Euphorbiaceae. Tanaman jarak pagar adalah pohon kecil atau semak besar yang dapat mencapai tinggi hingga 5 meter. Pertumbuhannya sangat dipengaruhi oleh kondisi iklim lingkungannya dan dormansi dapat terjadi karena fluktuasi curah hujan serta temperatur atau cahaya. Batangnya mengandung getah dan pada tanaman yang berasal dari biji (seedling) perakarannya satu di tengah dan empat di tepi (peripheral). Daunnya palmate berlobus dangkal sebanyak 5 – 7, memiliki panjang dan lebar bervariasi dari 6 sampai 15 cm dan tersusun secara alternate (Heller, 1996) (Gambar 2).
J. curcas adalah tanaman tahunan berumah satu (monoecious), diploid (Paramathma et al., 2005). Bunga jarak pagar juga bersifat protandrous di mana seringkali ditemukan dalam satu tanaman adanya bunga jantan dan bunga betina dengan rasio 29 : 1 dan kadang-kadang bunga hermaprodit. Bunga jantan dan bunga betina membuka secara sinkron sehingga dengan sistem pembungaan seperti itu memungkinkan penyerbukan berlangsung secara xenogamy maupun secara geitonogamy (Raju dan Ezradanam (2002). Karyotype J. curcas tersusun atas 22 kromosom metasentrik dan submetasentrik yang berukuran relatif kecil yang ukurannya berkisar antara 1.71 sampai dengan 1.24 μm (Carvalho et al., 2008; Soontornchainaksaeng dan Jenjittikul, 2003). Ukuran genom J. curcas relatif kecil yaitu sekitar 286 Mb (Sato et al., 2011).
7
kemungkinan bahwa Meksiko dan Amerika Tengah adalah sumber keragaman jarak pagar ditunjang kenyataan bahwa di Asia dan di Afrika tidak ditemui jarak pagar kecuali sebagai tanaman budidaya.
8
Dari kepulauan Karibia, jarak pagar kemungkinan disebarkan oleh penjelajah Portugis melalui Pulau Cape Verde dan Guinea Bissau di Afrika ke negara-negara lain di Afrika dan Asia (Lampiran 1). Burkill (1966 dalam Heller, 1996) berasumsi bahwa Portugis membawa jarak pagar ke Asia tetapi tidak sampai mencapai Malaka hingga datangnya penjajah Belanda sehingga orang Malaysia menyebut jarak pagar dengan suatu sebutan yang berarti jarak Belanda. Tanaman jarak telah tersebar di hampir seluruh kepulauan di Indonesia. Penyebaran spesies ini di Indonesia dapat diketahui dari hasil eksplorasi yang dilakukan oleh tim dari Pusat Penelitian dan Pengembangan Tanaman Perkebunan, Badan Litbang Pertanian, Kementrian Pertanian RI. Eksplorasi berhasil mendapatkan provenan-provenan hampir dari seluruh wilayah Indonesia yaitu dari: Sumatra Barat, Lampung, Banten, Jawa Barat, Jawa Timur, Nusa Tenggara Barat, Nusa Tenggara Timur dan Sulawesi Selatan (Mahmud, 2006).
Jatropha L. adalah genus yang tersebar luas dan memiliki 175 spesies (Airy Shaw, 1972). Pada umumnya terdapat di Amerika Tropika dan Afrika. Hanya beberapa spesies yang asli Asia Barat Daya dan Asia serta satu spesies di Madagaskar. Tidak ada spesies asli Asia tenggara, Australia atau Oceania. Sebagaimana di Thailand, di Indonesia ada lima spesies Jatropha yang dijumpai yaitu: J. curcas L. dan J.gossypifolia L. yang digunakan sebagai tanaman obat serta J. integerrima Jacq., J. multifida L. dan J. podagrica Hook. yang digunakan sebagai tanaman hias (Chayamarit et al., 2001, Hasnam, 2006).
Sebagian genus ini mempunyai jumlah khromosom 2n=44; J. curcas sendiri tebagi dalam dua kelompok 2n=22 dan 2n=24. Studi karyologi dari
9
ketiga spesies ini berada dalam kelompok yang berbeda pada subgenus Jatropha dan Subgenus Curcas. Sistematika genus Jatropha dapat dilihat pada Lampiran 2.
Pemuliaan Jarak Pagar
Kunci sukses program perbaikan genetik adalah pada ketersediaan keragaman genetik dari karakter yang dikehendaki (Heller, 1996). Eksplorasi global terhadap sumber genetik, introduksi, karakterisasi dan evaluasi akan menyediakan pijakan yang kuat bagi pengembangan varietas unggul dengan berbagai metode perbaikan. Pekerjaan koleksi, karaketrisasi dan evaluasi plasma nutfah untuk pertumbuhan, morfologi, karakteristik biji, sifat daya hasil jarak pagar masih dalam tahap paling awal (Rao et al., 2008). Pengujian provenan yang sistematik dengan skala yang diperlukan belum dilakukan dan materi genetik dari pusat keragaman belum dieksplorasi sepenuhnya. Sementara latar belakang genetik jarak pagar di Asia dan di Afrika belum begitu jelas (Divakara et al., 2009).
Jarak pagar adalah tanaman introduksi pada banyak negara di Asia, Afrika serta Amerika Latin dan belum ada usaha sistematik untuk perbaikan pada tanaman ini. Varietas unggul dengan sifat-sifat yang diinginkan untuk tempat tumbuh yang spesifik belum tersedia (Jongschaap et al., 2007). Tujuan perbaikan genetik jarak pagar sebaiknya diarahkan untuk: jumlah bunga betina lebih banyak, hasil biji dan kandungan minyak tinggi, genjah, tahan terhadap hama dan penyakit, toleran terhadap kekeringan, tanaman pendek dan percabangan banyak (Divakara et al., 2009). Jarak pagar berada pada satu famili dengan jarak kepyar (Ricinus communis L.) sehingga menurut Sujatha et al. (2008) perbaikan genetik dan domestikasi jarak pagar sebaiknya mengikuti alur pada jarak kepyar. Jarak pagar dapat diperbaiki melalui pendugaan keragaman kerabat liar dan seleksi genotip superior, melalui pemuliaan mutasi, transfer gen asing melalui persilangan interspesies dan penemuan bioteknologi yang membawa perubahan ke arah sifat yang diinginkan (Divakara et al., 2009).
10
esterifikasi. Tujuan pemuliaan paling penting saat ini untuk jarak pagar adalah peningkatan hasil minyak. Komponen yang mempunyai andil dalam hasil minyak per hektar adalah: jumlah bunga betina per tandan dan jumlah kapsul per tanaman, berat 1000 biji, kandungan minyak biji dan jumlah tanaman per hektar. Karena jumlah biji maksimum dalam kapsul terbatas dan faktor agronomi pada suatu kerapatan tanam juga terbatas maka untuk meningkatkan hasil dapat difokuskan pada peningkatan komponen hasil (Heller, 1996).
Aktivitas utama dalam perbaikan genetik adalah persilangan dan seleksi pada keragaman yang dimiliki. Jarak pagar adalah tanaman menyerbuk terbuka sehingga metode perbaikan genetik yang dapat diaplikasikan untuk mengeksploitasi keragaman genetik antara lain : (i) seleksi massa; (ii) seleksi berulang; (iii) pemuliaan mutasi; (iv) pemuliaan heterosis dan (v) persilangan interspesies (Divakara et al., 2009). Seleksi massa adalah metode pemuliaan paling sederhana yang dapat digunakan, di mana tanaman superior terpilih dikompositkan. Jika populasi terjaga tetap besar maka peningkatan akan dapat dicapai dengan memanfaatkan variasi genetik aditif. Metode seleksi berulang telah banyak dipakai untuk pemuliaan tanaman pohon. Kegiatannya meliputi seleksi yang dilakukan bersamaan baik yang menggunakan atau yang tanpa uji keturunan. Modifikasi dapat dilakukan pada metode ini. Selain itu kultivar hibrida juga dapat diusahakan dengan memanfaatkan efek heterosis (Heller, 1996). Persilangan intersepesies pada Jatropha mempunyai andil yang besar untuk perbaikan genetik dalam hal transfer beberapa sifat berguna seperti kandungan minyak tinggi, jumlah biji maksimum, bunga betina yang banyak dan cabang yang kuat (Parthiban et al., 2009).
11
Sejak tahun 1996 Puslitbang Perkebunan telah melakukan kegiatan seleksi terhadap koleksi plasma nutfah yang dimiliki. Pada siklus pertama telah diidentifikasi populasi IP-1A, IP-1M dan IP-1P yang memiliki rerata daya hasil masing-masing 88 ± 21, 52 ± 14.7 dan 114 ± 20 kapsul per tanaman (Hasnam, 2006a). Siklus seleksi kedua (dengan kriteria seleksi jumlah kapsul pertanaman lebih dari 400 pada tahun pertama) didapatkan IP-2 dengan target produksi 2 ton pada tahun pertama, 3 – 3.5 ton pada tahun ke dua, 4 – 4.5 ton pada tahun ke tiga dan 6 – 7 ton pada tahun ke empat (Hasnam et al., 2007). IP3-P dan IP3-A yang merupakan hasil seleksi dari IP2-P dan IP2-A telah dilepas pada tahun 2009. IP3-P mempunyai potensi produksi 2.3 – 2.6 ton/ha/tahun untuk tahun pertama dengan kadar minyak 36% dan bisa mencapai 8 s.d. 9 ton/ha pada tahun ke empat. IP3-A memiliki potensi produksi 2 – 2.5 ton/ha/tahun untuk tahun pertama dengan kadar minyak 35% dan bisa mencapai 8 s.d. 8.5 ton/ha pada tahun ke empat (Puslitbangbun, 2009).
Menurut kajian keekonomian yang dilakukan oleh Kemala (2006), budidaya jarak pagar dengan teknologi rendah (jarak tanam tidak teratur, persentase tumbuh ± 65 persen, bibit sapuan, pemakaian pupuk dan pestisida sedikit) baru akan menguntungkan jika pertanaman mampu menghasilkan biji kering sebanyak ≥ 2 ton/ha/tahun berdasarkan harga jual biji per kilogram berkisar antara Rp. 700 sampai dengan Rp. 1000. Sementara jika dibudidayakan dengan metode standar (Mahmud et al., 2006), kajian keekonomian yang dilakukan oleh Martono (2009) mendapatkan bahwa dengan harga jual biji Rp. 1.000 per kg dan asumsi produksi 5 ton/ha/th, petani masih akan merugi. Hasnam, (2006) membuat perhitungan sederhana yaitu dengan asumsi populasi 2.500 tanaman per hektar dengan faktor koreksi 20% akan menghasilkan biji kering > 2 ton/ha/tahun jika tiap tanaman dapat menghasilkan > 400 kapsul per tahun.
Marka Molekuler untuk Evaluasi Genetik
12
pengujian DNA (marka molekuler) (Semagn et al., 2006). Marka morfologi dikarakterisasi visual secara fenotipik seperti warna bunga, bentuk biji, tipe tumbuh atau pigmentasi. Marka isozym adalah marka yang dapat membedakan enzim yang dideteksi melalui elektroforesis dan merupakan penanda spesifik. Keterbatasan dari marka morfologi dan biokimia adalah dalam jumlah dan keterlibatan pengaruh faktor lingkungan atau fase perkembangan tanaman. Marka molekuler mempunyai keunggulan karena jumlahnya tidak terbatas dan tidak dipengaruhi oleh faktor lingkungan maupun fase perkembangan tanaman (Tanksley dan McCouch, 1997).
Teknik dasar marka molekuler dapat diklasifikasikan menjadi 2 katagori yaitu: (i) teknik yang tidak berdasar pada PCR (non-PCR-based techniques) atau teknik berdasar hibridisasi seperti RFLPs (Restriction Fragment Length Polymorphisms) dan (ii) teknik berdasar PCR (PCR-based techniques) seperti RAPD (Random Amplified Polymorphic DNA), AFLP (Amplified Fragment Length Polymorphism), SSRs (Simple Sequence Repeats), SCAR (Sequence Characterized Amplified Region), ISSR (Inter Simple Sequence Repeat) (Agarwal et al., 2008; Semagn et al., 2006).
13
Karena karakteristiknya maka teknik RAPD memungkinkan penapisan dengan cepat polimorfisme pada daerah berbeda dalam genom (Williams et al., 1990) dan merupakan alat yang sangat berguna untuk pemetaan (Reiter et al., 1992), memungkinkan deteksi marka yang terkait dengan gen untuk sifat penting (Mitchelmore et al., 1991; Martin et al., 1991) dan kloning berdasar peta terhadap gen yang besangkutan. Sebagai contoh adalah gen fad3 yaitu gen omega-3 desaturase dari Arabidopsis thaliana dan gen RPS2 untuk ketahanan terhadap penyakit telah berhasil diklon dengan pendekatan ini (Arondel et al., 1992; Bent et al., 1994). Marka RAPD juga telah digunakan secara luas untuk berbagai keperluan di antaranya untuk evaluasi keragaman genetik pada barley (Albayrak dan Gözükirmizi, 1999), jeruk (Karsinah et al., 2002) dan jute (Hossain et al., 2002). Marka ini juga dimanfaatkan untuk determinasi hasil persilangan pada kelapa sawit (Thawaro dan Te-chato, 2009), kol (Liu et al., 2007), kapas (Dongre et al., 2011) dan tebu (Zhang et al., 2008).
SSR (mikrosatelit) adalah sekuen DNA yang pendek dan mempunyai motif berulangan secara tandem dengan 2 sampai 5 unit nukleotida yang tersebar dan meliputi seluruh genom, terutama pada organisme eukariota. Pasangan primer mikrosatelit (forward dan reverse) mengamplifikasi daerah yang diapit (flanking region) yang terjaga (conserved) untuk satu lokus pada kromosom (Akkaya et al., 1992; Powell et al., 1996). Keragaman pada SSR dapat terjadi karena dua hal yaitu karena kejadian pindah silang yang unik selama miosis dan slip pada saat replikasi DNA (Richard et al., 2008).
Genom eukariota mengandung SSR dalam jumlah sangat banyak sehingga memungkinkan untuk digunakan dalam pembentukan saturated map dan gene tagging. Keuntungan SSR secara alami adalah : (i) multipel alel SSR dapat dideteksi pada lokus tungal menggunakan penapisan berbasis PCR (ii) SSR biasanya tersebar pada seluruh genom (iii) sifatnya ko-dominan (iv) jumlah DNA yang dibutuhkan hanya sedikit dan (v) analisis dapat dilakukan secara semi otomatis (Robinson et al., 2004).
14
1996). Metode ini membutuhkan banyak waktu dan mahal. Jika data sekuen DNA telah banyak tersedia maka akan lebih efisien untuk mendesain primer SSR berdasarkan data tersebut dengan bantuan komputer (Robinson et al., 2004). Beberapa marka SSR telah dikembangkan berdasarkan basis data sekuen DNA seperti pada tanaman cabai (Sanwen et al., 2000) dan buncis (Guerra-Sanz, 2004). Marka SSR mempunyai banyak keunggulan dibandingkan dengan marka lain jika dikembangkan berdasarkan informasi sekuen DNA dari spesies yang bersangkutan. Jika data sekuen DNA tidak memadai untuk suatu spesies maka marka SSR dapat dikembangkan dari spesies yang berkerabat dekat (Park et al., 2009). Wen et al. (2010) telah berhasil mengembangkan marka SSR dari M. esculenta untuk evaluasi keragaman genetik pada jarak pagar. Beberapa penelitian telah dilakukan dengan memanfaatkan marka SSR yang dikembangkan dari spesies kerabat seperti pada kedelai (Peakall et al., 1998), rumputan (Zeid et al., 2010) dan bawang merah (Lee et al., 2011). Marka SSR juga telah dimanfaatkan untuk identifikasi dan determinasi hasil persilangan di antaranya pada padi (Ye-yun et al., 2005), jagung (Wang et al., 2002), tebu (Tarpomanova et al., 2009) dan bunga matahari (Antonova et al., 2006).
ISSR adalah segmen DNA yang teramplifikasi di antara dua daerah berulangan (mikrosatelit) yang identik dengan orientasi berlawanan. Teknik ini menggunakan mikrosatelit sebagai primer dan sasarannya adalah banyak lokus pada genom serta mengamplifikasi utamanya ISSR yang berbeda ukuran. Mikrosatelit yang digunakan sebagai primer untuk ISSR dapat berupa dinukleotida, trinukleotida tetranukleotida maupun pentanukleotida (Gupta et al., 1994; Wu et al., 1994). ISSR menunjukkan spesifikasi seperti marka mikrosatelit tetapi tidak memerlukan informasi sekuen untuk mensintesis primer sehingga mempunyai kemudahan seperti menggunakan primer acak (random primer) (Joshi et al., 2000). Teknik ini juga sederhana, cepat dan penggunaan radioaktif tidak terlalu penting serta menunjukkan polimorfisme tinggi (Kojima et al., 1998).
15
terkait dengan sifat tertentu (misalnya pita RAPD muncul pada galur tahan penyakit tetapi tidak muncul pada galur yang tidak tahan). Marka SCAR lebih menguntungkan daripada RAPD karena hanya mendeteksi satu lokus, amplifikasinya kurang sensitif terhadap kondisi reaksi dan potensial untuk dikonversi menjadi marka kodominan (Paran and Michelmore, 1993).
Keragaman Jarak Pagar Berbasis Marka Morfologi dan Biokimia
16
Penelitian-penelitian berikutnya untuk mengevaluasi keragaman genetik jarak pagar masih dilakukan berdasarkan pengamatan morfologis. Makkar et al. (1997) melaporkan keragaman yang tinggi pada jarak pagar asal Afrika Barat dan Timur, Amerika Utara dan Tengah serta Asia. Keragaman tersebut meliputi karakter berat biji (0.49 – 0.86 g/biji), persentase berat kernel (54 – 64 %), kandungan protein kasar (19 – 31 %) dan kandungan minyak (43 – 59 %). Keragaman pada kandungan minyak dicatat oleh Ginwal (2004) pada evaluasi populasi tanaman yang biji-bijinya diperoleh dari tempat yang berbeda-beda di India. Variasi kandungan minyak dari biji antara 33.03 dampai dengan 39.12% dan 47.08 sampai dengan 58.12% kandungan minyak dari kernel. Pada parameter lain diamati perbedaan yang signifikan di antara populasi yaitu tinggi tanaman, diameter batang, jumlah cabang, luas daun dan ketahanan hidup di lapangan. Karakter pertumbuhan juga menunjukkan korelasi satu dengan yang lain. Heritabilitas dalam arti luas nilainya tinggi pada parameter luas daun, tinggi dan diameter batang.
Variasi pada ukuran biji, berat 100 biji dan kandungan minyak dilaporkan dari penelitian Kaushik et al. (2007) terhadap 24 aksesi yang dikoleksi dari beberapa tempat yang memiliki agroklimat yang berbeda di Propinsi Haryana, India. Kandungan minyak bervariasi antara 28 – 38.8%. Koefisien korelasi fenotipik yang tinggi dibandingkan dengan koefisien korelasi genotipik menunjukkan besarnya pengaruh lingkungan. Heritabilitas tinggi pada karakter kandungan minyak menunjukkan adanya aksi gen aditif. Berat biji berkorelasi positif dengan panjang biji dan kandungan minyak.
17
sedikit. Jumlah bunga betina pada fase pertumbuhan yang berbeda menunjukkan variasi terbesar sementara bunga jantan tidak terlalu bervariasi. Korelasi yang besar ditunjukkan antara tinggi tanaman dan panjang cabang, jumlah cabang dengan diameter batang yang berguna untuk seleksi genotip superior. Variasi genetik terbesar ditunjukkan pada analisis polyphenol oxidase.
Rao et al. (2008) mengevaluasi asosiasi dan keragaman genetik pada biji dan karakter pertumbuhan pada 32 kandidat jarak pagar berdaya hasil tinggi dari berbagai wilayah yang tersebar lebih dari 150.000 km2
Keragaman pada koleksi plasma nutfah Indonesia yang berasal dari berbagai daerah di Indonesia telah dicatat yaitu pada karakter bentuk dan warna daun, warna batang dan daun muda serta potensi produksi (Sudarmo et al., 2007) di India. Perbedaan karakter yang signifikan diamati pada semua karakter biji seperti morfologi biji, kandungan minyak dan pada karakter pertumbuhan seperti tinggi tanaman, rasio bunga jantan dan betina serta hasil biji pada uji keturunan. Secara umum heritabilitas dalam arti luas tinggi yaitu lebih dari 80% untuk semua sifat biji yang dipelajari. Sementara Sunil et al. (2008) mencacat karakter fenotipik tanaman jarak pagar secara in situ pada 4 ekogeografi yang berbeda di India. Mereka menyatakan adanya perbedaan pada 9 karakter yang diamati dari 162 aksesi di 4 daerah tersebut. Sebagai contoh yaitu tinggi tanaman dari 80% aksesi pada satu daerah kurang dari 1.5 m sementara pada daerah yang lain 60% kurang dari 1.5 m. Perbedaan yang sama juga dinyatakan pada jumlah biji dan kandungan dan komposisi minyak biji.
18
serta percabangan (Mardjono et al., 2007). Pada karakter-karakter yang berkaitan dengan daya hasil biji, keragaman ditemukan pada karakter umur mulai berbunga dan berbuah, jumlah tandan per tanaman dan jumlah kapsul per tandan. Pada tahun I penanaman dijumpai beberapa nomor yang dapat menghasilkan lebih dari 100 kapsul per tanaman (Hasnam et al., 2007; Sudarmo et al., 2007).
Kecenderungan genotip yang berasal dari ekogeografi yang sama untuk mengumpul menjadi satu klaster dicatat oleh Golil dan Pandya (2008) di India. Sementara Freitas et al. (2011) yang mempelajari keragaman genetik plasma nutfah jarak pagar Brazilia mencatat koefisien keragaman genetik yang tinggi pada hampir semua karakter morfologi yang diamati tetapi tidak menemukan hubungan pengelompokan dengan keragaman geografi asal aksesi.
19
Studi untuk menduga keragaman genetik jarak pagar menggunakan marka biokimia tidak banyak dilakukan. Prabakaran dan Sujatha (1999) menggunakan isozim peroksidase dan superoksida dismutase untuk menentukan filogeni dari jarak pagar dengan spesies-spesies lain. Yunus (2007) berhasil membuat diferensiasi intra spesies jarak pagar dari daerah berbeda di Jawa Tengah berdasarkan marka isoenzim sorbitol dehidrogenase, shikimate dehidrogenase, alkohol dehidrogenase dan isositrat dehidrogenase.
Keragaman Jarak Pagar Berbasis Marka Molekuler
Marka molekuler telah digunakan untuk menduga keragaman genetik aksesi-aksesi jarak pagar India dan beberapa aksesi tidak beracun dari Mexico menggunakan marka RAPD dan ISSR. Keragaman genetik aksesi dari India tergolong rendah dengan tingkat polimorfisme menggunakan marka RAPD dan ISSR masing-masing sebesar 42% dan 64%. Marka ISSR mampu membedakan aksesi-aksesi tidak beracun yang berasal dari Meksiko dan telah berhasil dikembangkan marka spesifik populasi SCAR untuk membedakan antara aksesi yang beracun dan tidak beracun yaitu marka ISPJ1 dan ISPJ2 (Basha dan Sujatha, 2007). Hasil yang hampir sama diperoleh Jubera et al., (2009) menggunakan 4 marka RAPD pada 7 aksesi jarak pagar India. Jarak genetik yang diperoleh berkisar 81.8 - 100. Sebaliknya hasil evaluasi yang diperoleh Subramanyam et al. (2009) pada 40 aksesi jarak pagar dari wilayah geografis berbeda di India ternyata menunjukkan keragaman berkisar 0 – 100% menggunakan 10 primer RAPD terpilih.
Ranade et al. (2008) mencoba menggunakan marka berbeda untuk mengakses keragaman jarak pagar India dan menunjukkan bahwa marka SPAR (Single Primer Amplification Reaction ) dapat membedakan aksesi-aksesi dari daerah berbeda. Aksesi yang diuji adalah jarak pagar yang telah dikenal maupun tanaman liar dan ternyata mempunyai keragaman genetik tinggi sehingga dapat dimanfaatkan untuk bahan perbaikan genetik jarak pagar di India.
20
polimorfisme berkisar 100-33.3 dan nilai PIC (Polymorphism Information Content) bervariasi 0.89-0.65. Dari 78 kombinasi, delapan kombinasi yang ditemukan menunjukkan polimorfisme 100% dan nilai PIC-nya berkisar 0.86-0.38. Berdasarkan dendrogram 17 genotip dikelompokkan menjadi 5 klaster pada nilai kesamaan 72%. (Umamaheswari et al., 2010).
Marka yang berbeda digunakan Tatikonda et al. (2009) untuk mengevaluasi keragaman genetik 48 aksesi jarak pagar dari enam negara bagian di India. Marka AFLP menghasilkan 680 (88%) fragmen polimorfik, 59 fragmen (8.7%) merupakan fragmen unik (pada aksesi tertentu) dan 108 (15.9%) fragmen jarang muncul (hadir dalam kurang dari 10% aksesi). Secara umum, aksesi yang berasal dari Andhra Pradesh beragam dan aksesi dari Chhattisgarh mempunyai banyak fragmen unik/langka.
Evaluasi keragaman genetik dilakukan terhadap 160 individu mewakili 8 populasi jarak pagar yang tersebar di Kenya menggunakan marka RAPD. Sepuluh primer acak menghasilkan 251 lokus dan hasil analisis menunjukkan keragaman antar populasi sebesar 53% sementara keragaman dalam populasi sebesar 47% (Machua et al., 2011).
21
Evaluasi keragaman genetik dilakukan terhadap jarak pagar di tempat yang diduga sebagai daerah asal penyebaran yaitu Meksiko menggunakan marka AFLP. Polimorfisme dari 152 marka yang digunakan sebesar 81.18% dan berdasarkan analisis klaster diperoleh nilai keragaman maksimum sebesar 89% (Medina et al., 2011).
Keragaman genetik dari 192 koleksi jarak pagar dari seluruh Brazil telah dipelajari menggunakan 96 marka RAPD dan 6 marka SSR. Hanya 23 marka RAPD dan 1 marka SSR yang polimorf. Lima dari 6 marka SSR yang digunakan menunjukkan bahwa semua aksesi yang diuji homosigot. Nilai kemungkinan bahwa aksesi-aksesi yang tidak dapat dibedakan adalah merupakan duplikat berkisar antara 83 sampai 99%. Tidak ditemukan adanya hubungan antara pengelompokan berdasar marka molekuler dengan daerah asal aksesi (Rosado et al., 2010). Sementara Santos et al. (2010) mendapatkan hasil berbeda pada evaluasi hubungan genetik 12 aksesi jarak pagar dari Brazilia berdasarkan marka AFLP. Nilai keragaman dalam populasi dari 12 aksesi diketahui sebesar 70%, sementara keragaman di antara aksesi sebesar 27.5%. Pengelompokan berdasar dendrogram cenderung mengikuti daerah asal aksesi masing-masing.
Menggunakan 2.500 biji dari 17 varietas komersial yang berasal dari Afrika, Asia Timur, Amerika Tengah dan Amerika Selatan Ambrosi et al. (2010) melakukan evaluasi dengan marka RAPD, ISSR dan SSR. Benih komersial tersebut ternyata tidak merepresentasikan gene pool jarak pagar dan pengelompokan yang dihasilkan tidak berkorelasi dengan wilayah geografis. Marka ISSR yang digunakan oleh Tanya et al. (2011) dapat membedakan aksesi-aksesi jarak pagar yang berasal dari daerah berbeda (Meksiko, Cina, Vietnam dan Thailand) tetapi keragaman yang ditunjukkan dalam grup hanya sebesar 37%.
22
Sekitar 225 aksesi jarak pagar dari lebih dari 30 negara di Amerika Latin, Afrika dan Asia dianalisis menggunakan marka AFLP. Keragaman genetik rendah ditemukan pada aksesi dari Afrika dan India sementara keragaman tinggi ditemukan pada aksesi dari Guatemala dan negara Amerika Latin lainnya (Montes
et al., 2009).
Pendugaan keragaman genetik menggunakan RAPD, AFLP dan cTBP (Combinatorial Tubulin Based Polymorphism) pada 38 aksesi jarak pagar dari 13 negara pada 3 benua menunjukkan keragaman yang rendah. Sementara pada aksesi yang sama dijumpai adanya keragaman pada beberapa sifat fenotipik, fisiologi dan biokimia penting yaitu ukuran buah, efisiensi penggunaan air dan kandungan minyak biji. Hal ini mengimplikasikan adanya mekanisme epigenetik pada jarak pagar yang menarik untuk dipelajari (Popluecai et al., 2009).
23
BAB III
PENGEMBANGAN MARKA
SIMPLE SEQUENCE REPEAT
(SSR) UNTUK
Jatropha
spp.
Abstrak
Pemuliaan tanaman jarak pagar (Jatropha curcas L.) untuk menghasilkan kultivar berdaya hasil dan berkadar minyak tinggi perlu dilakukan. Penggunaan marka molekuler dapat membantu mempercepat tercapainya tujuan pemuliaan tanaman jarak pagar. Marka simple sequence repeat (SSR) merupakan marka ko-dominan yang efektif untuk mendukung program pemuliaan tanaman, tetapi penerapannya pada jarak pagar masih terbatas. Penelitian yang dilakukan bertujuan: (i) mendesain primer spesifik SSR menggunakan aksesi DNA jarak pagar yang tersedia di GenBank DNA database dan (ii) mengevaluasi efektivitas pasangan primer yang didesain untuk menghasilkan marka SSR yang polimorfik untuk jarak pagar dan J. multifida. Dua puluh delapan pasang primer spesifik SSR telah berhasil didesain menggunakan aksesi DNA asal jarak pagar yang ada di GenBank DNA database. DNA genomik jarak pagar dan J. multifida yang diisolasi dapat digunakan sebagai templat untuk amplifikasi PCR. Dari 28 pasang primer yang dikembangkan, semuanya mampu menghasilkan marker SSR dari genom jarak pagar dan hanya 14 pasang primer yang menghasilkan marker SSR dari genom J. multifida. Dari 19 pasangan primer spesifik SSR yang dievaluasi mampu dihasilkan 44 alel dengan ukuran produk amplifikasi berkisar antara 100 bp – 360 bp. Sebanyak 35 alel (79.5%) yang diamati merupakan alel yang polimorfik. Marker SSR yang didapatkan tidak polimorfik aksesi jarak pagar atau intra-aksesi J. multifida tetapi polimorfik untuk inter-aksesi kedua spesies. Karena marker SSR yang dihasilkan bersifat polimorfik untuk aksesi jarak pagar dengan aksesi J. multifida maka dapat digunakan sebagai marka untuk mendeteksi hasil persilangan F1 inter-spesies J. curcas x J. multifida.
24
Development of Simple Sequence Repeats (SSRs) Markers for Jatropha spp.
Abstract
Breeding of physic nut (Jatropha curcas L.) to obtain new cultivars that have high yield and high oil content need to be conducted. Molecular marker could be used to assist breeding of physic nut. Simple sequence repeat (SSR) marker is a co-dominant marker and theoretically it could be used to support physic nut breeding program. However, only limited information were available regarding molecular analysis of physic nut. The objectives of this research were: (i) to design SSR specific primer based on DNA sequences available in the GenBank DNA database and (ii) to evaluate effectiveness of the primer pairs to produce polymorphic SSR markers for J. curcas and J. multifida. Twenty eight primer pairs were designed and developed using physic nut DNA available in the GenBank DNA database. Total genomic DNA isolated from J. curcas and J. multifida could be used as DNA templates for PCR amplification. The 28 primer pairs developed in this research yielded SSR marker using J. curcas genomic DNA, while only 14 out of 28 primer pairs developed yielded SSR markers using J. multifida genomic DNA. As many as 44 alleles with the size of amplified products ranged from 100 bp – 360 bp were identified. Thirty five alleles (79.5%) out of 44 identified ones were polymorphic. Results of analysis indicated that identified SSR markers generated using the designed primers were not polymorphic intra accession of J. curcas nor intra-accession of J. multifida. However, the generated SSR markers were polymorphic for inter-accession of the two Jatropha species. Since the generated markers were only polymorphic for J. curcas and J. multifida, they could be used as marker for identifying interspecific F1 hybrids derived from crossing between J. curcas and J. multifida.
25
Pendahuluan
Jarak pagar (Jatropha curcas L.) adalah tanaman tahunan multifungsi, yang merupakan anggota Euphorbiaceae. Pada awalnya jarak pagar digunakan sebagai tanaman obat. Beberapa tahun terakhir ini jarak pagar lebih dikenal sebagai tanaman penghasil minyak yang dapat digunakan untuk biodiesel. Jarak pagar berasal dari Mexico dan Amerika Tengah yang kemudian menyebar ke daerah tropis maupun subtropis. Tanaman jarak pagar pada awalnya ditanam sebagai pagar pengaman atau untuk keperluan tradisional lainnya (Heller, 1996).
Pada beberapa tahun terakhir ini nilai ekonomi jarak pagar sebagai penghasil biodiesel meningkat pesat (Fairless, 2007). Hal penting yang harus dilakukan untuk dapat menggunakan jarak pagar sebagai penghasil biodiesel adalah pengembangan kultivar yang memiliki hasil biji dan kadar minyak tinggi serta dapat beradaptasi dengan baik pada berbagai kondisi lingkungan (Divakara
et al., 2010).
Plasma nutfah jarak pagar telah dikoleksi dan dianalisis di Brazil, Indonesia, dan Cina (Ou et al., 2009; Tatikonda et al., 2009). Jarak pagar memiliki genom heterosigot dan beberapa penelitian menunjukkan nilai interaksi genetik dan lingkungan yang cukup besar (Makkar et al., 1997; Kaushik et al., 2007) sehingga program pemuliaan konvensional tidak akan efektif. Strategi pemuliaan berbasis genom sangat penting dilakukan. Sampai saat ini pemetaan genetik jarak pagar belum dilakukan dan informasi genetik berdasarkan marka molekuler masih sangat sedikit ketersediaannya.
26
khususnya pada spesies yang menunjukkan variasi genetik rendah, pada populasi inbred dan populasi yang diperoleh dari daerah-daerah berdekatan sehingga sulit dipilah-pilahkan dengan pendekatan lain (Röder et al., 1995). Keuntungan SSR secara alami adalah: (i) multipel alel SSR dapat dideteksi pada lokus tunggal menggunakan penapisan berbasis PCR, (ii) SSR biasanya terdistribusi pada seluruh genom, (iii) sifatnya ko-dominan, (iv) jumlah DNA yang dibutuhkan hanya sedikit, dan (v) analisis dapat dilakukan secara semi otomatis (Robinson et al., 2004).
Jarak pagar di Indonesia berpotensi besar sebagai penghasil bahan bakar nabati sehingga perlu dilakukan penelitian untuk mengoptimalkan keberadaan plasma nutfah tersebut dengan program pemuliaan tanaman. Sampai saat ini belum ada penelitian yang memanfaatkan marka SSR untuk evaluasi keragaman genetik jarak pagar Indonesia. Penelitian ini dilakukan untuk mengawali langkah-langkah pemuliaan tanaman pada jarak pagar dengan basis marka SSR.
Tujuan penelitian ini adalah adalah untuk: (i) mendesain primer spesifik SSR menggunakan aksesi DNA jarak pagar yang tersedia di GenBank DNA database dan (ii) mengevaluasi efektivitas pasangan primer yang didesain untuk menghasilkan marka SSR yang polimorfik untuk jarak pagar dan J. multifida L. Marka-marka SSR yang ditemukan pada penelitian ini diharapkan dapat memberikan sumbangan bagi percepatan kegiatan pemuliaan tanaman jarak pagar untuk mendapatkan varietas unggul baru.yaitu melalui evaluasi keragama genetik.
Bahan dan Metode
Analisis molekuler dan penelusuran serta analisis menggunakan perangkat lunak online dilakukan di Laboratorium Biologi Molekuler Tanaman, Departemen Agronomi dan Hortikultura, Fakultas Pertanian, IPB. Penelitian dilakukan dari bulan Mei sampai dengan bulan Nopember 2008.
27
Aksesi J. multifida digunakan untuk mengevaluasi peluang penggunaan primer yang dievaluasi pada spesies Jatropha yang masih sekerabat dengan jarak pagar.
Ekstraksi DNA
Ekstraksi DNA pertama kali dilakukan dengan metode CTAB standar yang dikembangkan oleh Doyle dan Doyle (1990) dan digunakan untuk ekstraksi jarak pagar oleh Basha dan Sujatha (2007). Modifikasi dilakukan dengan meningkatkan konsentrasi NaCl pada buffer ekstraksi dari 1.4 M menjadi 3.5 M dan menambahkan NaCl hinga konsentrasi 2 M pada saat presipitasi bersamaan dengan penambahan isopropanol sesuai dengan metode yang dikembangkan oleh Sudheer et al. (2009). Metode modifikasi selengkapnya diuraikan sebagai berikut. Sebanyak 0.1 g daun muda (daun berwarna keunguan, sedikit transparan dengan lebar sekitar 2-3 cm) dari tanaman sampel yang ditumbuhkan di lapangan, digerus dengan 500 µL buffer ekstraksi (CTAB 2%, 100 mM Tris HCl pH 8, 3.5 M NaCl, 0.5 M EDTA) dan 1% polyvinylpolypyrolydone (PVP). Ekstrak daun kemudian dipindahkan ke dalam tabung mikro berukuran 2.000 µL, ditambahkan 1.5% β -merkaptoetanol dan diinkubasi pada suhu 65o
Total DNA genomik yang didapat dikuantifikasi menggunakan spektrofotometer UV (Shimadzu UV - 1800) pada λ 260 nm dan kemurniannya
ditentukan dengan menghitung rasio absorban pada λ 260 dan 280 nm sesuai dengan prosedur oleh Sambrook et al. (1989). Konsentrasi dan kemurnian DNA C selama 90 menit. Setelah inkubasi, ke dalam campuran ditambahkan kloroform : isoamil alkohol (24:1) dengan volume sebanding dan dikocok perlahan selama 10 menit.
28
juga dicek dengan perbandingan hasil elektroforesis sampel DNA dengan standar pada gel agarosa 1%.
Desain primer SSR
Penelusuran aksesi DNA jarak pagar yang ada di GenBank database dilakukan secara online pada situs. Setiap aksesi DNA jarak pagar yang diperoleh dari basis data DNA, dievaluasi ada tidaknya runutan nukleotida yang merupakan simple sequence repeat (SSR). Panjang sekuen serta posisi SSR dievaluasi untuk melihat mungkin atau tidaknya primer didesain. Selain itu, agar tidak duplikasi, semua aksesi dianalisis kesamaan runutan nukleotidanya dengan multiple alignment menggunakan perangkat lunak online ClustalW yang tersedia di situs http://www.ebi.ac.uk/Tools/clustalw2/index.html. Selanjutnya, dari aksesi DNA yang unik dan positif teridentifikasi membawa SSR, didesain primer spesifik yang mengapit runutan nukleotida SSR-nya. Desain primer SSR dilakukan dengan menggunakan perangkat lunak Primer3 (Rozen et al., 2000)yang tersedia secara online pada situs http://frodo.wi.mit.edu/. Primer yang berhasil didesain digunakan untuk mengamplifikasi sampel DNA jarak pagar dan J. multifida.
Amplifikasi DNA dan separasi hasil amplifikasi
DNA yang telah diekstraksi diamplifikasi PCR menggunakan primer yang berhasil didesain. Volume reaksi amplifikasi PCR yang digunakan adalah 25 µL, yang masing-masing terdiri atas 1X buffer reaksi, 0.1 µM dNTPs, 1 unit Real Taq DNA Polymerase (Real Biotech Corporation) dan 20 ng templat DNA. Reaksi amplifikasi PCR dilakukan menggunakan GeneAmp PCR System 2400 (Perkin Elmer) dengan denaturasi awal pada 95oC selama 5 menit; diikuti dengan 35 siklus yang masing-masing siklus terdiri atas tahapan denaturasi pada suhu 94oC selama 30 detik, penempelan primer (primer annealing) pada suhu yang sesuai untuk masing-masing pasangan primer selama 1 menit, pemanjangan primer (primer elongation) pada suhu 72oC selama 1 menit. Pada akhir reaksi ditambahkan satu siklus finalextension pada suhu 72o
DNA hasil amplifikasi dievaluasi dengan dua sistim elekktroforesis, yaitu elektroforesis gel agarosa (1.5%) yang digunakan untuk konfirmasi keberadaan
29
hasil amplifikasi dan PAGE (polyacrylamide gel electrophoresis) yang digunakan untuk skoring alel SSR. Hasil elektroforesis gel agarosa divisualisasi dengan menggunakan pewarnaan etidium bromida dan difoto di bawah penyinaran UV menggunakan UV transluminescent. Elektroforesis dengan gel akrilamid digunakan untuk memperoleh resolusi yang lebih baik sehingga komposisi alel SSR yang muncul dapat teridentifikasi. Analisis menggunakan PAGE (40% akrilamid/bis-akrilamid, 10% amonium persulfat, 5X buffer TBE, urea dan TEMED) dilakukan dengan Dedicated Height Sequencer (Cole-Parmer) menggunakan buffer TBE 1X pada tegangan konstan 1.100 V selama 3 jam. Volume hasil PCR yang dianalisis adalah 1.8 µL dengan 60 sampel per gel. Hasil PAGE divisualisasi dengan pewarnaan menggunakan pewarnaan perak (silver staining). Marka DNA berukuran kelipatan 100 bp (100 bp ladder) digunakan untuk membantu menentukan ukuran potongan DNA hasil amplifikasi PCR.
Hasil
Ekstraksi DNA
Ekstraksi DNA tanaman sampel (Tabel 1) dengan metode CTAB standar menghasilkan DNA yang kurang baik dan tidak konsisten. Pengujian sampel DNA total pada gel agarosa menunjukkan sinyal yang kurang jelas bahkan dua sampel (2 dan 4) hampir tidak menunjukkan sinyal sama sekali. Intensitas pengotor sangat tinggi tampak pada bagian bawah gel (Gambar 3A). Ekstraksi dengan metode yang dikembangkan oleh Sudheer et al. (2009) menghasilkan DNA yang cukup baik. Pengujian dengan gel agarosa menunjukkan sinyal yang terang, jernih serta konsisten dan intensitas pengotor lebih sedikit (Gambar 3B). Pengukuran kuantitas DNA total mendapatkan hasil sekitar 100 – 150 ng/µL.
Desain primer SSR
30
runutan DNA tinggi hanya dipilih satu sebagai representasi kelompoknya agar tidak terjadi duplikasi lokus yang diamplifikasi. Pemilihan pasangan primer yang akan dievaluasi juga mempertimbangkan kesesuaian melting point antara pasangan primer forward dan reverse-nya, yaitu dipilih yang melting point-nya sama atau paling berdekatan (Dieffenbach et al., 1993). Setelah semua faktor yang diperlukan dijadikan pertimbangan, 28 pasang primer SSR spesifik telah terpilih sebagai hasil akhir kegiatan (Tabel 2).
Tabel 1 Aksesi jarak pagar dan kerabatnya yang digunakan untuk evaluasi primer SSR yang dikembangkan dalam penelitian
No. Genotip/ Aksesi Asal provenan 1. J. multifida / # 2 Sukabumi 2. J. multifida / # 1 Bogor 3. J. curcas /PT 26-2 Lampung 4. J. curcas /IP -1A-2 NTB 5. J. curcas /HS 49-2 NTT
6. J. curcas /SP 6-3 Sulawesi Selatan 7. J. curcas /IP-M-3 Jawa Timur
Gambar 3