DETEKSI DIFERENSIAL POTYVIRUS DAN FABAVIRUS
DENGAN REVERSE TRANSCRIPTION-POLYMERASE CHAIN
REACTION (RT-PCR)
VISHORA SATYANI
DEPARTEMEN PROTEKSI TANAMAN
FAKULTAS PERTANIAN
INSTITUT PERTANIAN BOGOR
BOGOR
ABSTRAK
VISHORA SATYANI. Deteksi Diferensial Potyvirus dan Fabavirus dengan Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Dibimbing oleh GEDE SUASTIKA.
Tanaman nilam (Pogostemon cablin Benth) sangat potensial untuk dikembangkan karena minyak nilam merupakan bahan baku yang penting dalam industri. Terdapat banyak kendala dalam upaya peningkatan pengembangan produksi tanaman nilam, salah satunya adalah adanya gangguan dari organisme pengganggu tumbuhan (OPT). Potyvirus dan Fabavirus merupakan virus yang yang sudah diketahui dapat menyerang tanaman nilam. Gejala yang ditimbulkan pada tanaman nilam oleh kedua virus ini hampir tidak bisa dibedakan. Penelitian ini bertujuan untuk menyiapkan metode diferensial yang dapat membedakan dengan tepat infeksi Potyvirus dan Fabavirus pada tanaman nilam baik secara tunggal maupun ganda melalui RT-PCR. Sumber virus merupakan tanaman nilam yang terinfeksi alami Potyvirus atau Fabavirus yang telah diverifikasi dengan uji serologi. RNA total diekstraksi dari tanaman nilam sumber virus dengan Bench-Top Protocols for Xprep Plant RNA Mini Kit (PKT Korea). cDNA (complementary DNA) disintesis dengan teknik Reverse Transcription (RT). PCR dilakukan dengan satu siklus pada 95 ºC selama 5 menit; 10 siklus pada 95 ºC selama 1 menit, pada 51 ºC selama 1 menit, dan pada 72 ºC selama 1 menit; 30 siklus pada 94 ºC selama 1 menit, pada 54 ºC selama 1 menit, dan pada 72 ºC selama 2 menit untuk; dan satu siklus pada 72 ºC selama 10 menit. Primer yang digunakan untuk deteksi Potyvirus adalah CPUP-F (5’-TGAGGATCCTGGTGY ATHGARAAYGG-3’, Y=C/T, H=A/T/C, R=A/G), CP9502-R (5’-GCGGATCCT TTTTTTTTTTTTTTTT-3’) dan untuk Fabavirus adalah BBWVVSSP (5’-GTBTCDAGTGCTYTDGAAGG-3’,B=C/G/T,D=A/G/T,Y=C/T),BBWVKMRM (5’-TDGWDCCATCVAGICKCATTTT-3’, W=A/T; V=A/C/G; I=Inosine; K=G/T). Kedua pasang primer ini dapat mengamplifikasi masing-masing genom Potyvirus sepanjang 800 bp maupun Fabavirus sepanjang 322 bp secara spesifik baik dalam uji terpisah maupun campuran. Metode deteksi diferensial ini berhasil diterapkan untuk mendeteksi infeksi Potyvirus dan Fabavirus pada sampel tanaman nilam bergejala mosaik yang dikoleksi dari wilayah Bogor yaitu Gunung Bunder dan Cicurug. Pada deteksi tersebut dengan jelas dapat dibedakan tanaman-tanaman yang hanya terinfeksi Potyvirus atau Fabavirus saja atau terinfeksi ganda kedua virus tersebut. Oleh karena itu, metode berbasis RT-PCR menggunakan campuran pasangan primer CPUP-F/CP952-R dan BBWVVSSP/BBWVKMRM valid digunakan untuk deteksi diferensial.
Kata kunci : Potyvirus, Fabavirus, RT-PCR, deteksi diferensial, nilam, Pogostemon cablin
DETEKSI DIFERENSIAL POTYVIRUS DAN FABAVIRUS
DENGAN REVERSE TRANSCRIPTION-POLYMERASE CHAIN
REACTION (RT-PCR)
VISHORA SATYANI
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Pertanian pada Departemen Proteksi Tanaman, Fakultas Pertanian
Institut Pertanian Bogor
DEPARTEMEN PROTEKSI TANAMAN
FAKULTAS PERTANIAN
INSTITUT PERTANIAN BOGOR
BOGOR
LEMBAR PENGESAHAN
Judul : Deteksi Diferensial Potyvirus dan Fabavirus dengan Reverse Transcription-Polymerase Chain Reaction (RT-PCR)
Nama : Vishora Satyani
NRP : A34070024
Disetujui,
Diketahui,
Ketua Departemen Proteksi Tanaman
Dr. Ir. Abdjad Asih Nawangsih, M.Si. NIP. 19650621 198910 2 001
Tanggal Lulus :
Pembimbing I
Dr. Ir. Gede Suastika, M.Sc. NIP.19620607 198703 1 003
RIWAYAT HIDUP
Penulis dilahirkan di Pati pada tanggal 25 Agustus 1989 dari pasangan Harno (Alm) dan Sri Hadi Kuswariati. Penulis merupakan anak pertama dari dua bersaudara.
Penulis menyelesaikan pendidikan menengah umum di SMA Negeri 1 Pati pada tahun 2007. Pada tahun yang sama penulis diterima sebagai mahasiswa program studi Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor melalui jalur Ujian Saringan Masuk IPB (USMI).
Selama kuliah, penulis mengikuti kegiatan kepanitiaan dan organisasi di IPB, yaitu Himpunan Mahasiswa Proteksi Tanaman (HIMASITA) sebagai Staf Divisi Fasilitas dan Properti periode 2009-2010. Penulis pernah mengikuti kegiatan Magang di Laboratorium Pengamat Hama Penyakit Dinas Pertanian dan Peternakan Kabupaten Pati pada tahun 2009. Penulis juga aktif mengikuti kegiatan Program Kreativitas Mahasiswa bidang Penelitian (PKM-P) yang berjudul “Pemanfaatan Bakteri Kitinolitik dalam Pengendalian Pascapanen Penyakit Antraknosa (Colletotrichum gloeosporioides) pada Buah Cabai Merah” pada tahun 2009-2010. Selain itu juga sebagai penyaji pada International Seminar and The 21st National Congress of The Indonesian Phytopathological Society 2011.
PRAKATA
Puji serta syukur penulis panjatkan ke hadirat Allah SWT atas limpahan rahmat dan karunia-Nya, sehingga penulis dapat menyelesaikan penulisan skripsi yang berjudul “Deteksi Diferensial Potyvirus dan Fabavirus dengan Reverse Transcription-Polymerase Chain Reaction (RT-PCR)”. Skripsi ini disusun sebagai syarat untuk memperoleh gelar Sarjana Pertanian pada program studi Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor.
Rasa terima kasih yang tulus penulis sampaikan untuk kedua orang tua tercinta Harno (Alm), Sri Hadi Kuswariati, adikku tersayang Desyana Rachmawati, serta keluarga yang selalu memberikan kasih sayang, semangat, nasihat, dan doa. Terima kasih kepada Dr. Ir. Gede Suastika, M.Sc. yang telah bersedia menjadi dosen pembimbing skripsi dan dosen pembimbing akademik yang telah memberikan arahan kepada penulis sehingga skripsi ini dapat terselesaikan dengan baik. Penulis juga mengucapkan terima kasih kepada Dr. Ir. R Yayi Munara Kusumah, M.Si. selaku dosen penguji tamu yang telah menyediakan waktu dan perhatiannya.
Pada kesempatan ini, penulis mengucapkan terima kasih kepada rekan-rekan Laboratorium Virologi Tumbuhan Mbak Tuti, Mbak Miftah, Mbak Pipit, Mbak Mel, Mbak Dwi, Kak Aceu, Pak Irwan, Ibu Asni, Ibu Rita, Pak Edi, Sherly, Harwan, Erika, Rizki, Avanty, Santi, Rita, Mia, Taher dan Fitri yang telah membantu penulis selama di laboratorium. Terima kasih untuk sahabat-sahabatku tercinta, Triyastuti Prasetyoningrum, Mey Fitriyani, Tatit Satrini dan Sani Nihlatussania yang setia menemani dan membantu penulis serta senantiasa memberikan motivasi, doa dan kasih sayang yang tulus. Terima kasih juga untuk Anik, Icha, Riska, Osmond, dan teman-teman DPT 44 semua serta semua pihak yang tidak dapat disebutkan satu per satu.
Penulis menyadari bahwa skripsi ini masih jauh dari kesempurnaan. Oleh karena itu, penulis mengharapkan kritik dan saran yang bersifat membangun untuk kedepannya. Akhir kata penulis serahkan skripsi ini dengan penuh rasa bangga.
Bogor, Maret 2012
DAFTAR ISI
Halaman
DAFTAR TABEL ... viii
DAFTAR GAMBAR ... ix PENDAHULUAN ... 1 Latar Belakang ... 1 Tujuan Penelitian ... 2 Manfaat Penelitian ... 2 TINJAUAN PUSTAKA ... 3
Tanaman Nilam (Pogostemon cablin Benth) ... 3
Potyvirus ... 4
Fabavirus ... 5
Enzyme-Linked Immunosorbent Assay (ELISA) ... 7
Reverse Transcription-Polymerase Chain Reaction (RT-PCR) ... 8
BAHAN DAN METODE ... 10
Tempat dan Waktu Penelitian ... 10
Metode Penelitian ... 10
Pengambilan Sampel Tanaman Nilam Bergejala Mosaik di Lapangan ... 10
Deteksi Potyvirus melalui I-ELISA ... 10
Deteksi Fabavirus melalui DAS-ELISA ... 11
Deteksi Diferensial Potyvirus dan Fabavirus melalui RT-PCR ... 11
Ekstraksi RNA Total ... 12
Sintesis cDNA ... 12
Amplifikasi DNA dengan PCR ... 13
Elektroforesis ... 14
HASIL DAN PEMBAHASAN ... 17
Validasi Pasangan Primer untuk Deteksi Potyvirus ... 18
Validasi Pasangan Primer untuk Deteksi Fabavirus ... 19
Validasi Pasangan Primer untuk Deteksi Simultan Potyvirus dan Fabavirus ... 20
Penerapan Metode RT-PCR untuk Deteksi Simultan Potyvirus dan Fabavirus pada Sampel dari Lapangan ... 21
KESIMPULAN DAN SARAN ... 24
Kesimpulan ... 24
Saran ... 24
DAFTAR TABEL
Halaman 1. Reagensia PCR dan konsentrasi yang diperlukan untuk mendeteksi
validitas pasangan primer Potyvirus yang digunakan secara terpisah terhadap 3 template cDNA yang berbeda (Potyvirus, Fabavirus
dan keduanya) ... 15 2. Reagensia PCR dan konsentrasi yang diperlukan untuk mendeteksi
validitas pasangan primer Fabavirus yang digunakan secara terpisah terhadap 3 template cDNA yang berbeda (Potyvirus, Fabavirus
dan keduanya) ... 15 3. Reagensia PCR dan konsentrasi yang diperlukan untuk mendeteksi
validitas pasangan primer Potyvirus dan Fabavirus yang digunakan secara bersamaan terhadap 3 template cDNA yang berbeda
DAFTAR GAMBAR
Halaman 1. Partikel Potyvirus berbentuk seperti benang, memanjang
(filamentous) dan lentur (flexuous). (Natsuaki et al. 1994) ... 4 2. Partikel Fabavirus berbentuk seperti bulat (isometric)
(Natsuaki et al. 1994) ... 6 3. Gejala mosaik pada daun nilam yang disebabkan oleh Potyvirus
dan atau Fabavirus ... 17 4. Hasil amplifikasi DNA genom virus dengan metode RT-PCR
menggunakan pasangan primer spesifik Potyvirus ... 18 5. Hasil amplifikasi DNA genom virus dengan metode RT-PCR
menggunakan pasangan primer spesifik Fabavirus ... 19 6. Hasil amplifikasi DNA genom virus dengan metode RT-PCR
menggunakan campuran primer Potyvirus dan Fabavirus ... 21 7. Hasil amplifikasi DNA genom virus dengan metode RT-PCR
menggunakan campuran pasangan primer Potyvirus dan Fabavirus
PENDAHULUAN
Latar Belakang
Minyak atsiri yang berasal dari minyak nilam (Pogostemon cablin Benth) merupakan komoditi andalan Indonesia. Hampir 90% kebutuhan minyak nilam dunia dipasok dari Indonesia, karena mutunya dinilai yang paling baik. Ekspor minyak nilam Indonesia dari tahun ke tahun terus mengalami peningkatan (hampir 6% per tahun) sesuai dengan meningkatnya permintaan minyak nilam dipasaran internasional. Volume ekspor ini juga tidak lepas dari usaha perluasan areal pertanaman diberbagai daerah di Indonesia. Minyak nilam terutama digunakan sebagai bahan fiksatif dan pewangi dalam industri parfurn, sabun dan kosmetika. Selain itu minyak nilam juga digunakan sebagai bahan baku obat-obatan dan pestisida. Hampir seluruh pertanaman nilam di Indonesia merupakan pertanaman rakyat yang melibatkan 32.870 kepala keluarga petani (Direktorat Jenderal Perkebunan 2006).
Namun demikian, produksi minyak atsiri di Indonesia masih rendah. Salah satu sebabnya adalah adanya serangan organisme pengganggu tumbuhan (OPT) yang menyerang tanaman nilam terutama virus. Penyakit virus yang banyak menyerang tanaman nilam adalah penyakit mosaik. Tanaman sakit memperlihatkan gejala berupa perubahan warna pada daun yaitu mosaik hijau tua dan hijau muda dan terjadi malformasi. Tanaman menjadi kerdil sehingga sangat mengurangi kualitas dan kuantitas panen biomassa. Noveriza et al. (2010) melaporkan bahwa penyakit mosaik pada tanaman nilam di daerah Jawa Barat berasosiasi dengan infeksi dua jenis virus yang berbeda yaitu Potyvirus dan Fabavirus. Kedua jenis virus ini ditemukan menginfeksi tanaman nilam di lapangan baik secara tunggal maupun ganda dengan gejala yang tidak dapat dibedakan.
Menurut Hartono et al. (2006) Patchouli mottle virus (PaMoV) merupakan patogen penyebab penyakit yang sangat penting dan menyebabkan kerugian hasil di sentra produksi nilam di Indonesia. Di samping PaMOV, Patchouli mild mosaic virus (PaMMV) dilaporkan menyerang tanaman nilam di Jepang (Natsuaki et al. 1994).
2
PaMoV merupakan spesies virus yang termasuk ke dalam genus Potyvirus, sedangkan PaMMV merupakan spesies virus yang termasuk ke dalam genus Fabavirus (Natsuaki et al. 1994). Gejala yang ditimbulkan oleh PaMoV maupun PaMMV secara sendiri-sendiri maupun bersama pada tanaman nilam tidak dapat dibedakan (Natsuaki et al. 1994). Hasil pengujian ELISA sampel tanaman nilam dari Bogor dan Cianjur menunjukkan reaksi positif terhadap Potyvirus dan CMV (Sukamto et al. 2007).
Hal ini menunjukkan bahwa Potyvirus dan Fabavirus telah dilaporkan keberadaannya di pertanaman nilam di Indonesia. Oleh karena itu, diperlukan suatu metode yang dapat mendeteksi kedua virus ini secara terpisah (differensial diagnostic metode). Pada penelitian ini, digunakan dua pasang primer dalam Reverse Transcription-Polymerase Chain Reaction (RT-PCR) yang dapat mengamplifikasi sebagian genom Potyvirus dan Fabavirus secara terpisah.
Tujuan Penelitian
Penelitian ini bertujuan untuk menyiapkan metode deteksi diferensial Potyvirus dan Fabavirus yang menginfeksi tanaman nilam.
Manfaat Penelitian
Metode yang diperoleh dalam penelitian ini akan sangat berguna untuk mendeteksi Potyvirus dan Fabavirus yang menginfeksi tanaman nilam secara akurat.
TINJAUAN PUSTAKA
Tanaman Nilam (Pogostemon cablin Benth)
Nilam (Pogostemon cablin Benth) merupakan bahan baku minyak nilam (patchouli oil) yang merupakan komoditi ekspor terbesar (60%) dari ekspor minyak atsiri Indonesia (Balai Besar Pelatihan Pertanian 2011). Nilam menjadi
komoditas ekspor penting di Indonesia karena 90% kebutuhan dunia akan minyak nilam dipasok oleh Indonesia. Minyak nilam banyak digunakan sebagai bahan baku, bahan pencampur dan fiksatif (pengikat wangi-wangian) dalam industri parfum, farmasi dan kosmetik, serta industri makanan dan minuman (Balai
Penelitian Tanaman Rempah dan Obat 1991 & Mustofa 1991). Hal ini disebabkan
karena daya lekatnya yang kuat sehingga aroma wangi tidak mudah hilang karena tercuci atau menguap, dapat larut dalam alkohol dan dapat dicampur dengan minyak eteris lainnya.
Terdapat tiga jenis tanaman nilam yang tumbuh di Indonesia, yaitu : nilam Aceh (Pogostemon cablin Benth) yang kandungan minyaknya tinggi, yaitu 2,5-5%, nilam Jawa atau nilam hutan (Pogostemon heyneanus Benth) dan nilam sabun (Pogostemon hortensis Backer) kandungan minyaknya masing-masing 0,5-1,5%. Pada bulan Agustus tahun 2005 Menteri Pertanian telah melepas tiga varietas nilam unggul yaitu : Sidikalang, Lhokseumawe dan Tapak Tuan dengan kadar minyak cukup tinggi yaitu 2-4% dan produksi minyak per hektar relatif tinggi. Produk yang dihasilkan dari usaha tani nilam adalah terna (daun dan ranting), melalui proses penyulingan dihasilkan minyak nilam.
Tanaman nilam dapat tumbuh dan berproduksi pada daerah dengan ketinggian 0-1.200 m di atas permukaan laut (dpl), namun akan tumbuh dan berproduksi optimum pada daerah dengan ketinggian 10-400 m dpl. Curah hujan yang dikehendaki sekitar 2.300-3.000 mm per tahun dan kelembaban lebih dari 60%. Suhu udara antara 24-28 oC dengan intensitas penyinaran matahari berkisar 75-100%. Tanah yang dikehendaki adalah subur, gembur dan mengandung bahan organik dengan pH 5,5-7. Membutuhkan banyak air, tetapi tidak tahan genangan air sehingga perlu dibuat drainase yang baik.
4
Potyvirus
Potyvirus merupakan virus yang menyerang tanaman nilam dan termasuk ke dalam famili Potyviridae yang merupakan famili terbesar dalam virus tanaman yang tergolong positif sense single-stranded RNA (ssRNA(+)) yang dikenal sekarang ini dan merupakan genus terbesar dalam famili tersebut. Semua anggota dari genus tersebut memiliki virion berbentuk filamen dan fleksibel (Gibs et al. 2008). Seperti yang dilaporkan juga oleh Natsuaki et al. (1994), bahwa partikel Potyvirus berbentuk seperti benang, memanjang (filamentous) dan lentur (flexuous) (Gambar 1).
Gambar 1 Partikel Potyvirus berbentuk seperti benang, memanjang (filamentous) dan lentur (flexuous) (Natsuaki et al. 1994)
Taksonomi Potyvirus, sebagai kelompok virus tumbuhan terbesar yang menyerang tanaman, sampai saat ini masih menjadi perdebatan karena besarnya variasi antara spesies (Ward & Shukla 1990). Runutan asam amino protein selubung (coat protein/CP) telah dicoba digunakan untuk menilai kekerabatan 17 isolat dari delapan spesies Potyvirus (Shukla & Ward 1988). Hasil kajian menunjukkan antara spesies Potyvirus yang berbeda terdapat tingkat kesamaan runutan asam amino dari CP sebesar 38-71% dan antara galur yang berbeda dalam spesies virus yang sama mempunyai tingkat kesamaan mencapai 99% (Akin 2002).
Genus Potyvirus memiliki inang yang cukup banyak dan bervariasi dari beberapa famili tanaman diantaranya yaitu Amaranthaceae, Chenopodiaceae, Compositae, Labiateae, dan Solanaceae. Anggota dari genus Potyvirus memiliki panjang partikel minimal 700 nm. Genom dikarakteristikkan oleh 5’untranslated region (5’UTR) dimana terhubung dengan genome-linked protein (VPg), mayor tunggal open reading frame (ORF) dan 3’UTR region mengakhiri di ujung
poly-5
adenylated. Kode ORF merupakan polyprotein tunggal dan besar yang kemudian diproses menjadi 10 protein fungsional (Adam et al. 2005). Protein tersebut yaitu first protein (P1), helper component protease (HC-Pro), third protein (P3), 6K1, cylindrical inclusion protein (CI), 6K2, small nuclear inclusion protein (NIa; including the VPg and protease (NIa-Pro) domains), large nuclear inclusion protein (NIb; replicase) dan coat protein (CP).
Anggota dari genus Potyvirus ditularkan oleh kutu daun secara nonpersisten dan menginfeksi banyak spesies tanaman monokotil dan dikotil (Shukla et al. 1998) dan sebagian juga ditularkan melalui benih yang berasal dari tanaman sakit (Gibs et al. 2008). Beberapa ratus spesies kutu daun diketahui sebagai vektor dalam penyebaran Potyvirus dan kebayakan berasal dari subfamili Aphidinae diantaranya yaitu beberapa spesies dari Aphis sp, Myzus sp dan Rhopalosiphum sp (Gibs et al. 2008). Pembagian genus dalam famili tersebut berdasarkan penularan yang dilakukan oleh vektor pada virus tersebut dan karakteristik genom (Berger et al. 2005).
Fabavirus
Virus yang berasal dari genus Fabavirus (famili Comoviridae) dapat menginfeksi tanaman dalam berbagai kisaran inang, termasuk tanaman yang memiliki nilai ekonomi tinggi dan tanaman holtikultura dalam berbagai spesies. Virus ini sangat berpotensi menyebabkan kehilangan hasil yang sangat tinggi pada tanaman di dunia setiap tahunnya (Ferrer et al. 2007). Sekarang ini, terdapat tiga spesies yang telah diakui termasuk dalam genus Fabavirus yaitu Broad bean wilt virus 1 (BBWV 1), Broad bean wilt virus 2 (BBWV 2), Lamium mild mosaic virus (LaMMV) dan kandidat spesies baru, Gentian mosaic virus (GeMV) (Kobayashi 2005).
BBWV 1 dan BBWV 2 dapat dibedakan berdasarkan uji serologi (Uyemoto 1974). Pada penelitian sebelumnya, telah ditentukan bahwa seluruh sekuen nukleotida dari RNA BBWV-1 dan BBWV-2 dilaporkan memiliki hubungan genetik satu dengan lainnya (Kobayashi et al. 2003). Tanaman yang terdeteksi tunggal BBWV memiliki gejala mosaik, vein-clearing, rugosity, dan
6
malformasi pada daun (Kondo et al. 2005). BBWV memiliki kisaran inang yang luas pada tanaman dikotil dan beberapa famili tanaman monokotil (Qi et al. 2000). Patchouli mild mosaic virus (PaMMV) juga telah diklasifikasikan sebagai spesies yang termasuk dalam Fabavirus (Ferrer 2005 & Kobayashi 2003) yang telah diusulkan sebagai salah satu isolat BBWV-2 (Ikegami 1998 & 2001).
Virus yang termasuk kedalam genus Fabavirus dapat ditularkan oleh kutu daun secara nonpersisten dan dapat menyebabkan penyakit pada bagian tanaman dan buah (Kobayashi 2005). Selain itu, dapat juga ditularkan secara mekanis, namun tidak melalui benih.
Partikel dari Fabavirus berbentuk icosahedral, diameter sekitar 30 nm, dan terbagi menjadi 3 komponen yaitu bentuk T, M, dan B, selama density gradient uhra-centrifugation (DGC) (Wellink et al. 2000). Genom RNA terdiri dari dua molekul single strand dari 6.0-6.3 kb (RNA1) dan 3.9-4.5 kb (RNA2) dimana terjadi pemisahan encapsid pada komponen B dan M secara berturut-turut. Masing-masing dari 3 komponen tersebut disusun dari 2 protein yang berbeda yaitu Mr 40 x 103 sampai 45 x 103 (large coat protein; LCP) dan 21 x 103 sampai 27 x 103 (small coat protein; SCP)(Kobayashi 2005). RNA Fabavirus ditranslasikan menjadi single polyprotein awal, dimana protein fungsional diperoleh dari pemecahan proteolytic yang terjadi seperti pada Comoviridae (Wellink 2000). Seperti tampak pada Gambar 2, partikel Fabavirus berbentuk seperti bulat (isometric) (Natsuaki et al. 1994).
Gambar 2 Partikel Fabavirus berbentuk seperti bulat (isometric) (Natsuaki et al. 1994)
7
Enzyme Linked Immunosorbent Assay (ELISA)
Enzyme Linked Immunosorbent Assay (ELISA) merupakan teknik serologi canggih yang menjanjikan untuk deteksi dan identifikasi patogen tumbuhan (Seal & Elpninstone 1994, Converse & Martin 1990). Teknik ini dapat diterima secara luas oleh penggunanya, karena: (1) efisien menggunakan bahan kimia, 1,0 ml antiserum dapat digunakan untuk menguji 10-20 ribu sampel; (2) bahan kimia yang digunakan tidak berbahaya dan memiliki daya simpan lama; (3) bahan yang diuji dapat langsung berupa ekstrak tanaman sakit tanpa harus mengisolasi
patogennya terlebih dahulu; (4) mempunyai kepekaan deteksi tinggi (1-10 ng virus/ml dan 103-104 sel bakteri/ml); (5) prosedurnya relatif sederhana
dan cepat, antara 5-24 jam; (6) hasilnya dapat kuantifikasi; (7) dapat digunakan untuk menguji sampel dalam jumlah besar sekaligus; dan (8) dapat digunakan langsung di lapangan (Thomas et al. 1989, Converse & Martin 1990). Seiring dengan perkembangannya, teknik ELISA mengalami berbagai modifikasi baik dari segi praktis maupun kehandalannya, sehingga muncul berbagai variannya (Randles et al. 1996 & Seal 1997). Sebagai teknik serologi, prinsip dasar ELISA adalah reaksi antara antigen (Ag) dengan antibodi (Ab) menjadi molekul Ag-Ab yang lebih besar dan mudah mengendap. Perbedaannya, pengamatan hasil reaksi pada serologi biasa berdasarkan endapan molekul Ag-Ab, sedangkan pada ELISA berdasarkan perubahan warna yang terjadi pada substrat pereaksi sesuai dengan label atau imunoprob (immuno probe) konjugat antibodi-enzim. Perubahan warna terjadi akibat hidrolisa enzimatik pada reaksi antara konjugat antibodi-enzim dengan substratnya, sehingga hasil ELISA lebih peka dan dapat dikuantifikasi (Converse & Martin1990). Tahapan umum ELISA meliputi penempelan (trapping) Ag atau Ab pada media reaksi (solid phase), seperti cawan ELISA, diikuti penambahan konjugat, dan diakhiri dengan penambahan substrat serta bufer penghenti reaksi (blocking buffer).
Dalam perkembangannya, metode ini mengalami modifikasi dalam prosedur pelaksanaan pengujian, diantaranya adalah pengujian standar (direct) Double Antibody Sandwich-Enzyme Link Immunosorbent Assay (DAS ELISA) dan Indirect-Enzyme Link Immunosorbent Assay (I-ELISA). Perbedaan kedua metode ini adalah pada tempat enzim terikat. Bila konjugasi enzim dilakukan pada
8
imunoglobulin antivirus maka metode itu termasuk DAS-ELISA, tetapi bila konjugasi enzim dilakukan pada imunoglobulin dari serum darah hewan maka metode tersebut diklasifikasikan sebagai I-ELISA (Badan Karantina Tanaman 2009).
Reverse Transcription-Polymerase Chain Reaction(RT-PCR)
Sebagian besar komponen genetik virus tanaman adalah RNA, terutama jenis-jenis viroid yang hanya terdiri dari RNA. Untuk melipat gandakan DNA dari cetakan yang berupa RNA maka sebelumnya perlu dilakukan sebuah tahapan yaitu transkripsi balik (reverse transcription). Pada tahap ini cetakan RNA terlebih dahulu diubah menjadi cDNA (complementary DNA) menggunakan enzim reverse transcriptase.
Metode Polymerase Chain Reaction (PCR) merupakan metode deteksi yang sensitif dan cepat dalam mendeteksi virus. PCR merupakan sebuah metode yang digunakan untuk memperbanyak suatu fragmen DNA yang spesifik secara invitro. Posisi fragmen DNA yang spesifik tersebut ditentukan oleh sepasang primer yang akan menjadi cetakan awal untuk proses perbanyakan fragmen DNA selanjutnya dengan bantuan enzim polimerase dan deoxyribonucleotide triposphate (dNTPs) yang dikondisikan pada suhu tertentu. Fragmen DNA, yang pada awalnya terdapat dalam konsentrasi yang sangat rendah akan diperbanyak menjadi cetakan fragmen DNA baru yang cukup untuk dapat divisualisasi pada gel agarosa (Badan Karantina Tanaman 2009).
Secara prinsip, PCR merupakan proses yang diulang-ulang antara 20–30 kali. Setiap siklus terdiri dari tiga tahap. Berikut adalah tiga tahap kerja PCR dalam satu siklus:
1. Tahap peleburan (melting) atau denaturasi. Pada tahap ini (berlangsung pada suhu tinggi, 94–96 °C) ikatan hidrogen DNA terputus (denaturasi) dan DNA menjadi berutas tunggal. Biasanya pada tahap awal PCR tahap ini dilakukan agak lama (sampai 5 menit) untuk memastikan semua berkas DNA terpisah. Pemisahan ini DNA tidak stabil dan siap menjadi template bagi primer. Durasi tahap ini 1–2 menit.
9
2. Tahap penempelan atau annealing. Primer menempel pada bagian DNA template yang komplementer urutan basanya. Ini dilakukan pada suhu antara 45–60 °C. Penempelan ini bersifat spesifik. Suhu yang tidak tepat menyebabkan tidak terjadinya penempelan atau primer menempel di sembarang tempat. Durasi tahap ini 1–2 menit.
3. Tahap pemanjangan atau elongasi. Suhu untuk proses ini tergantung dari jenis DNA polymerase yang dipakai. Dengan Taq-polymerase, proses ini biasanya dilakukan pada suhu 76 °C. Durasi tahap ini biasanya 1 menit.
BAHAN DAN METODE
Tempat dan Waktu Penelitian
Survei penyakit mosaik dan koleksi sampel tanaman nilam sakit dilakukan di Kebun Percobaan Balai Tanaman Obat dan Aromatik (BALITTRO) di daerah Gunung Bunder dan Cicurug, Bogor, Jawa Barat. Deteksi virus dilakukan di Laboratorium Virologi, Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor. Penelitian dilakukan dari bulan Maret sampai November 2011.
Metode Penelitian
Pengambilan Sampel Tanaman Nilam Bergejala Mosaik di Lapangan
Sampel tanaman nilam yang bergejala mosaik diperoleh dari Kebun Percobaan Balai Penelitian Tanaman Obat dan Aromatik (BALITTRO) di daerah Gunung Bunder dan Cicurug, Bogor, Jawa Barat. Sampel daun yang diambil dari lapangan dideteksi di laboratorium.
Deteksi Potyvirus melalui I-ELISA
Teknik deteksi diawali dengan menggerus sampel tanaman nilam sebanyak 0,1 g dalam buffer coating pH 9,6 dengan perbandingan 1:5 (b/v). Sebanyak 100 µl sampel tanaman kemudian dimasukkan ke dalam masing-masing sumuran plat mikrotiter ELISA dan diinkubasi semalam pada suhu 4 °C. Cairan sampel tanaman dalam plat mikrotiter dibuang kemudian plat di cuci dengan phosphate buffer saline tween-20 (PBST) sebanyak dua kali kemudian masing-masing sumuran diisi dengan 100 µl skim milk 2%. Plat diinkubasi selama 30 menit pada suhu 37 °C. Setelah itu buang dan keringkan plat mikrotiter kemudian masing-masing sumuran diisi dengan antiserum Potyvirus sebanyak 100 µl dan diinkubasikan selama 2-4 jam pada suhu 37 °C. Antiserum tersebut telah diencerkan terlebih dahulu menggunakan buffer conjugate dengan perbandingan 1:1000. Plat mikrotiter kemudian dicuci dengan PBST sebanyak tiga kali dan kemudian diisi dengan 100 µl konjugat yang dilarutkan dalam conjugate buffer dengan perbandingan 1:1000. Plat mikrotiter kemudian diinkubasi selama 2 jam pada suhu 37 °C. Setelah inkubasi selama 2 jam plat
11
mikrotiter dicuci menggunakan PBST sebanyak tiga kali. Masing-masing sumuran kemudian diisi dengan 100 µl PNP yang telah dilarutkan dalam substrate buffer dan diinkubasi pada suhu ruang dan di tempat gelap. Plat mikrotiter kemudian diuji secara kuantitatif menggunakan ELISA reader (Bio-RAD 550) pada panjang gelombang 405 nm setiap 30 menit selama 60 menit. Dalam setiap pengujian disertakan kontrol negatif yaitu tanaman sehat dan bufer. Pengujian dikatakan positif jika nilai absorban sampel yang diuji 2 kali lebih besar daripada kontrol negatif tanaman sehat.
Deteksi Fabavirus melalui DAS-ELISA
Teknik deteksi diawali dengan mengencerkan antiserum Fabavirus pada coating buffer dengan perbandingan 1:500. Sebanyak 100 µl antiserum dimasukkan kedalam masing-masing sumuran pada plat mikrotiter dan diinkubasikan selama 2-4 jam pada suhu 37 °C. Plat mikrotiter kemudian dicuci menggunakan PBST sebanyak 2 kali. Sampel tanaman sebanyak 0,1 g digerus menggunakan extract buffer dengan perbandingan 1:5 (b/v) kemudian 100 µl dimasukan ke dalam masing-masing sumuran dan diinkubasi pada suhu 4 °C selama semalam. Plat mikrotiter kemudian dicuci menggunakan PBST sebanyak 3 kali. Pada masing-masing sumuran dimasukkan 100 µl konjugat yang telah diencerkan pada conjugate buffer dengan perbandingan 1:500 dan diinkubasikan selama 4 jam pada suhu 37 °C. Masing-masing sumuran dicuci kembali menggunakan PBST sebanyak 3 kali dan kemudian ditambahkan 100 µl PNP yang telah dilarutkan dalam substrate buffer dan diinkubasi pada suhu ruang dan di tempat gelap. Plat mikrotiter kemudian diuji secara kuantitatif menggunakan ELISA reader (Bio-RAD 550) pada panjang gelombang 405 nm setiap 30 menit selama 60 menit. Dalam setiap pengujian disertakan kontrol negatif yaitu tanaman sehat dan bufer. Pengujian dikatakan positif jika nilai absorban sampel yang diuji 2 kali lebih besar daripada kontrol negatif tanaman sehat.
Deteksi Diferensial Potyvirus dan Fabavirus Melalui RT-PCR
Untuk dapat membedakan Potyvirus dan Fabavirus yang menginfeksi tanaman nilam, dilakukan deteksi virus melalui metode RT-PCR dan
12
menggunakan primer khusus yang dapat digunakan dalam RT-PCR yang dapat mengamplifikasi virus secara terpisah.
Ekstraksi RNA total. RNA total diekstraksi dari jaringan daun tanaman nilam bergejala penyakit mosaik dengan menggunakan Bench-Top Protocols for Xprep Plant RNA Mini Kit (PKT Korea). Tanaman yang diekstraksi merupakan tanaman yang telah diuji menggunakan ELISA dan positif Potyvirus dan Fabavirus. Tahapannya adalah sebanyak 0,1 g sampel daun digerus dengan menggunakan mortar dan pistil steril dengan bantuan nitrogen cair. Hasil gerusan dimasukkan ke dalam tabung mikro 2 ml dan ditambahkan 450 µl bufer XPRB yang mengandung 1% mercaptoethanol, kemudian dihomogenkan. Sampel dipipet, lalu dimasukkan ke dalam filter coloumn putih dan ditempatkan pada tabung koleksi 2 ml, lalu disentrifuse pada kecepatan 12000 rpm selama 2 menit. Supernatan dipipet tanpa menyentuh pelet dalam tabung koleksi, ukur volume supernatan yang diperoleh lalu dipindahkan ke dalam tabung mikro 2 ml baru. Kemudian ditambahkan ethanol 96% (setengah dari volume supernatan) dan dicampur dengan rata. Sampel dimasukkan ke dalam XPPLR mini coloumn merah, kemudian ditempatkan pada tabung koleksi 2 ml lalu disentrifuse pada kecepatan 12000 rpm selama 1 menit. Cairan yang terdapat pada tabung koleksi dibuang, kemudian ditambahkan 500 µl wash buffer 1 ke dalam XPPLR mini coloumn, lalu ditutup dengan baik dan disentrifuse pada kecepatan 12000 rpm selama 1 menit. Cairan yang terdapat pada tabung koleksi dibuang, kemudian ditambahkan 700 µl wash buffer 2 ke dalam XPPLR mini coloumn, lalu ditutup dengan baik dan disentrifuse pada kecepatan 12000 rpm selama 1 menit. Cairan yang terdapat pada tabung koleksi dibuang, kemudian untuk mengeringkan XPPLR mini coloumn disentrifuse selama 3 menit pada 12000 rpm. Untuk meyakinkan bahwa coloumn telah kering, coloumn dipindahkan pada tabung koleksi baru. Selanjutnya, 50 µl RNAse free water ditambahkan ke dalam pusat membran XPPLR mini coloumn, didiamkan 1 menit lalu disentrifuse pada kecepatan 12000 rpm selama 2 menit. Siapan RNA total ini digunakan sebagai template dalam reaksi RT-PCR.
Sintesis cDNA. RNA hasil ekstraksi selanjutnya ditranskripsi balik menjadi cDNA (complementary DNA) dengan menggunakan teknik Reverse Transcription
13
(RT). Reaksi RT dibuat dengan total volume 10 µl yang mengandung 2 µl RNA total, 2 µl bufer RT 10X, 0,35 µl 50 mM DTT (dithiothreitol), 2 µl 10 mM dNTP (deoksiribonukleotida triphosphat), 0,35 µl M-MuLV Rev, 0,35 µl RNase inhibitor, 0,75 µl oligo (dT), dan 2,2 µl H2O. Komponen-komponen tersebut
digunakan untuk satu kali reaksi RT. Reaksi RT dilakukan dalam sebuah Automated Thermal cycler (Gene Amp PCR System 9700; PE Applied Biosystem, USA) yang diprogram untuk satu siklus pada suhu 25 ºC selama 5 menit, 42 ºC selama 60 menit, dan 70 ºC selama 15 menit. cDNA hasil RT digunakan sebagai DNA template dalam reaksi PCR.
Amplifikasi DNA dengan PCR. Amplifikasi DNA virus dilakukan dengan metode PCR dengan menggunakan pasangan primer yang telah didesain khusus untuk mengamplifikasi virus secara terpisah. Pasangan primer yang spesifik digunakan untuk mendeteksi Potyvirus yaitu CPUP(F) (5’-TGAGGATCCTGGTGYATHGARAAYGG-3’, Y = C/T, H = A/T/C, R = A/G), spesifik untuk coat protein pada Potyvirus dan CP9502(R) (5’-GCGGATCCTTTTTTTTTTTTTTTTT-3’) spesifik untuk ujung 3’ genom Potyvirus dengan prediksi ukuran produk 800 bp (Singh et al. 2009). Sedangkan pasangan primer yang spesifik digunakan untuk mendeteksi Fabavirus yaitu [BBWVVSSP (5’- GTBTCDAGTGCTYTDGAAGG-3’, B = C, G, atau T; D = A, G, atau T; Y = C atau T) dan BBWVKMRM (5’-TDGWDCCATCVAGICK CATTTT-3’, W = A atau T; V = A, C, atau G; I = Inosine; K = G atau T)] dengan prediksi ukuran produk 322 bp mencakup wilayah dari C-terminal dari large coat protein (LCP) ke N-terminal small coat protein (SCP) (Kondo et al. 2005).
Metode yang digunakan dalam penelitian ini untuk mengamplifikasi DNA Potyvirus, Fabavirus, dan keduanya yaitu digunakan pasangan primer Potyvirus, pasangan primer Fabavirus, dan pasangan primer Potyvirus dan Fabavirus yang dicampur untuk mengamplifikasi DNA Potyvirus, Fabavirus, dan campuran kedua DNA tersebut. Reaksi PCR dengan total volume 25 µl, terdiri atas 1 µl masing-masing primer, 12,5 µl Go Tag Green Master Mix 2x (Promega, Madison, USA), 9,5 µl H2O, dan 1 µl DNA template. Amplifikasi ini dilakukan pada
Automated Thermal cycler (Gene Amp PCR System 9700; PE Applied Biosystem, USA). Untuk Potyvirus amplifikasi ini didahului dengan denaturasi
14
awal pada 94 ºC selama 5 menit. Kemudian dilanjutkan dengan 45 siklus yang terdiri dari denaturasi pada 94 ºC selama 1 menit, penempelan primer (annealing) pada 54 ºC selama 2 menit, dan pemanjangan pada 72 ºC selama 1 menit. Khusus untuk siklus terakhir, ditambahkan 10 menit pada 72 ºC untuk tahapan sintesis, dan siklus berakhir pada suhu 4 ºC. Untuk Fabavirus amplifikasi ini didahului dengan denaturasi awal pada 95 ºC selama 5 menit. Kemudian dilanjutkan dengan 35 siklus yang terdiri dari denaturasi pada 95 ºC selama 1 menit, penempelan primer (annealing) pada 51 ºC selama 1 menit, dan pemanjangan pada 72 ºC selama 1 menit. Khusus untuk siklus terakhir, ditambahkan 5 menit pada 72 ºC untuk tahapan sintesis, dan siklus berakhir pada suhu 4 ºC. Untuk pengujian yang dilakukan bersamaan menggunakan primer Potyvirus dan Fabavirus (Mix) amplifikasi ini didahului dengan denaturasi awal pada 95 ºC selama 5 menit. Kemudian dilanjutkan dengan 10 siklus yang terdiri dari denaturasi pada 95 ºC selama 1 menit, penempelan primer (annealing) pada 51 ºC selama 1 menit, dan pemanjangan pada 72 ºC selama 1 menit, kemudian dilanjutkan dengan 30 siklus yang terdiri dari denaturasi pada 94 ºC selama 1 menit, penempelan primer (annealing) pada 54 ºC selama 1 menit, dan pemanjangan pada 72 ºC selama 2 menit. Khusus untuk siklus terakhir, ditambahkan 10 menit pada 72 ºC untuk tahapan sintesis, dan siklus berakhir pada suhu 4 ºC. Setelah dilakukan PCR, maka hasil yang diperoleh dapat dielektroforesis.
Elektroforesis. Agarose sebanyak 0,3 gr dimasukkan ke dalam tabung Erlenmeyer 100 ml, lalu ditambahkan 30 ml bufer Tris-Borate EDTA (TBE) 0,5x (0,045 M Tris-Borate, 0,01 M EDTA), untuk pembuatan gel agarose. Kemudian dipanaskan dalam microwave selama 2 menit atau sampai agarose larut. Larutan agar didinginkan terlebih dahulu selama kurang lebih 15 menit, kemudian ditambahkan 1,5 µl ethidium bromida kemudian diaduk. Cetakan gel telah disiapkan terlebih dahulu dan ‘sisir’ gel diletakkan di bagian atas pencetak gel kemudian larutan agarose dituang ke dalam cetakan. Gel didiamkan sampai mengeras (30-45 menit). Setelah mengeras, gel diambil dan diletakkan ke dalam bak elektroforesis yang berisi buffer TBE 0,5 kali. DNA hasil PCR dimasukkan ke dalam sumur gel elektroforesis sebanyak 5 µl dan 100 bp DNA ladder dimasukkan pada sumuran gel elektroforesis yang berada di posisi sebelah kiri
15
sebanyak 5 µl. Elektroforesis dilakukan dengan tegangan 100 volt selama 25 menit. Hasil elektroforesis divisualisasikan dengan transluminator ultraviolet. Pita DNA yang terbentuk pada hasil elektroforesis tersebut difoto dengan menggunakan kamera digital.
PCR dilakukan berkali-kali untuk melihat validitas pasangan primer Potyvirus, pasangan primer Fabavirus, dan pasangan primer keduanya.
Tabel 1 Reagensia PCR dan konsentrasi yang diperlukan untuk mendeteksi validitas pasangan primer Potyvirus yang digunakan secara terpisah terhadap 3 template cDNA yang berbeda (Potyvirus, Fabavirus dan keduanya).
Komponen Volume(µl)1 Volume(µl)2
H2O 9,5 28,5
Gotag Green Master Mix 2x 12,5 37,5
Primer CPUP-F 1 3
Primer CP9502-R 1 3
cDNA 1 3
Total Volume (µl) 25 75
1)Volume total yang diperlukan sebanyak 25 µl untuk 1X reaksi; 2) Volume total yang diperlukan
sebanyak 75 µl untuk 3X reaksi
Tabel 2 Reagensia PCR dan konsentrasi yang diperlukan untuk mendeteksi validitas pasangan primer Fabavirus yang digunakan secara terpisah terhadap 3 template cDNA yang berbeda (Potyvirus, Fabavirus dan keduanya).
Komponen Volume(µl)1 Volume(µl)2
H2O 9,5 28,5
Gotag Green Master Mix 2x 12,5 37,5
Primer BBWVVSSP 1 3
Primer BBWVKMRM 1 3
cDNA 1 3
Total Volume (µl) 25 75
1)Volume total yang diperlukan sebanyak 25 µl untuk 1X reaksi; 2) Volume total yang diperlukan
16
Tabel 3 Reagensia PCR dan konsentrasi yang diperlukan untuk mendeteksi validitas pasangan primer Potyvirus dan Fabavirus yang digunakan secara bersamaan terhadap 3 template cDNA yang berbeda (Potyvirus, Fabavirus dan keduanya).
Komponen Volume(µl)1 Volume(µl)2
H2O 7,5 22,5
Gotag Green Master Mix 2x 12,5 37,5
Primer CPUP-F 1 3 Primer CP9502-R 1 3 Primer BBWVVSSP 1 3 Primer BBWVKMRM 1 3 cDNA 1 3 Total Volume (µl) 25 75
1)Volume total yang diperlukan sebanyak 25 µl untuk 1X reaksi; 2) Volume total yang diperlukan
HASIL DAN PEMBAHASAN
Metode deteksi yang dilakukan untuk mengetahui keberadaan Potyvirus dan Fabavirus di pertanaman nilam yaitu dengan DAS-ELISA untuk mendeteksi Fabavirus, I-ELISA untuk mendeteksi Potyvirus dan RT-PCR untuk mendeteksi keduanya secara molekuler. Sampel tanaman yang diambil dari areal pertanaman nilam di daerah Bogor yaitu di Gunung Bunder dan di Cicurug memiliki gejala mosaik. Berdasarkan hasil ELISA pada sampel di lapangan, diketahui bahwa tanaman nilam positif terserang Potyvirus, Fabavirus dan keduanya. Tanaman yang positif terserang berdasarkan uji ELISA kemudian diekstraksi dan dilakukan uji molekuler menggunakan RT-PCR.
Penyakit mosaik tanaman nilam yang disebabkan oleh Potyvirus dan Fabavirus memiliki gejala yang tidak dapat dibedakan di lapangan (Gambar 3). Oleh karena itu deteksi dilakukan dengan menggunakan pasangan primer yang telah didesain khusus.
N
Gambar 3 Gejala mosaik pada daun nilam yang disebabkan oleh Potyvirus dan atau Fabavirus
Deteksi dengan RT-PCR memerlukan sepasang primer yang didesain khusus untuk mendeteksi virus secara terpisah. Primer-primer yang digunakan dalam metode ini yaitu CPUP(F)(5’-TGAGGATCCTGGTGYATHGARAAYGG-3’, Y = C/T, H = A/T/C, R = A/G), spesifik untuk coat protein pada Potyvirus dan CP9502(R)(5’-GCGGATCCTTTTTTTTTTTTTTTTT-3’) spesifik untuk ujung 3’ genom Potyvirus dengan prediksi ukuran produk 800 bp. Sedangkan pasangan primer yang spesifik digunakan untuk mendeteksi Fabavirus yaitu [BBWVVSSP
18
(5’- GTBTCDAGTGCTYTDGAAGG-3’, B = C, G, atau T; D = A, G, atau T; Y = C atau T) dan BBWVKMRM (5’-TDGWDCCATCVAGICKCATTTT-3’, W = A atau T; V = A, C, atau G; I = Inosine; K = G atau T)] dengan prediksi ukuran produk 322 bp mencakup wilayah dari C-terminal dari large coat protein (LCP) ke N-terminal small coat protein (SCP).
Validasi Pasangan Primer untuk Deteksi Potyvirus
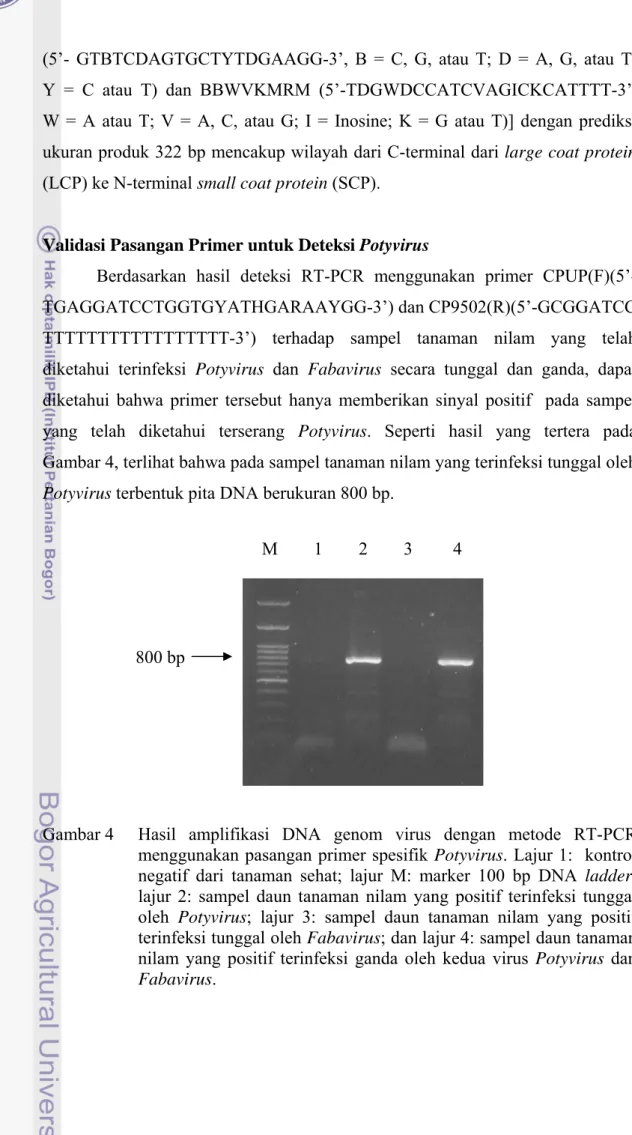
Berdasarkan hasil deteksi RT-PCR menggunakan primer CPUP(F)(5’-TGAGGATCCTGGTGYATHGARAAYGG-3’) dan CP9502(R)(5’-GCGGATCC TTTTTTTTTTTTTTTTT-3’) terhadap sampel tanaman nilam yang telah diketahui terinfeksi Potyvirus dan Fabavirus secara tunggal dan ganda, dapat diketahui bahwa primer tersebut hanya memberikan sinyal positif pada sampel yang telah diketahui terserang Potyvirus. Seperti hasil yang tertera pada Gambar 4, terlihat bahwa pada sampel tanaman nilam yang terinfeksi tunggal oleh Potyvirus terbentuk pita DNA berukuran 800 bp.
Gambar 4 Hasil amplifikasi DNA genom virus dengan metode RT-PCR menggunakan pasangan primer spesifik Potyvirus. Lajur 1: kontrol negatif dari tanaman sehat; lajur M: marker 100 bp DNA ladder; lajur 2: sampel daun tanaman nilam yang positif terinfeksi tunggal oleh Potyvirus; lajur 3: sampel daun tanaman nilam yang positif terinfeksi tunggal oleh Fabavirus; dan lajur 4: sampel daun tanaman nilam yang positif terinfeksi ganda oleh kedua virus Potyvirus dan Fabavirus.
800 bp
19
Pada sampel tanaman yang terinfeksi tunggal oleh Fabavirus tidak muncul pita karena Fabavirus tidak dapat dideteksi oleh primer Potyvirus. Pada sampel tanaman yang terinfeksi ganda oleh Potyvirus dan Fabavirus terlihat hanya satu pita DNA saja, yaitu pita DNA berukuran 800 bp saja yang merupakan pita DNA Potyvirus dan tidak terdapat pita yang berukuran 322 bp yang merupakan pita DNA Fabavirus. Demikian juga pada sampel tanaman yang sehat tidak terdapat pita DNA. Hasil ini menunjukkan kespesifikan pasangan primer CPUP(F) dan CP9502(R) untuk mendeteksi keberadaan Potyvirus.
Validasi Pasangan Primer untuk DeteksiFabavirus
Berdasarkan hasil deteksi RT-PCR menggunakan primer BBWVVSSP (5’- GTBTCDAGTGCTYTDGAAGG-3’, B = C, G, atau T; D = A, G, atau T; Y = C atau T) dan BBWVKMRM (5’-TDGWDCCATCVAGICKCATTTT-3’, W = A atau T; V = A, C, atau G; I = Inosine; K = G atau T) terhadap sampel tanaman nilam yang telah diketahui terinfeksi Potyvirus dan Fabavirus secara tunggal dan ganda, dapat diketahui bahwa primer tersebut hanya memberikan sinyal positif pada sampel yang telah diketahui terserang Fabavirus. Seperti hasil yang tertera pada Gambar 5, terlihat bahwa pada sampel tanaman nilam yang terinfeksi tunggal oleh Fabavirus terbentuk pita DNA berukuran 322 bp.
Gambar 5 Hasil amplifikasi DNA genom virus dengan metode RT-PCR menggunakan pasangan primer spesifik Fabavirus. Lajur 1: kontrol negatif dari tanaman sehat; lajur M: marker 100 bp DNA ladder; lajur 2: sampel daun tanaman nilam yang positif terinfeksi tunggal oleh Potyvirus; lajur 3: sampel daun tanaman nilam yang positif terinfeksi tunggal oleh Fabavirus; dan lajur 4: sampel daun tanaman nilam yang positif terinfeksi ganda oleh kedua virus Potyvirus dan Fabavirus.
322 bp
20
Pada sampel tanaman yang terinfeksi tunggal oleh Potyvirus tidak muncul pita karena Potyvirus tidak dapat dideteksi oleh primer Fabavirus. Pada sampel tanaman yang terinfeksi ganda oleh Potyvirus dan Fabavirus terlihat hanya satu pita DNA saja, yaitu pita DNA berukuran 322 bp saja yang merupakan pita DNA Fabavirus dan tidak terdapat pita yang berukuran 800 bp yang merupakan pita DNA Potyvirus. Demikian juga pada sampel tanaman yang sehat tidak terdapat pita DNA. Hasil ini menunjukkan kespesifikan pasangan primer BBWVVSSP dan BBWVKMRM untuk mendeteksi Fabavirus.
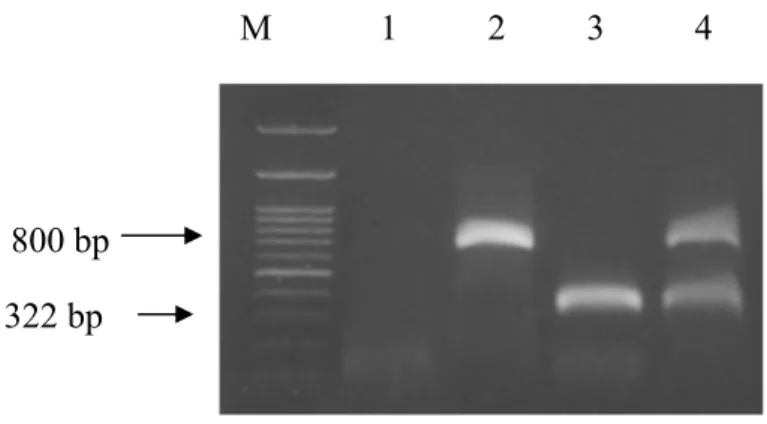
Validasi Pasangan Primer untuk Deteksi Simultan Potyvirus dan Fabavirus Metode RT-PCR dengan menggunakan primer Potyvirus dan Fabavirus telah terbukti dapat mendeteksi kedua virus secara terpisah. Untuk mendeteksi secara simultan Potyvirus dan Fabavirus pada tanaman nilam yang terinfeksi ganda digunakan metode yang berbeda dimana kedua pasangan primer Potyvirus dan Fabavirus digunakan secara bersamaan tercampur dengan komponen PCR yang lain. Hasil yang diperoleh jika pasangan primer tersebut digunakan secara bersamaan terlihat pada Gambar 6.
Seperti terlihat pada gambar tersebut, pada lajur 1 tidak terdapat pita DNA yang muncul karena sampel merupakan kontrol tanaman sehat. Pada lajur 2 yang merupakan sampel positif terinfeksi tunggal oleh Potyvirus terlihat hanya pita DNA Potyvirus berukuran 800 bp. Hal ini disebabkan pasangan primer Potyvirus hanya mendeteksi virus yang spesifik yaitu Potyvirus saja, sedangkan Fabavirus tidak terdeteksi. Begitu pula jika sampel yang digunakan merupakan sampel yang terinfeksi tunggal oleh Fabavirus seperti ditunjukkan pada lajur 3 maka pita DNA yang muncul yaitu pita DNA Fabavirus dengan panjang 322 bp.
21
Gambar 6 Hasil amplifikasi DNA genom virus dengan metode RT-PCR menggunakan campuran primer Potyvirus dan Fabavirus. Lajur 1: kontrol negatif dari tanaman sehat; lajur M: marker 100 bp DNA ladder; lajur 2: sampel daun tanaman nilam yang positif terinfeksi tunggal oleh Potyvirus; lajur 3: sampel daun tanaman nilam yang positif terinfeksi tunggal oleh Fabavirus; dan lajur 4: sampel daun tanaman nilam yang positif terinfeksi ganda oleh kedua virus Potyvirus dan Fabavirus.
Pada lajur 4, yang merupakan sampel tanaman yang terinfeksi ganda oleh Potyvirus dan Fabavirus, terdapat 2 pita DNA yang muncul karena kedua virus tersebut terdeteksi oleh kedua pasangan primer Potyvirus dan Fabavirus sehingga kedua pasangan primer akan menempel pada pasangan DNA masing-masing. Pasangan primer Potyvirus akan menempel pada DNA Potyvirus dan pasangan primer Fabavirus akan menempel pada DNA Fabavirus. Meskipun terdapat pada satu lajur, kedua pita tersebut dapat terlihat jelas karena perbedaaan ukurannya. Pita DNA Potyvirus berada di bagian atas dengan panjang 800 bp sedangkan pita DNA Fabavirus berada di bagian bawah dengan panjang 322 bp.
Dengan metode pencampuran kedua primer ini maka dapat diketahui bahwa kedua primer Potyvirus dan Fabavirus dapat digunakan untuk mendeteksi kedua virus ini, baik yang terinfeksi tunggal maupun yang terinfeksi ganda. Selain untuk mendeteksi virus, metode RT-PCR dengan kedua pasang primer ini juga dapat diterapkan untuk diagnostik sampel dari lapangan.
Penerapan Metode RT-PCR untuk Deteksi Simultan Potyvirus dan Fabavirus pada Sampel dari Lapangan
Hasil deteksi dengan menggunakan RT-PCR untuk sampel yang berasal dari lapangan dapat dilihat pada Gambar 7. Pada lajur 1,2 dan 3 berfungsi sebagai
800 bp
M 1 2 3 4
22
kontrol, yaitu untuk lajur 1 merupakan sampel tanaman sehat, lajur 2 merupakan sampel tanaman yang positif terinfeksi tunggal Potyvirus dan lajur 3 oleh Fabavirus. Lajur 4 sampai lajur 16 merupakan sampel dari lapangan, yaitu dari Gunung Bunder dan Cicurug, keduanya dari wilayah Bogor.
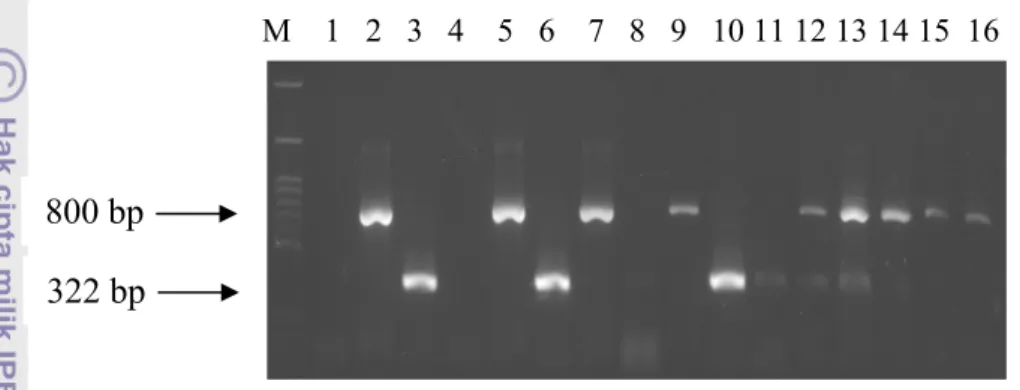
Gambar 7 Hasil amplifikasi DNA genom virus dengan metode RT-PCR menggunakan campuran pasangan primer Potyvirus dan Fabavirus terhadap sampel dari lapangan. Daun tanaman nilam yang positif sehat (lajur 1), sampel yang positif terinfeksi tunggal oleh Potyvirus (lajur 2), sampel yang positif terinfeksi tunggal oleh Fabavirus (lajur 3), sampel yang berasal dari Cicurug (lajur 4, 7, 11, 15), Gunung Bunder (lajur 5, 6, 8, 9, 10, 12, 13, 14, 16), dan marker 100 bp DNA ladder (lajur M).
Berdasarkan RT-PCR yang dilakukan dengan menggunakan pasangan primer Potyvirus dan Fabavirus yang dicampur dapat diketahui bahwa sampel yang positif terinfeksi oleh Potyvirus ditandai adanya pita DNA dengan panjang 800 bp dan yang terinfeksi oleh Fabavirus ditandai oleh adanya pita DNA 322 bp. Terlihat juga bahwa dua sampel lajur 4 yang berasal dari Cicurug dan lajur 8 yang berasal dari Gunung Bunder tidak terdeteksi terinfeksi Potyvirus maupun Fabavirus. Namun sampel tanaman lainnya yang diuji menunjukkan hasil yang positif. Ada yang terinfeksi tunggal Potyvirus seperti pada lajur 5,9,14 dan 16 yang berasal dari sampel Gunung Bunder dan lajur 7 dan 15 yang berasal dari sampel Cicurug. Sampel yang positif terinfeksi Fabavirus dapat dilihat pada lajur 6 dan 10 yang berasal dari Gunung Bunder dan lajur 11 yang berasal dari Cicurug. Selain itu, dapat diketahui pula sampel yang terinfeksi ganda oleh Potyvirus dan
800 bp 322 bp
23
Fabavirus seperti yang ditunjukkan pada lajur 12 dan 13 yang keduanya merupakan sampel dari Gunung Bunder.
Berdasarkan gejalanya pada tanaman nilam, Potyvirus dan Fabavirus tidak dapat dibedakan. Namun dengan metode RT-PCR, infeksi Potyvirus dan Fabavirus pada tanaman nilam dapat dibedakan dari besarnya ukuran panjang pita DNA yang terbentuk setelah dilakukan amplifikasi.
KESIMPULAN DAN SARAN
Kesimpulan
Metode RT-PCR dengan pasangan primer CPUP-F dan CP9502-R yang spesifik untuk Potyvirus dan BBWVVSSP dan BBWVKMRM yang spesifik untuk Fabavirus dapat digunakan untuk mendeteksi virus-virus tersebut secara simultan pada tanaman nilam baik yang terinfeksi tunggal maupun ganda.
Saran
Perlu pengujian dalam skala yang lebih luas untuk lebih meyakinkan keefektifan metode ini dalam mendeteksi kedua virus tersebut terhadap sampel dari lapangan.
DAFTAR PUSTAKA
Adams MJ, Antoniw JF, Beaudoin F. 2005. Overview and analysis of the polyprotein cleavage sites in the family Potyviridae. Mol Plant Pathol 6: 471–487.
Akin HM dan Sudarsono. 2002. Variasi genetika galur Peanut stripe potyvirus dan hubungan evolusinya dengan subkelompok Bean common mosaic virus. Hayati 9(1): 5-9.
Balai Besar Pelatihan Pertanian. 2011. Nilam, primadona tanaman aromatik Indonesia. Lembang. www.bbpp-lembang.deptan.go.id. [11 November 2011].
Balai Penelitian Tanaman Rempah dan Obat. 1991. Perkembangan dan permasalahan usaha tani nilam dan tanaman atsiri lain di Aceh. Di dalam: Prosiding Forum Komunikasi Ilmiah Pengembangan Atsiri di Sumatera, Bukittinggi, 31 Agustus 1991. Balai Penelitian Tanaman Rempah dan Obat, Bogor. Hlm 36−37.
Berger PH, et al. 2005. Potyviridae. Di dalam: Fauquet CM, et al. (eds). Virus Taxonomy. VIIIth Report of The International Committee on Taxonomy of Viruses. Elsevier. London: Academic Press.
Converse RH, Martin RR. 1990. ELISA methods for plant viruses. Di dalam: Hampton, R., E. Ball, and S. De Boer (eds). Serological Methods for Detection and Identification of Viral and Bacterial Plant Patogens. St Paul: APS Press. 179-196.
Ditjen Perkebunan. 2006. Nilam. Statistik Perkebunan Indonesia.
[Deptan] Badan Karantina Tumbuhan. 2009. Pedoman diagnosis OPTK golongan virus. Deptan: Jakarta.
Ferrer RM, et al. 2005. The complete sequence of a Spanish isolate of Broad bean wilt virus 1 (BBWV-1) reveals a high variability and conserved motifs in the genus Fabavirus. Arch Virol 150: 2109–2116.
Ferrer RM, et al. 2007. Detection and identification of species of the genus Fabavirus by RT–PCR with a single pair of primers. J Virol Methods 144: 156–160.
Gibbs A J, Trueman W H 2008. The Bean common mosaic virus lineage of potyviruses: where did it arise and when. Arch Virol 153: 2177–2187.
Hartono S, et al. 2006. Pemurnian dan deteksi serologi Patchouli mottle virus pada tanaman nilam. Jurnal Perlindungan Tanaman Indonesia 12(2)74-82. Ikegami M, Kawashima H, Natsuaki T, Sugimura N. 1998. Complete nucleotide
sequence of the genome organization of RNA2 of Patchouli mild mosaic virus, a new Fabavirus. Springer 2431–2434.
26
Ikegami M, Onobori Y, Sugimura N, Natsuaki T. 2001. Complete nucleotide sequence and the genome organization of Patchouli mild mosaic virus RNA1. Springer 355–358.
Kobayashi YO, et al. 2003. Analysis of genetic relations between Broad bean wilt virus 1 and Broad bean wilt virus 2. J Gen Plant Pathol 69: 320–326. Kobayashi YO, et al. 2005. Gentian mosaic virus: a new species in the genus
Fabavirus. Phytopathology 95: 192–197.
Kondo T, et al. 2005. Broad bean wilt virus 2 in yams. J Gen Plant Pathol(71): 441-443.
Mustofa A. 1991. Pola agroindustri atsiri di pedesaan. Di dalam: Prosiding Forum Komunikasi Ilmiah Tanaman Atsiri di Sumatera. Bukittinggi, 31 Agustus 1991. Balai Penelitian Tanaman Rempah dan Obat, Bogor.
Natsuaki KT, et al. 1994. Characterization of two viruses isolatet from patchouli in Japan. Plant Dist. 78: 1097-1097.
Noveriza R, Suastika G, Hidayat S.H, Kartosuwondo U. 2010. Identification of a Potyvirus Associated with Mosaic Disease on Patchouli Plant in Indonesia (Abstrak). ISSAAS International Congress 2010’Agricultural Adaption in Response to Climate Change’; Denpasar, 14-18 Nov 2010: 227-273 (17). Qi Y, Zhou X, Li D. 2000. Complete nucleotide sequence and infectious cDNA
clone of the RNA1 of a Chinese isolate of Broad bean wilt virus 2. Virus Genes 20: 201–207.
Randles JW, Hodgson RAJ, Weffels E. 1996. The rapid and sensitive detection of plant patogens by molecular methods. Australasian Plant Pathol. 25:71-85.
Seal S, Elphinstone J. 1994. Advances in identifycation and detection of P. solanacearum. Di dalam: Hayward, A.C. and G.L. Hartman (eds). The Disease and Its Causative Agent, P. solanacearum. CAB International, Wallingford: UK. pp 42-57.
Seal S. 1997. Molecular methods for detection and discrimination of R. solanacearum. Di dalam: Prior, P., C. Allen, and J.Elphinstone (eds). Bacterial Wilt Disease: Molecular and Ecological Aspects. Springer 103-109.
Singh MK, V Chandel, V Hallan, R Ram, AA Zaid. 2009. Occurrence of Peanut stripe virus on patchouli and raising of virus-free patchouli plants by meristem tip culture. Journal of Plant Diseases and Protection 116(1): 2–6. Shukla DD, Ward CW. 1988. Amino acid sequence homology of coat proteins as a basis for identification and classification of the Potyvirus group. J Gen Virol 69: 2703-2710.
Shukla DD, Ward CW, Brunt AA, Berger PH. 1998. Potyviridae family. Design and application of two novel degenerate primer pairs for the detection and complete genomic characterization of Potyviruses. Plant Viruses (366).
27
Sukamto IB, Rahardjo, Sulyo Y. 2007. Detection of Potyvirus on patchouli plant (Pogostemon cablin Benth.) from Indonesia. Di dalam: Prosiding International Seminar on Essential Oil. Jakarta 7-9 November 2007. 72-77. Thomas JE, Wong WC, Goanlock DH. 1989. Modern methods for the detection
of plant pathogens. Queensland Agric 49-53.
Uyemoto JK, Provvidenti R. 1974. Isolation and identification of two serotypes of Broad bean wilt virus. Phytopathology 64: 1547–1548.
Ward CW, Shukla DD. 1990. Taxonomy of Potyvirus: current problems and some solutions. Intervrology 32: 269-296.
Wellink J, et al. 2000. Family Comoviridae pp 691-701. Di dalam: M. H. V. van Regenmortel, et al. (eds). Virus Taxonomy. Seventh Report of the International Committee on Taxonomy of Viruses. Academic Press: New York.