LAPORAN PRAKTIKUM
REKAYASA GENETIKA
LAPORAN II
(ISOLASI DNA GENOM)
KHAIRUL ANAM
P051090031/BTK
BIOTEKNOLOGI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
ISOLASI DNA GENOM
TUJUAN
Praktikum ini bertujuan untuk mengisolasi DNA genom bakteri dan menganalisa dengan
16s rRNA.
TINJAUAN PUSTAKA
Isolasi DNA genom sangat penting dalam rangka pengklonan DNA atau gen sasaran.
Konstruksi pustaka genom dan pengklonan DNA membutuhkan DNA yang utuh agar fragmen
DNA betul-betul berasal dari proses pemotongan enzimatik yang sangat spesifik. Bila
terpotongnya DNA bukan karena reaksi enzimatik, maka fragmen DNA tersebut sulit
disambungkan dengan DNA dari vektor pengklonan. Oleh sebab itu isolasi DNA genom yang
utuh sangat diperlukan bila DNA tersebut akan diproses untuk pengklonan (Suharsono dan
Widyastuti, 2006).
16S rRNA
Berbagai metode analisis DNA yang cepat telah diperkenalkan, antara lain yang
mentarget keseluruhan genom seperti AFLP, RAPD, ERIC, BOX, REP, dan PFGE. Analisis
juga dapat mentarget hanya suatu klaster gen seperti ribotyping pada operon rrn, atau bahkan gen individual seperti ARDRA dan T-RFLP pada gen penyandi 16S rRNA, intergenic spacer regions/ISR, serta elemen genetik mobile. Di antara berbagai teknik yang digunakan, RNA ribosomal paling banyak digunakan sebagai penanda molekuler. Pada prokaryota terdapat tiga
jenis RNA ribosomal, yaitu 5S, 16S, dan 23S rRNA. Di antara ketiganya, 16S rRNA yang paling
sering digunakan. Molekul 5S rRNA memiliki urutan basa terlalu pendek, sehingga tidak ideal
dari segi analisis statistika, sementara molekul 23S rRNA memiliki struktur sekunder dan tersier
yang cukup panjang sehingga menyulitkan analisis. Analisis gen penyandi 16S rRNA telah
menjadi prosedur baku untuk menentukan hubungan filogenetik dan menganalisis suatu
ekosistem.
16S rRNA dapat digunakan sebagai penanda molekuler karena molekul ini bersifat
ubikuitus dengan fungsi yang identik pada seluruh organisme. Molekul ini juga dapat berubah
Molekul 16S rRNA memiliki beberapa daerah yang memiliki urutan basa yang relatif konservatif
dan beberapa daerah urutan basanya variatif. Perbandingan urutan basa yang konservatif
berguna untuk mengkonstruksi pohon filogenetik universal karena mengalami perubahan relatif
lambat dan mencerminkan kronologi evolusi bumi. Sebaliknya, urutan basa yang bersifat variatif
dapat digunakan untuk melacak keragaman dan menempatkan galur-galur dalam satu spesies.
Jika urutan basa 16S rRNA menunjukkan derajat kesamaan yang rendah antara dua taksa,
deskripsi suatu takson baru dapat dilakukan tanpa hibridisasi DNA-DNA (Biasanya jika derajat
kesamaan urutan basa gen penyandi 16S rRNA kurang dari 97% dapat dianggap sebagai
spesies baru. Analisis gen penyandi 16S rRNA praktis untuk definisi spesies, karena molekul ini
bersifat ubikuitus, sehingga dapat dirancang suatu primer yang universal untuk seluruh
kelompok. Penentuan spesies baru pun dapat dilakukan tanpa mengisolasi mikroorganisme
yang bersangkutan. Taksa baru yang ditetapkan hanya berdasarkan data molekular oleh The International Committee on Systematic Bacteriology diberi status provisional candidatus (Pangastuti, 2006).
BAHAN DAN METODE KERJA
Isolasi DNA Genom Bakteri
Bakteri yang digunakan dalam praktikum ini adalah Bacillus I3
Satu koloni bakteri E. coli ditumbuhkan dalam 50 ml media LB (bacto tryptone 10g/l, bacto-yeast extract 5g/l, NaCl 10 g/l) yang mengandung ampicillin lalu diinkubasi di atas shaker
dengan kecepatan 250 rpm pada suhu 37°C selama semalam. Kemudian 1,5 ml kultur
dipindahkan ke dalam tabung 1,5 ml lalu diendapkan dengan sentrifugasi 8.000 rpm (Tomy
MRX-150) 4°C selama 3 menit, supernatan dibuang. Lalu endapan bakteri disuspensikan ke
dalam 600 ul larutan CTAB lalu diresuspensi. Larutan diinkubasi pada suhu 65°Cselama 30-60
menit, lalu taruh di dalam es selama 5 menit. Tambahkan larutan CI (chloroform:isoamilalkohol,
4:1) lalu tabung dibolak-balik secara perlahan beberapa kali. Tabung disentrifugasi selama 10
menit pada kecepatan 14.000 rpm. Akan terbentuk dua fase pada larutan yang ada di dalam
tabung yaitu fase air dan fase kloroform. Di ambil 500 ul fase air lalu dimasukkan ke dalam
tabung baru yang kemudian ditambahkan dengan 500 ul PCI (phenol:chloroform:isoamilalkohol,
25:24:1). Bolak-balik tabung selama beberapa kali secara perlahan lalu disentrifugasi pada
kecepatan 14.000 rpm selama 10 menit. Fase air yang terbentuk diambil dan dimasukkan ke
etanol absolut, kemudian campur secara perlahan. Taruh ke dalam pendingin -20°C selama 30
menit. Larutan yang ada di dalam tabung disentrifugasi selama 15 menit pada kecepatan
14.000 rpm. Buang semua supernatant. Ke dalam tabung yang berisi pelet, ditambahkan 500 ul
etanol 70% lalu disentrifugasi selama 5 menit pada kecepatana 14.000 rpm. Setelah itu, buang
supernatant secara hati-hati dan keringkan pelet dengan vakum. Pelet adalah DNA genom. Ke
dalam tabung, ditambahkan 10 ul dH2O lalu ditambahkan 1 ul RNase dan di inkubasi pada suhu
37°C selama 1 jam hingga semalaman.
PCR 16S rRNA
Dilakukan PCR dengan menggunakan Primer spesifik 16s rRNA, yaitu primer F =
5’CAGGCCTAACACATGCAAGTC 3’ dan R = 5’GGGCGGWGTGTACAAGGC 3’ dimana W
adalah mewakili A dan T, dengan kondisi PCR sebagai berikut, prePCR = 95°C; 5 menit;
denaturasi = 84°C, 1 menit; annealing = 50°C; 30 detik, extension = 72°C; 30 detik, post PCR =
72°C; 5 menit; Penyimpanan = 15°C; 15 menit. Komposisi larutan PCR adalah DNA genom = 1
ul (100ng), F= 1 ul (0,5 uM), R=1 ul (0,5 uM), dNTP mix 2uM= 2 ul (200 uM), buffer taq 10x = 2
Dapat diketahui bahwa dalam proses mengisolasi plasmid dari suatu bakteri, ada tiga
tahap penting yang perlu dilakukan, yaitu 1. Lisis membran sel bakteri, 2. Ektraksi DNA, 3.
Pengendapan DNA.
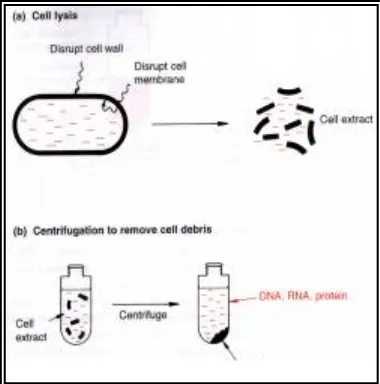
Proses lisis diawali dengan adanya pemberian CTAB dimana CTAB berperan untuk
melisis membran sel bakteri untuk mengeluarkan DNA genom. Peran CTAB persis sama
dengan SDS yang berfungsi sebagai detrjen untuk melisis dinding atau membran sel yang
terdiri dari lipid (fosfolipid). Terjadinya proses lisis ditandai dengan terbentuknya lendir. Akan
tetapi dalam praktikum kali ini tidak terbentuk lendir seperti biasanya. Tidak terbentuknya lendir
dimungkinkan karena proses lisis tidak berjalan dengan baik. Tidak berjalan dengan baik bisa
dikarenakan kurang kuatnya CTAB untuk melisis membran sel bakteri atau kurangnya
perlakuan terhadap sel bakteri seperti dilakukannya vortex untuk membantu proses lisis. Akan
mendapatkan DNA yang utuh sehingga ditakutkan apabila divortex DNA keluar akan tetapi
akan terpotong-potong.
Gambar 1. Lisis dinding dan membran sel bakteri
Proses tetap dilanjutkan dengan mengekstraksi larutan setalah lisis dengan CI. Setelah
Pada larutan diekstraksi dengan CI dan diambil fase airnya yang terdapat DNA proses
diteruskan dengan ekstraksi dengan penambahan PCI (Phenol-Chloroform-Isoamyl Alcohol)
dengan tujuan untuk memisahkan antara DNA dan komponen lainnya dimana
Phenol-Chloroform berfungsi sebagai pelarut dari senyawa organik dan komponen lipid. Dengan
dilakukannya ekstraksi menggunakan PCI maka setelah disentrifugasi terbentuklah 3 fase
dimana terdiri dari fase air yang ada di paling atas tempat DNA plasmid berada, protein yang
terkoagulasi di fase yang ada di tengah dan fase Phenol-Chloroform yang ada di paling bawah
karena sifat chloroform yang berat jenisnya besar. Proses ekstraksi dilakukan dua kali untuk
pemisahan DNA dengan komponen lainnya menjadi lebih baik.
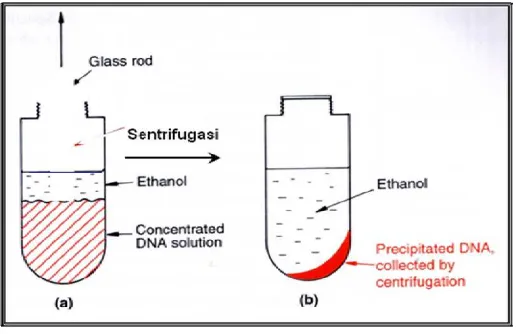
Fase air yang diambil kemudian diendapkan menggunakan sodium acetat untuk
menciptakan kondisi netral dan alkohol untuk mengikat air yang sebelumnya terikat pada DNA
sehingga DNA mengendap. Penambahan RNase dapat diberikan untuk menghilangkan
sisa-sisa fragmen RNA.
Gambar 3. Pemurnian DNA dengan etanol
Analisis Kemurnian DNA dan Kuantifikasi DNA dengan Spektrofotometer
Dari DNA plasmid yang diisolasi diperoleh dilakukan kuantifikasi dengan menggunakan
metode spektrofotometri dengan menggunakan mesin nanodrops didapatkan hasil seperti pada
Tabel 1.
Tabel 1. Nilai absorbansi larutan DNA pada panjang gelombang 260 dan 280 nm
Sampel Konsentrasi Unit A260
1 171 ng/µl 3.421
Dari hasil pengukuran secara spektrofotometri maka diperoleh konsentrasi sampel DNA genom
Analisis DNA dengan Elektroforesis Gel Agarosa
Dengan diketahuinya konsentrasi DNA genom maka dapat dihitung berapa ul yang
dibutuhkan untuk mendapatkan 100 ng DNA untuk proses elektroforesis, yaiu 0.9 ul larutan
DNA yang kemudian ditambahkan loading dye sehingga diperoleh hasil elektroforesis sebagai berikut seperti yang terlihat pada Gambar 4.
Dari Gambar 4 diketahui bahwa semua sampel tidak mengandung pita yang
menandakan keberadaan genom. Hal ini bisa dikarenakan DNA genom tidak keluar selama
proses lisis karena pada proses lisis tidak terbentu lendir yang menandakan adanya protein dan
DNA yang keluar dari sel. Ataupun juga karena bobot genom yang besar mengakibatkan
genom terperangkap di dalam sumur sehingga dibutuhkan waktu lama atau tegangan yang
lebih tinggi.
Gambar 4. Hasil elektroforesis DNA genom
PCR 16S rRNA
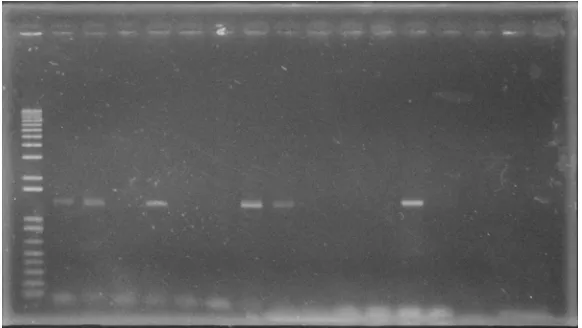
Pada praktikum kali ini dilakukan PCR 16s rRNA dengan hasil seperti yang terlihat pada
Gambar 5. 16S rRNA ditujukan sebagai penanda molekuler karena molekul ini bersifat ubikuitus
dengan fungsi yang identik pada seluruh organisme dimana untuk organism prokariot seperti
Bacillus I3 dan I2 memiliki kesamaan pada gen penghasil 16S rRNA ini. Dengan dilakukannya
PCR 16S rRNA ini maka dapat diketahui mana yang merupakan bakteri dan yang bukan bakteri
seperti missal organism eukariot memiliki 18s rRNA maka apabila terdapat pita yang berada
bukan pada 16s rRNA maka organism tersebut bukan bakteri dan atau PCR yang dilakukan
kurang sempurna yang mengakibatkan hasil PCR tidak dapat digunakan sebagai acuan untuk
Gambar 5. Hasil PCR 16s rRNA
Dari gambar 5 juga dapat diketahui bahwa tidak semua hasil isolasi DNA genom
memiliki pita pada ukuran 1600 pasang basa. Hal ini mengindikasikan bahwa PCR yang
dilakukan tidak berhasil, bisa dikarenakan kurang banyaknya DNA yang terbentuk akibat terlalu
banyak penambahan bobot molekul dari DNA yang akan di PCR sehingga primer dan DNA Taq
Polimerase tidak bekerja secara maksimal maka amplifikasi yang dilakukan tidak maksimal.
SIMPULAN
1. Dari isolasi DNA plasmid, diperoleh DNA plasmid yang kurang murni dan diperoleh
konsentrasi plasmid sebesar 110.6 ng/ul.
2. 16s rRNA digunakan sebagai penanda molekular untuk organism prokariot.
DAFTAR ACUAN
Suharsono dan Widyastuti, U. 2006. Penuntun Praktikum Pelatihan Teknik Pengklonan Gen.
Pusat Penelitian Sumber Daya Hayati dan Bioteknologi, IPB
Pangastuti, A. 2006. Definisi Spesies Prokaryota Berdasarkan Urutan Basa Gen Penyandi 16s rRNA dan Gen Penyandi Protein.Biodiversitas, Volume 7, Nomor 3 : Halaman: 292-296 Bahan Kuliah dan Praktikum Rekayasa Genetika