PENGKLONAN DAN EKSPRESI GEN PENYANDI
PARTIKULAT METAN MONOOKSIGENASE DOMAIN
CUPREDOXIN (spmoB) PADA Escherichia
coli
TIRA SITI NUR AFIAH
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Pengklonan dan Ekspresi Gen Penyandi Partikulat Metan Monooksigenase Domain Cupredoxin (spmoB) pada Escherichia coli adalah benar karya saya dengan arahan dari komisi pembimbing Prof Dr Ir Suharsono, DEA serta Dr Ir Iman Rusmana, MSi dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
TIRA SITI NUR AFIAH. Pengklonan dan Ekspresi Gen Penyandi Partikulat Metan Monooksigenase Domain Cupredoxin (spmoB) pada Escherichia coli. Dibimbing oleh SUHARSONO dan IMAN RUSMANA.
Metan merupakan gas yang berkontribusi terhadap pemanasan global dan menyerap radiasi inframerah yang lebih tinggi dibandingkan CO2. Gas metan
dapat digunakan sebagai sumber karbon oleh bakteri metanotrof. Jalur metabolisme awal pada bakteri metanotrof adalah reaksi konversi metan menjadi metanol yang dikatalisis oleh enzim metan monoksigenase (MMO). Terdapat dua tipe MMO yaitu soluble MMO (sMMO) dan partikulat MMO (pMMO). Enzim pMMO adalah enzim yang dominan dalam mengoksidasi metan di alam.
Enzim pMMO adalah protein integral membran yang terdiri dari tiga subunit yaitu subunit pmoC, pmoA, dan pmoB yang disandikan oleh operon pmoCAB. Bakteri metanotrof memiliki laju pertumbuhan yang lambat, sehingga dilakukan pendekatan lain yaitu dengan mengekspresikan operon penyandi pMMO pada Escherichia coli. Namun demikian, ekspresi seluruh gen penyandi pMMO di E. coli sulit dilakukan sehingga menimbulkan dugaan bahwa protein integral ini bersifat toksis bagi bakteri ini. Oleh karena itu, pada penelitian ini dilakukan pendekatan lain yaitu dengan mengekspresikan hanya sisi aktif dari enzim pMMO; subunit pmoB domain cupredoxin yang disandikan oleh gen spmoB di E. coli,
Gen spmoB yang digunakan adalah gen dari bakteri Methylococcus capsulatus (Bath). Urutan nukleotida gen ini diperoleh dari situs National Center for Biotechnology Information (NCBI) untuk proses pembuatan gen sintetik. Beberapa modifikasi dilakukan sehingga gen ini diekspresikan di sitoplasma. Gen spmoB diekspresikan di E. coli BL21 (DE3) menggunakan promotor T7 yang terdapat di dalam vektor ekspresi, yaitu pET15b. Analisis ekspresi dilakukan dengan SDS-PAGE dan pengujian aktivitas oksidasi metan oleh protein spmoB dilakukan dengan mengukur akumulasi metanol yang dihasilkan.
Ukuran gen penyandi domain cupredoxin adalah 891 pb. Gen ini berhasil diekspresikan di E. coli BL21 (DE3) dibawah promotor T7. Analisis ekspresi gen dengan SDS-PAGE menunjukan bahwa ekspresi berlebih dapat dilakukan dengan induksi isopropyl β-D-thiogalactoside (IPTG) konsentrasi 0.1, 0,5, dan 1.0 mM. Suhu 27 oC dan 37 oC dapat digunakan sebagai suhu inkubasi. protein spmoB menghasilkan protein rekombinan berukuran sekitar 38.9 kDa. Pengujian aktivitas
protein spmoB menunjukan jumlah metanol yang terakumulasi selama oksidasi metan oleh bakteri rekombinan adalah sebesar 0.114 mmol mL kultur-1 jam-1.
Protein rekombinan ini tidak menimbulkan toksisitas bagi E. coli sehingga memungkinkan untuk analisis lebih lanjut terhadap potensi aplikasi protein dan gen penyandi spmoB.
SUMMARY
TIRA SITI NUR AFIAH. Cupredoxin Domain of Particulate Methane Monooxygenase (spmoB) Gene Cloning and Expression in Escherichia coli. Supervised by SUHARSONO and IMAN RUSMANA.
Methane is one of the gas that contributes in global warming and absorbs more infrared radiation significantly than CO2. Methane can be used by
methanotroph bacteria as a source of carbon. The first step in the metabolic pathway of methanotrophs bacteria is reaction of methane convertion to methanol. This reaction is catalyzed by methane monooxygenase (MMO) enzyme. There are two types of MMO; soluble MMO (sMMO) and particulate MMO (pMMO), in which pMMO is the dominant enzyme of methane oxidation in nature.
Particulate methane monooxygenase (pMMO) is an integral membrane protein that is composed of three subunits pmoC, pmoA, and pmoB, encoded by
pmoCAB operon. Methanotroph have a slow growth rate that make researchers have to develop an alternative approach by expressing the pMMO operon in Escherichia coli. However, it was very difficult to express all the pMMO encoded
genes in E. coli and it was suspected that this integral protein might be toxic to E. coli. Therefore, this research tried another approach by expressing the active site
of pMMO enzyme; cupredoxin domain of pmoB subunit encoded by spmoB gene.
The spmoB gene used was the gene from Methylococcus capsulatus (Bath). The sequence of this gene was obtained from National Center for Biotechnology Information (NCBI) for the manufacture of artificial gene. Several modifications were made so spmoB gene would be expressed in the cytoplasm. The spmoB gene was expressed in E. coli BL21 (DE3) under T7 promoter and
pET15b as the expression vector. Expression analysis of spmoB gene was
performed by SDS - PAGE and assay of methane oxidation activity by spmoB protein has be done by measuring the amount of methanol accumulated.
The size of cupredoxin domain encoded gene was 891 bp. This gene was successfully expressed in E. coli BL21 (DE3) under T7 promoter. Expression
analysis with SDS-PAGE showed that overexpression of this gene could be done at concentrations 0.1 mM, 0.5 mM, and 1.0 mM of isopropyl β-D-thiogalactoside (IPTG). Temperature of 27 oC and 37 oC could be used as incubation temperature. The spmoB gene expression produced a recombinant protein with a size
approximately 38.9 kDa. Assay of spmoB protein activity showed that the amount of methanol accumulated during methane oxidation by the recombinant strain was 0.114 mmol mL culture-1 h-1. The expression of this gene did not cause
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Bioteknologi
PENGKLONAN DAN EKSPRESI GEN PENYANDI
PARTIKULAT METAN MONOOKSIGENASE DOMAIN
CUPREDOXIN (spmoB) PADA Escherichia coli
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan September 2014 hingga bulan Agustus 2015 ini ialah Pengklonan dan Ekspresi Gen Penyandi Partikulat Metan Monooksigenase Domain Cupredoxin (spmoB) pada Escherichia coli.
Terima kasih penulis ucapkan kepada Bapak Prof Dr Ir Suharsono, DEA dan Bapak Dr Ir Iman Rusmana, MSi selaku pembimbing, atas segala arahan, saran, dan solusi selama pelaksanaan penelitian dan penyusunan karya ilmiah ini. Terima kasih kepada Bapak Prof Dr Aris Tri Wahyudi, MSi selaku penguji luar komisi atas saran yang diberikan pada saat ujian tesis. Di samping itu, penulis mengucapkan terima kasih kepada DIKTI melalui program beasiswa unggulan tahun 2013/2014, yang telah memberikan dana perkuliah selama studi penulis di IPB.
Terima kasih kepada Bapak Jaka dan rekan-rekan di laboratorium Mikrobiologi IPB serta Laboratorium PPSHB IPB atas bantuan selama pelaksanaan penelitian. Terima kasih kepada teman-teman seperjuangan Natalia, Nabilah, Retno, Albert, dan Jekmal, atas segala motivasi dan keceriaan selama masa studi. Ungkapan terima kasih tak terhingga juga disampaikan kepada Bapak, Mama, serta seluruh keluarga, atas segala dukungan, doa, serta kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2015
DAFTAR ISI
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
TINJAUAN PUSTAKA 3
Bakteri Metanotrof 3
Oksidasi Metan oleh Bakteri Metanotrof 4
Enzim Partikulat Metan Monooksigenase (pMMO) 5
Gen pmoC, pmoA, dan pmoB 7
METODE 9
Kerangka Penelitian 9
Waktu dan Tempat Penelitian 10
Desain dan Perbanyakan Gen Sintetik spmoB M. capsulatus (Bath) 10
Subklon Gen spmoB 10
Isolasi Plasmid Rekombinan dan Verifikasi Transforman 11 Pengklonan Gen spmoB pada Vektor Ekspresi pET15b 11 Analisis Ekspresi Gen spmoB dengan SDS-PAGE 12
Pengujian Aktivitas Oksidasi Metan 13
HASIL DAN PEMBAHASAN 13
Perbanyakan Gen Sintetik dan Subklon Gen spmoB 13 Konstruksi Vektor Ekspresi dan Verifikasi Transforman 15 Analisis Ekspresi dan Pengujian Aktivitas Oksidasi Metan 16
SIMPULAN DAN SARAN 178
Simpulan 18
Saran 18
DAFTAR PUSTAKA 19
LAMPIRAN 22
DAFTAR GAMBAR
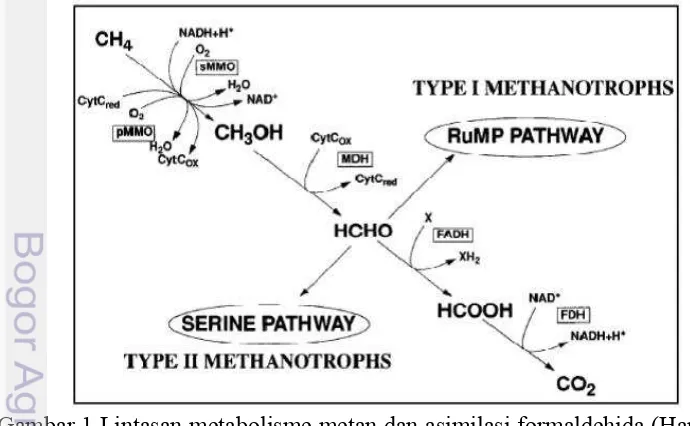
1 Lintasan metabolisme metan dan asimilasi formaldehida 3
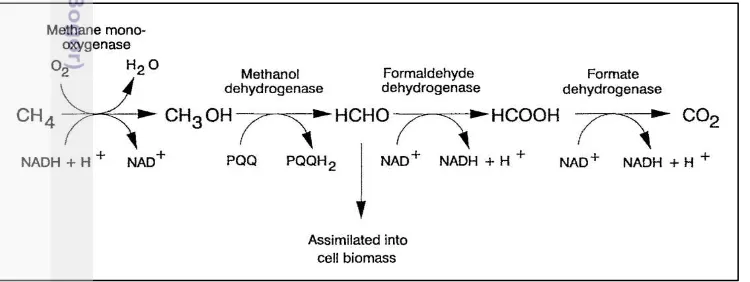
2 Lintasan oksidasi metan oleh metanotrof 4
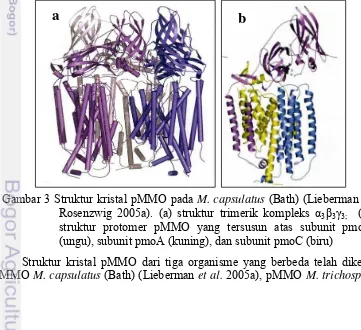
3 Struktur kristalpMMO pada M. capsulatus (Bath) 5
4 Struktur subunit pmoB 6
5 Gen penyandi metan monooksigenase 7
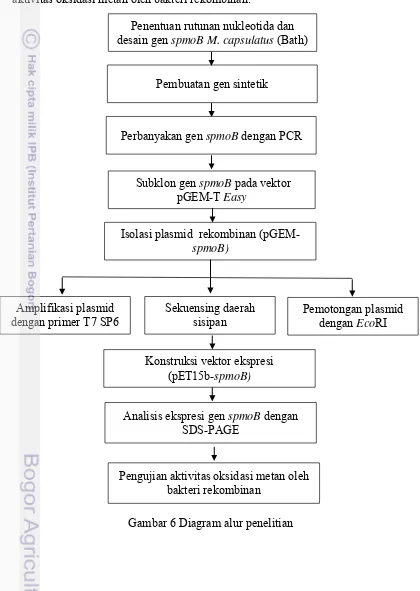
6 Diagram alur penelitian 9
7 Peta fisik vektor pGEM-T Easy (Promega) 11
8 Peta fisik vektor ekspresi pET15b (Novagen) 12 9 Peta fisik dan urutan nukleotida daerah penyisipan pada vektor pET15b 12 10 Gen sintetik penyandi spmoB yang dikonstruksi 14 11 Gen sintetik spmoB yang diamplifikasi dengan PCR 14 12 Prediksi titik isoelektrik (pI) dan ukuran berat molekul (Mw) protein 14 13 Verifikasi gen spmoB dengan menggunakan plasmid dari bakteri E. coli
DH5α transforman yang mengandung pGEM-spmoB 15
14 Vektor ekspresi pET15b-spmoB yang dikonstruksi 16 15 Hasil pemotongan plasmid pET15b-spmoB dengan enzim NdeI dan
BamHI 16
16 Analisis ekspresi gen spmoB yang diinduksi IPTG dengan SDS-PAGE 17
DAFTAR LAMPIRAN
1 Pembuatan sel kompeten dengan perlakuan CaCl2 22
2 Komposisi gel poliakrilamida 22
3 Perhitungan nilai Rf marker 22
4 Kurva standar marker 22
5 Komposisi reagen Sodium Nitroprusside (SNP) 23
6 Prosedur pengukuran kadar metanol dengan reagen SNP 23 7 Urutan nukleotida gen spmoB M. capsulatus (Bath) 23
PENDAHULUAN
Latar Belakang
Metan merupakan gas kedua tertinggi yang berkontribusi terhadap pemanasan global dan menyerap radiasi infra merah yang lebih tinggi dibandingkan CO2 (Lelieveld et al. 1993; Tol et al. 2003). Gas metan secara alami diproduksi oleh arkea metanogenik yang hidup dalam kondisi lingkungan anaerobik seperti danau, rawa, dan lahan sawah basah. Lahan sawah basah merupakan salah satu sumber emisi metan yang menghasilkan sekitar 575 Tg metan per tahun dan sekitar 100 Tg dilepaskan ke atmosfer (Hanson & Hanson 1996). Emisi metan yang dihasilkan oleh aktivitas pertanian dan industri mengalami peningkatan sebanyak dua kali lipat selama 2 abad (Hanson & Hanson 1996). Berdasarkan laporan dari Emisi Global Akibat Kegiatan Manusia dari Gas Rumah Kaca Non Karbon Dioksida, Environmental Protection Agency (EPA) (2010), Indonesia berada pada urutan ketujuh sebagai negara penghasil emisi metan dan sebagian besar emisi ini dihasilkan oleh sawah (http://www.epa.gov/).
Metanotrof adalah bakteri yang mampu tumbuh dengan menggunakan metan sebagai sumber karbon dan energi. Bakteri ini memiliki lintasan metabolisme oksidasi metan menjadi metanol. Reaksi oksidasi metan ini dikatalisis oleh enzim metan monoksigenase (MMO). Oleh karena itu, bakteri metanotrof dan enzim MMO mempunyai potensi untuk mereduksi gas metan di atmosfer. Akan tetapi, aplikasi bakteri metanotrof terhalang oleh laju pertumbuhannya yang lambat serta densitas sel yang rendah (Yu et al. 2003; Gou et al. 2006).
2
pmoC belum diketahui perannya (Lieberman & Rosenzweig 2005a; Lieberman & Rosenzweig 2005b). Subunit pmoB terdiri dari domain N-terminal cupredoxin (spmoBd1) dan domain C-terminal cupredoxin (spmoBd2) yang dihubungkan oleh dua transmembran heliks. Kedua domain cupredoxin berada pada periplasma dan merupakan satu-satunya domain yang soluble (Culpepper & Rosenzweig 2012). Residu 32 asam amino awal diduga merupakan sinyal peptida (Semrau et al. 1995). Gen penyandi domain cupredoxin pada subunit pmoB, yaitu gen spmoB, merupakan penyandi sisi aktif pMMO. Hal ini dibuktikan oleh Smith et al. (2011b) yang mengekspresikan hanya gen spmoB di sitoplasma dan tetap memiliki aktivitas oksidasi metan.
Pada penelitian ini, gen penyandi subunit pmoB bakteri M. capsulatus (Bath) yang telah dihilangkan gen penyandi transmembran heliks dan sinyal peptidanya diekspresikan di E. coli BL21 (DE3). Methylococcus capsulatus (Bath) dipilih karena keseluruhan informasi genomnya, termasuk operon pmoCAB, telah diketahui (Ward et al. 20004). Informasi struktur dan sisi aktif pMMO bakteri ini juga telah diketahui (Lieberman & Rosenzweig 2005a; Smith et al. 2011b; Culpepper & Rosenzweig 2012). Gen ini diekspresikan di E. coli BL21 (DE3) dibawah kendali promotor T7 yang terdapat di dalam vektor ekspresi pET15b.
Perumusan Masalah
Indonesia sebagai salah satu negara yang memiliki lahan sawah tergenang yang luas turut menghasilkan emisi gas metan. Metanotrof dan enzim pMMO dengan kemampuannya mengoksidasi metan menjanjikan sebagai agen pereduksi emisi gas metan, akan tetapi bakteri ini memiliki laju pertumbuhan yang relatif lambat. Ekspresi keseluruhan domain pMMO bersifat toksik bagi E. coli sehingga diperlukan pendekatan lain. Penelitian tentang pengklonan gen penyandi pMMO domain cupredoxin dan mengekspresikannya perlu dilakukan .
Tujuan Penelitian
Penelitian ini bertujuan untuk mengklon gen penyandi pMMO domain cupredoxin dan mengekspresikannya di Escherichia coli menggunakan vektor ekspresi pET15b.
Manfaat Penelitian
Hasil dari penelitian ini adalah gen sintetik penyandi pMMO subunit pmoB domain cupredoxin dan bakteri transforman yang mampu mengekspresikan gen ini secara berlebih tanpa menimbulkan toksisitas serta memiliki kemampuan untuk mengoksidasi metan. Gen penyandi pMMO serta bakteri transforman ini berpotensi sebagai agen pereduksi emisi gas metan.
Ruang Lingkup Penelitian
3
TINJAUAN PUSTAKA
Bakteri Metanotrof
Bakteri metanotrof adalah kelompok bakteri Gram negatif yang mampu menggunakan metan sebagai satu-satunya sumber karbon dan energi (Semrau et al. 1995). Bakteri metanotrof dapat ditemukan pada lumpur, rawa, sungai, lahan sawah, laut, tanah padang rumput, sedimen, serasah, kolam, serta endapan limbah (Hanson & Hanson 1996). Beberapa bakteri metanotrof strain asidofilik, alkalifilik, termofilik, dan psikrofilik dilaporkan dapat diisolasi dari berbagai lingkungan yang ekstrim (Lieberman & Resenzweig 2004). Bakteri metanotrof memiliki peranan yang penting dalam siklus karbon, oksigen, dan nitrogen (Hanson & Hanson 1996). Metanotrof tumbuh dengan baik pada kondisi aerob, meskipun bakteri ini juga dapat tumbuh pada lingkungan mikroaerofil.
Bakteri metanotrof dapat diklasifikasikan menjadi tiga kelompok berdasarkan lintasan metabolisme metan dan asimilasi formaldehida (Hanson & Hanson 1996; Semrau et al. 2010). Terdapat dua jalur metabolisme asimilasi formaldehida yaitu jalur ribulosa monofosfat (RuMP) dan jalur serin (Gambar 1). Metanotrof tipe I adalah Gammaproteobacteria yang terdiri dari genera Methylomonas, Methylobacter, Methylobacterium, Methylocaldum, dan Methylosphaera. Kelompok bakteri ini memiliki berkas pada membran intrasitoplasmik yang pradominan dengan asam lemak 14 dan 16 karbon, serta menggunakan jalur RuMP untuk asimilasi karbon. Metanotrof tipe II adalah Alphaproteobacteria yang terdiri dari Methylosinus, Methylocella, Methylicapsa, dan Methylocystic. Bakteri ini memiliki membran intrasitoplasmik yang tersusun dari asam lemak 18 karbon, serta menggunakan jalur serin untuk asimilasi karbon. Kelompok yang ketiga adalah matanotrof tipe X seperti Methylocaldum dan Methylococcus capsulatus (Bath) yang memiliki ciri-ciri tipe I, yaitu memiliki fosfolipid asam lemak 16 karbon serta jalur metabolisme RuMP. Tipe X dibedakan dari tipe I karena tipe X memiliki enzim ribulosa-bifosfat karboksilase, suatu enzim yang juga terdapat dalam siklus Calvin-Benson (Bowman et al. 1993; Hanson & Hanson 1996).
4
Metanotrop tipe X tumbuh pada temperatur yang lebih tinggi dari pada tipe I dan II, serta memiliki persentase GC yang lebih tinggi dibandingkan dengan kebanyakan metanotrof tipe I (Hanson & Hanson 1996). Selain metanotrof yang tergabung dalam filum gamma dan Alfaproteobakteria, terdapat genus Methylacidiphila yang dimasukkan ke dalam filum Verrucomicrobia dan secara filogenetik berkerabat dekat dengan metanotrof tipe I (Semrau et al. 2010). Anggota metanotrof ini merupakan termoasidofilik yang dapat tumbuh pada temperatur lebih dari 50 °C dan pH di bawah 5.
Oksidasi Metan oleh Bakteri Metanotrof
Oksidasi metan dapat terjadi baik dalam keadaan aerob maupun anaerob di berbagai lingkungan seperti lahan basah, sawah, tanah gambut, tanah hutan, dan tambang batu bara (Han et al. 2009). Berbagai penelitian dilakukan untuk mempelajari oksidasi metan pada metanotrof. Patel & Hoare (1971) melaporkan bahwa M. capsulatus hanya dapat tumbuh pada media dengan metan atau metanol sebagi sumber karbon dan energi. Methylobacter albus BG8 dan Methylosinus trichosporium OB3b juga mampu mengoksidasi metan pada kultur batch dengan substrat metanol (Benstead et al. 1998). Matheson et al. (1997) melaporkan tentang terhambatnya proses oksidasi metan pada M. capsulatus (Bath) ketika dipaparkan dengan hidroklorofluorokarbon 21, difluoroklorometan, fluorodiklorometan, dan berbagai metan terflorinasi.
Proses oksidasi metan secara aerobik dikatalisis oleh enzim metan monooksigenase (MMO). Tahap pertama oksidasi metan akan menghasilkan metanol yang kemudian dioksidasi menjadi formaldehida. Formaldehida kemudian diasimilasi ke dalam biomassa sel atau dioksidasi lebih lanjut menjadi karbondioksida untuk menghasilkan energi pereduksi untuk biosintesis dan hidroksilasi metan (Gambar 2) (Murrel 1994).
Murrel et al. (2000) menyatakan bahwa sebagian besar metanotrof tipe I hanya mempunyai MMO yang berikatan dengan membran intrasitoplasmik yang disebut partikulat MMO (pMMO), sedangkan metanotrof tipe II dan Methylococcus juga mempunyai MMO yang berada di sitoplasma yang dinamakan soluble MMO (sMMO). Dedysh et al. (2000) kemudian mengkarakterisasi metanotrof asidofilik yang hanya mengekspresikan sMMO dan tidak memiliki pMMO yaitu Methylocella palustris, Methylocella silvestris, dan Methylocella tundrae. Ketiga bakteri ini diketahui tidak hanya menggunakan
5 sumber karbon C1 untuk pertumbuhan, tetapi juga mampu menggunakan asetat, piruvat, suksinat, malat, dan etanol (Dedysh et al. 2005). Tidak adanya sistem membran internal yang mengandung pMMO membuat Methylocella secara morfologi dan metabolisme berbeda dengan metanotrof lain sehingga genus Methylocella digolongkan sebagai metanotrof fakultatif. (Dedysh et al. 2005).
Metanotrof yang memiliki pMMO sekaligus sMMO memiliki sistem regulasi yang dimediasi oleh konsentrasi ion tembaga (Cu2+) (Murrel et al. 2000).
Studi biokimia dan biofisik menunjukan bahwa aktivitas pMMO pada M. capsulatus (Bath) secara langsung dipengaruhi dan berkorelasi dengan konsentrasi ion tembaga (Cu2+)/total rasio protein membran. Enzim pMMO hanya
diekspresikan ketika kondisi lingkungan memiliki rasio Cu2+ yang tinggi dibandingkan dengan biomassa, sedangkan sMMO diekspresikan ketika konsentrasi Cu2+ yang rendah. Mekanisme regulasi resiprokal untuk kluster gen
pMMO dan sMMO belum sepenuhnya diketahui.
Enzim Partikulat Metan Monooksigenase (pMMO)
Enzim pMMO merupakan metaloenzim integral membran yang tersusun atas tiga subunit yaitu subunit γ (22 kDa), β (24 kDa), dan α (45 kDa) atau disebut juga subunit pmoC, pmoA, dan pmoB. Ketiga subunit membentuk struktur trimerik kompleks α3β3γ3 (Gambar 3a) (Lieberman & Rosenzwig 2005a;
Hakemian et al. 2008; Smith et al. 2011a). Setiap protomer terdiri dari satu salinan subunit pmoB, pmoA, dan pmoC (Gambar 3b) (Lieberman & Rosenzwig 2005a). Subunit pmoB terdiri dari domain N-terminal cupredoxin (spmoBd1), dua transmembran heliks, dan domain C-terminal cupredoxin (spmoBd2) (Gambar 4). Kedua domain cupredoxin berada pada periplasma dan merupakan satu-satunya domain yang soluble. Kedua subunit lainnya yaitu pmoA dan pmoC tersusun atas transmembran heliks (Lieberman & Rosenzweigh 2005a).
Struktur kristal pMMO dari tiga organisme yang berbeda telah diketahui, pMMO M. capsulatus (Bath) (Lieberman et al. 2005a), pMMO M. trichosporium
Gambar 3 Struktur kristal pMMO pada M. capsulatus (Bath) (Lieberman & Rosenzwig 2005a). (a) struktur trimerik kompleks α3β3γ3; (b)
struktur protomer pMMO yang tersusun atas subunit pmoB (ungu), subunit pmoA (kuning), dan subunit pmoC (biru)
6
OB3b (Hakemian et al. 2008), dan Methylocystis sp. strain M (Smith et al. 2011a). Tiga pusat metal telah ditemukan pada struktur pMMO M. capsulatus (Bath). Dua pusat metal, yaitu tembaga (Cu) mononuklear dan Cu dinuklear, berada pada daerah subunit pmoB (Lieberman & Rosenzweigh 2005a; Lieberman & Rosenzweigh 2005b). Pusat metal Cu mononuklear berada pada residu His 48 dan His 72 (Gambar 4). Pada M. trichosporium OB3b dan Methylocystis sp. strain M, residu yang mengikat Cu adalah residu asparagin. Pusat metal Cu dinuklear yang memiliki jarak Cu–Cu sekitar 2.5–2.7 Å berada pada residu His 33, His 137, dan His 139. Residu His 33 adalah ujung dari N-terminal subunit pmoB, sedangkan 32 residu awal adalah sekuen peptida sinyal yang dihilangkan secara in vivo. Ketiga residu histidin di daerah pusat ini sangat lestari pada seluruh anggota metanotrof, kecuali filum Verrucomicrobia (Culpepper & Rosenzweig 2012). Pusat metal yang ketiga berada dalam membran dan disusun oleh zink yang berada pada residu Asp 156, His 160, dan His 173 pada pmoC dan Glu 195 pada pmoA.
Gambar 4 Struktur subunit pmoB. pmoB domain N-terminal cupredoxin (spmoBd1) ditunjukkan dengan warna ungu, pmoB domain C-terminal cupredoxin (spmoBd2) ditunjukkan dengan warna hijau, dan dua transmembran heliks ditunjukan dengan warna biru (Balasubramanian et al. 2010)
7 pada pusat metal subunit pmoB domain cupredoxin. Smith et al. (2011b) kemudian mengekspresikan gen penyandi domain cupredoxin pada E. coli. Protein rekombinan yang dihasilkan mampu mengoksidasi metan menjadi metanol dengan penambahan Cu.
Selain mengoksidasi metan, pMMO juga mampu mengoksidasi hidrokarbon halogen seperti trikloroetilen (TCE), 1,2-diklorometan, dan kloroform (Hanson & Hanson 1996). TCE digunakan secara luas dalam industri tekstil, insektisida, serta sintesis polivinil krorid, dan diduga kuat bersifat karsinogenik bagi manusia (Lieberman & Rozenweig 2004). Dengan kemampuan tersebut, pMMO memiliki potensi sebagai agen bioremediasi serta pereduksi emisi gas metan.
Gen pmoC, pmoA, pmoB
Enzim pMMO disandikan oleh operon pmoCAB yang terdiri dari gen pmoC, pmoA, dan pmoB yang secara berurutan menyandikan subunit pmoC, pmoA, dan pmoB (Nguyen et al. 1997; Lieberman & Rosenzweig 2004) (Gambar 5). Pada M. capsulatus (Bath), operon ini ditranskripsikan menjadi mRNA polisistronik tunggal berukuran 3.3 kb (Nielsen et al. 1997).
Gambar 5 Gen penyandi metan monooksigenase. (a) operon pmoCAB yang menyandikan pMMO (b) pmoC, pmoA, dan pmoB secara berurutan menyandikan 3 subunit polipeptida penyusun pMMO yaitu subunit γ (22 kDa), β (24 kDa), dan α (45 kDa) (Lieberman & Rosenzweig 2004).
Stolyar et al. (1999) melaporkan analisis molekular pmo M. capsulatus (Bath). Bakteri ini memiliki dua salinan lengkap operon pmoCAB, dan salinan ketiga untuk pmoC. Operon ini masing-masing terdiri dari 783 pb penyandi pmoC, 756 pb penyandi pmoA, dan 1245 pb penyandi pmoB. Daerah kedua salinan pmoCAB hampir identik pada level urutan nukleotida, hanya terdapat 13 perbedaan nukleotida dari 3183 pb. Pada level asam amino, setiap produk hasil translasi hanya memiliki satu residu yang berbeda pada setiap salinan. Bakteri yang masing-masing operon ini telah dimutasikan masih tetap dapat tumbuh
8
(Stolyar et al. 2001). Hal ini menunjukan bahwa kedua salinan gen tersebut fungsional.
9
METODE
Kerangka Penelitian
Kerangka penelitian ini meliputi pembuatan gen penyandi pMMO domain cupredoxin (spmoB), subklon gen spmoB pada vektor pGEM-T Easy, konstruksi vektor ekspresi, analisis ekspresi gen spmoB dengan SDS-PAGE, dan pengujian aktivitas oksidasi metan oleh bakteri rekombinan.
Gambar 6 Diagram alur penelitian Penentuan rutunan nukleotida dan desain gen spmoB M. capsulatus (Bath)
Pembuatan gen sintetik
Subklon gen spmoB pada vektor pGEM-T Easy
Isolasi plasmid rekombinan (pGEM-spmoB)
Sekuensing daerah
sisipan Pemotongan plasmid dengan EcoRI Amplifikasi plasmid
dengan primer T7 SP6
Konstruksi vektor ekspresi (pET15b-spmoB)
Analisis ekspresi gen spmoB dengan SDS-PAGE
Perbanyakan gen spmoB dengan PCR
10
Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan September 2014 sampai dengan Juni 2015, bertempat di Laboratorium Mikrobiologi, Departemen Biologi, Fakultas Matematika dan Pengetahuan Alam, IPB.
Desain dan Perbanyakan Gen Sintetik spmoB M. capsulatus (Bath)
Urutan nukleotida gen spmoB spesies M. capsulatus (Bath) diperoleh dari situs National Center for Biotechnology Information (NCBI) (http://www.ncbi.nlm.nih.gov/) dengan nomor akses L40804.2. Pengeditan dan desain gen dilakukan dengan menggunakan program BioEdit. Pembuatan gen sintetik spmoB dilakukan melalui jasa perusahan Integrated DNA Technologies, Inc (IDT). Gen sintetik kemudian diperbanyak dengan metode Polymerase Chain Reaction (PCR) (Gene Amp® PCR system 2720). Primer forward adalah spmoBd1F (5′-GGAATTCCATATGCACGGTGAGAAATCGCGG-3′) dengan situs pengenalan enzim NdeI (urutan nukleotida yang digarisbawahi) dan primer reverse spmoBd2R (5′-CGGGATCCTTACATGAACGACGGGATCA-3’) dengan situs pengenalan enzim BamHI (urutan nukleotida yang digarisbawahi). Reaksi PCR terdiri dari DNA polimerase 5U (LA-Taq Takara), gen sintetik 1 ng, masing-masing primer 0.5 µM, bufer DNA-pol 1X, dNTPs 0.25 mM, dan ddH2O.
Amplifikasi dilakukan sebanyak 12 siklus. Gradien temperatur yang digunakan adalah pradenaturasi (94 C, 30 detik), denaturasi (94 C, 15 detik), penempelan (55 C, 15 detik), pemanjangan (72 C, 30 detik), dan pemanjangan akhir (72 C, 5 menit). Produk PCR kemudian dipurifikasi dan diklon pada plasmid pGEM-T Easy (Promega, USA).
Subklon Gen spmoB
Gen spmoB diligasikan pada daerah Multiple Cloning Site (MCS) plasmid pGEM-T Easy (Gambar 7). Reaksi ligasi terdiri dari 10 µL fragmen DNA (5 ng µL-1), 1 µL vektor pGEM-T Easy (50 ng µL-1), 2 µL bufer ligasi 10X, 1 µL
T4 (3 U µL-1) DNA ligase (Invitrogen, USA), dan 6 µL ddH
2O. Proses ligasi
dilakukan dengan perlakuan inkubasi selama 16 jam pada suhu 10 oC. Escherichia
11
Gambar 7 Peta fisik vektor pGEM-T Easy (Promega)
Isolasi Plasmid Rekombinan dan Verifikasi Transforman
Isolasi plasmid dilakukan pada koloni E. coli DH5α yang berwarna putih di media Luria agar (LA) selektif. Bakteri E. coli DH5α ditumbuhkan dalam 10 mL media LB yang mengandung 100 µg mL-1 ampisilin pada suhu 37 oC selama
12-16 jam. Plasmid rekombinan pGEM-spmoB diisolasi dengan High Speed Plasmid Mini Kit (Geneaid) sesuai dengan protokol. Hasil isolasi plasmid divisualisasi dengan elektroforesis pada gel agarosa 1% (w v-1).
Verifikasi E. coli DH5α transforman dilakukan dengan mengamplifikasi plasmid pGEM-spmoB menggunakan primer SP6 dan T7. Amplifikasi dilakukan sebanyak 30 siklus. Gradien temperatur yang digunakan adalah pradenaturasi (94 C, 5 menit), denaturasi (94 C, 30 detik), penempelan (55 C, 30 detik), pemanjangan (72 C, 1 menit), dan pemanjangan akhir (72 C, 10 menit). Untuk memastikan sisipan gen telah sesuai dengan target, dilakukan pemotongan plasmid pGEM-spmoB dengan enzim retriksi EcoRI. Plasmid dipotong dengan mereaksikan 5 µL DNA plasmid, 1 µL 10x bufer enzim restriksi, 1 µL enzim restriksi dan 3 µL ddH2O. Sampel diinkubasi pada suhu 37 °C selama semalam. Visualisasi produk PCR dan hasil pemotongan plasmid dilakukan dengan elektroforesis pada gel agarosa 1% (w v-1). Daerah penyandi spmoB diverifikasi dengan sekuensing DNA melalui jasa perusahaan 1st Base, Malaysia.
Pengklonan Gen spmoB pada Vektor Ekspresi pET15b
12
selama 60 detik. Bakteri tersebut disebar pada media LA yang mengandung antibiotik ampisilin 100 µg mL-1. Bakteri transforman diverifikasi dengan
memotong plasmid rekombinan pET15b-spmoB dengan enzim retriksi NdeI dan BamHI.
Gambar 8 Peta fisik vektor ekspresi pET15b (Novagen)
Gambar 9 Peta fisik dan urutan nukleotida daerah penyisipan pada vektor pET15b
Analisis Ekspresi Gen spmoB dengan SDS-PAGE
Escherichia coli BL21 (DE3) transforman ditumbuhkan dalam 3 mL media LB yang mengandung antibiotik ampisilin 100 µg mL-1 selama satu malam pada suhu 37 oC. Sebanyak 100 µL biakan kemudian dikulturkan pada 10 mL LB dan diinkubasi kembali pada suhu 27 oC atau 37 oC. Ketika kultur telah mencapai OD600 nm sebesar 0.6, ke dalam kultur ditambahkan isopropyl β-D-thiogalactoside
13 relatif (Rf) marker dan log berat molekul (BM) (Lampiran 3 & 4). Nilai Rf dihitung dengan rumus:
Sebagai perbandingan, berat molekul protein spmoB rekombinan diprediksi berdasarkan urutan asam amino melalui software ExPASy (http://web.expasy.org/compute_pi).
Pengujian Aktivitas Oksidasi Metan
E. coli BL21 (DE3) transforman ditumbuhkan dalam 3 mL media LB yang mengandung antibiotik ampisilin 100 µg mL-1 selama semalam pada suhu 27 oC.
Sebanyak 40 µL biakan tersebut ditambahkan ke dalam media LB 4 mL yang mengandung antibiotik ampisilin 100 µg mL-1 dan 10 µM CuSO
4 dalam tabung
berukuran 12 mL. Kultur diinkubasi pada suhu 27 oC hingga nilai OD 600 nm
mencapai 0.6. IPTG 0.1 mM kemudian ditambahkan dalam kultur. Tabung ditutup dengan sumbat karet, kemudian udara pada bagian head space diatur sehingga komposisinya mendekati 50% udara dan 50% gas metan. Kultur diinkubasi kembali pada suhu 27 oC selama 24 jam. Sebanyak 1 mL kultur diambil untuk diukur kadar metanol yang dihasilkan dengan reagen SNP (Lampiran 5 & 6) sesuai dengan metode Zhan et al.(2010).
HASIL DAN PEMBAHASAN
Perbanyakan Gen Sintetik dan Subklon Gen spmoB
Urutan nukleotida gen pmoB yang digunakan untuk mensintesis gen sintetik adalah urutan nukleotida dari bakteri metanotrof M. capsulatus (Bath). Methylococcus capsulatus (Bath) memiliki pMMO dan sMMO yang ekpresi keduanya dipengaruhi oleh konsentrasi ion tembaga (Cu2+) (Murrel et al. 2000). Bakteri inijuga sering menjadi model penelitian karena relatif mudah digunakan dalam sistem genetik (Chistoserdova et al. 2005).Rusmana dan Akhdiya (2009) telah mengisolasi bakteri metanotrof dari beberapa lahan sawah di Indonesia dan berhasil memperoleh isolat M. capsulatus dengan aktivitas oksidasi metan yang tertinggi dibandingkan isolat lainnya.
14
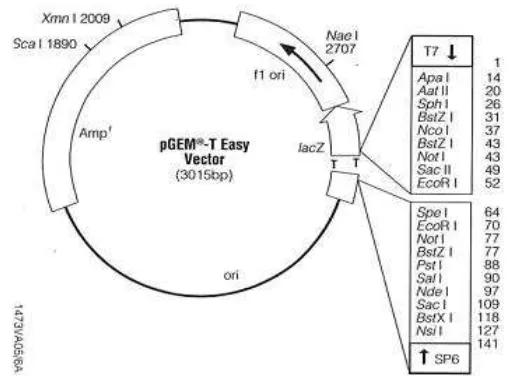
kodon awal. Kodon awal ini digantikan oleh sekuen situs retriksi NdeI sehingga proses translasi dapat berjalan. Domain transmembran heliks juga turut dihilangkan. Sampai saat ini, fungsi dari domain ini masih belum diketahui (Culpepper & Rosenzweig 2012). Asam amino Gly-Lys-Leu-Gly-Gly-Gly digunakan untuk menggantikan dua transmembran heliks sebagai penghubung residu 172 dan 265. Residu glisin dipilih karena memberikan pengaruh yang kuat terhadap fleksibilitas protein (Balasubramanian et al. 2010).
Gambar 10 Gen sintetik penyandi spmoB yang dikonstruksi
Hasil perbanyakan gen sintetik dengan primer spmoBd1F dan spmoBd2R menghasilkan produk berukuran 908 pb (Gambar 11). Modifikasi pada gen penyandi subunit pmoB menyebabkan ukuran gen menjadi lebih pendek, yaitu menjadi 891 pb. Basa tambahan sebanyak 17 pb merupakan sekuen pengenalan enzim retriksi NdeI dan BamHI. Gen ini diprediksi akan menghasilkan protein rekombinan dengan berat molekul 32.93 kDa (Gambar 12).
Gambar 11 Gen sintetik spmoB yang diamplifikasi dengan PCR. (M) marker 1kb; (1) gen spmoB memiliki ukuran 908 pb
10 20 30 40 50 60 MHGEKSQAAF MRMRTIHWYD LSWSKEKVKI NETVEIKGKF HVFEGWPETV DEPDVAFLNV 70 80 90 100 110 120 GMPGPVFIRK ESYIGGQLVP RSVRLEIGKT YDFRVVLKAR RPGDWHVHTM MNVQGGGPII 130 140 150 160 170 180 GPGKWITVEG SMSEFRNPVT TGKLGGGAGT MRGMKPLELP APTVSVKVED ATYRVPGRAM 190 200 210 220 230 240 RMKLTITNHG NSPIRLGEFY TASVRFLDSD VYKDTTGYPE DLLAEDGLSV SDNSPLAPGE 250 260 270 280 290 TRTVDVTASD AAWEVYRLSD IIYDPDSRFA GLLFFFDATG NRQVVQIDAP LIPSFM
Theoretical pI/Mw: 6.11 / 32.93462
15 Verifikasi keberhasilan penyisipan gen pada pGEM-T Easy dengan amplifikasi plasmid rekombinan pGEM-spmoB dengan primer T7 dan SP6 menghasilkan amplikon yang berukuran ±1000 pb (Gambar 13a). Ukuran ini lebih
besar dari ukuran sisipan dikarenakan primer tersebut turut serta mengamplifikasi daerah Multiple Cloning Site (MCS) yang berukuran 126 pb. Pemotongan plasmid rekombinan dengan enzim EcoRI menghasilkan dua fragmen DNA yang berukuran ±3000 pb dan ±900 pb (Gambar 13b). Ukuran ini telah sesuai dengan
ukuran gen spmoB yang disisipkan. Hasil sekuensing daerah sisipan menunjukkan gen spmoB tidak mengalami mutasi (Lampiran 7).
Gambar 13 Verifikasi gen spmoB dengan menggunakan plasmid dari bakteri E. coli DH5α transforman yang mengandung pGEM-spmoB (a)
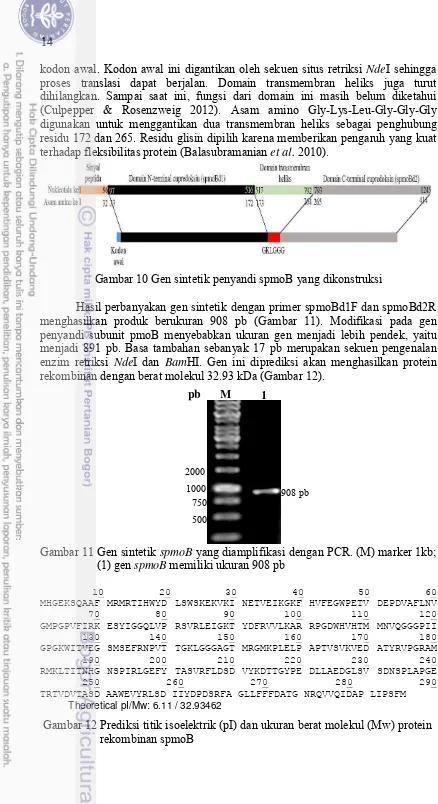
Fragmen gen spmoB diperoleh dengan memotong plasmid pGEM-spmoB menggunakan enzim NdeI dan BamHI. Pemotongan ini bertujuan untuk mengeluarkan gen sisipan dari vektor pGEM dengan kedua ujung fragmen gen yang sticky. Kedua ujung fragmen ini kemudian diligasikan dengan vektor ekspresi pET-15b. Proses ligasi ini akan menghasilkan plasmid pET15b rekombinan yang membawa gen spmoB dengan ukuran 6599 pb (Gambar 14).
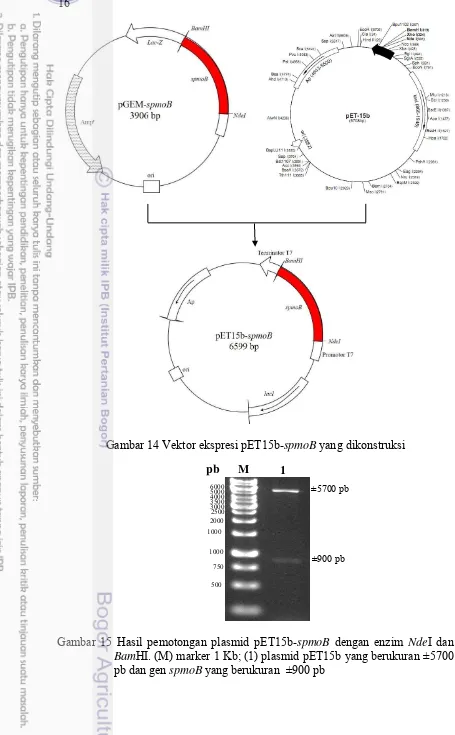
Vektor pET-15b yang telah membawa gen spmoB diintroduksikan pada E. coli BL21 (DE3) dengan metode renjatan panas (heat shock). Verifikasi keberhasilan proses introduksi dilakukan dengan PCR koloni. Plasmid dari E. coli BL21 (DE3) yang menunjukan hasil positif kemudian diisolasi dan dipotong kembali dengan enzim NdeI dan BamHI untuk memastikan bahwa sisipan telah sesuai dengan target. Hasil pemotongan ini menunjukan dua pita yang berukuran ±5700 pb dan ±900 pb (Gambar 15). Pita yang berukuran ±5700 pb merupakan plasmid pET15b tanpa sisipan dan pita berukuran ±900 pb merupakan gen spmoB.
16
Gambar 14 Vektor ekspresi pET15b-spmoB yang dikonstruksi
Gambar 15 Hasil pemotongan plasmid pET15b-spmoB dengan enzim NdeI dan BamHI. (M) marker 1 Kb; (1) plasmid pET15b yang berukuran ±5700 pb dan gen spmoB yang berukuran ±900 pb
M 1 pb
1000 750
500
±5700 pb
±900 pb 2000 2500
17
Analisis Ekspresi dan Pengujian Aktivitas Oksidasi Metan
Gen spmoB diekspresikan di E. coli BL21 (DE3). Kelebihan menggunakan E. coli sebagai organisme inang telah diketahui dengan baik. Bakteri ini memiliki kinetika pertumbuhan yang cepat, densitas sel yang tinggi, media pertumbuhan yang telah tersedia dengan komponen yang relatif murah, dan transformasi yang cepat dan mudah (Rosano & Ceccarelli 2014). Dibandingkan dengan pertumbuhan M. capsulatus (Bath) yang relatif lambat, batas densitas sel teoritis E. coli dalam kultur cair diperkirakan sekitar 200 g sel L-1 (Lee 1996; Shiloach & Fass 2005).
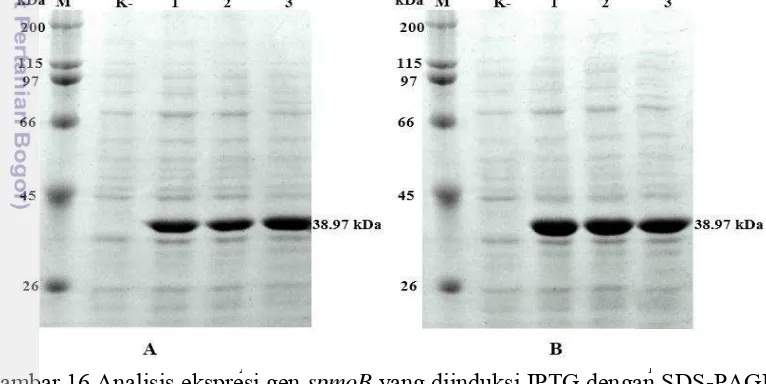
Ekspresi berlebih gen spmoB di E. coli dengan induksi IPTG telah berhasil dilakukan. Hal ini ditunjukkan dengan munculnya pita protein rekombinan pada E. coli BL21 (DE3) rekombinan yang telah diinduksi dengan IPTG (Gambar 16). Berdasarkan perhitungan nilai mobilitas relatif, protein rekombinan spmoB berukuran sekitar 38.97 kDa (Lampiran 8). Ukuran ini berbeda dari prediksi oleh ExPASy yang memperkirakan berat molekul spmoB sebesar 32.9 kDa. Perbedaan ini disebabkan karena adanya penambahan 6 residu histidin dan beberapa asam amino pada bagian hulu protein dari vektor pET15b. Balasubramanian et al. (2010) mengekspresikan gen ini dengan vektor pET21b dan menghasilkan ukuran protein rekombinan yang sama. Ukuran protein spmoB rekombinan menjadi lebih kecil dari pada ukuran subunit pmoB (45 kDa) karena terdapat penghilangan domain transmembran heliks.
Gambar 16 Analisis ekspresi gen spmoB yang diinduksi IPTG dengan SDS-PAGE (A) inkubasi pada suhu 27 oC (B) inkubasi pada suhu 37oC.
Keterangan: (M) Marker protein; (K-) bakteri transforman yang tidak diinduksi oleh IPTG; (1) induksi IPTG 0.1 mM; (2) induksi IPTG 0.5 mM; (3) induksi IPTG 1.0 mM
Beberapa konsentrasi IPTG dan suhu inkubasi digunakan untuk mengetahui pengaruhnya terhadap ekspresi. Pada beberapa kasus, suhu inkubasi sangat berpengaruh terhadap ekspresi protein rekombinan di E. coli (Gopal & Kumar 2013). Konsentrasi IPTG yang digunakan untuk menginduksi represor-promotor regulator lac dapat memberikan pengaruh nyata terhadap ekspresi (Khlebnikov & Keasling 2002). Pada beberapa protein diperlukan induksi transkripsi yang lambat
18
(dengan konsentrasi IPTG yang lebih rendah) dan beberapa kasus yang lain diharapkan produksi protein dengan jumlah yang lebih tinggi (Sambrook & Russel 2001). Oleh karena itu, pada penelitian ini dilakukan pengamatan pengaruh konsentrasi IPTG terhadap ekspresi gen spmoB. Hasilnya menunjukkan bahwa konsentrasi IPTG 0.1 mM, 0.5 mM, dan 1.0 mM IPTG dapat menginduksi ekspresi gen rekombinan dengan baik. Pada penelitian ini, konsentrasi 0.1 mM IPTG dapat menginduksi ekspresi sebaik konsentrasi 0.5 mM dan 1.0 mM. Hasil ini mengindikasikan bahwa konsentrasi 0.1 mM sudah mampu mengikat hampir keseluruhan protein represor lac. Kenaikan konsentrasi IPTG tidak akan memberikan perbedaan tingkat ekspresi jika seluruh protein represor lac di dalam sel telah diikat oleh IPTG. Bakteri E. coli BL21 (DE3) rekombinan juga dapat mengekspresikan protein rekombinan pada suhu 27 oC dan 37 oC. Kemampuan
untuk mengekspresikan protein rekombinan yang diinginkan dalam jumlah yang tinggi memungkinkan untuk karakterisasi biokimia, aplikasi dalam proses industri, dan juga aplikasi lainnya (Rosano & Ceccarelli 2014).
Pengukuran aktivitas spmoB pada E. coli BL21 (DE3) transforman telah
dilakukan. Kultur bakteri yang telah diinduksi oleh IPTG selama 24 jam pada media LB digunakan untuk mengukur aktivitas oksidasi metan. Aktivitas bakteri rekombinan adalah sebesar 0.114 mmol/mL kultur/jam. Pada E. coli BL21 (DE3)
non transforman tidak terbentuk metanol selama pengukuran aktivitas spmoB. Kontrol percobaan ini memastikan bahwa aktivitas oksidasi metan pada E. coli
BL21 (DE3) rekombinan disebabkan oleh aktivitas protein rekombinan spmoB. Aktivitas spmoB ini masih belum optimal. Tingkat ekspresi yang sangat tinggi diduga sebagai penyebab penurunan aktivitas ini. Pada ekspresi berlebih, akumulasi protein intraselular yang sangat cepat serta dalam jumlah yang besar akan meningkatkan kemungkinan terjadinya agregasi dan hal ini memicu ketidak stabilan protein (Jonasson et al. 2002; Palomares et al. 2004). Pembentukan
agregat protein ini diketahui sebagai inclusion bodies. Aktivitas protein yang
membentuk inclusion bodies dapat dikembalikan dengan proses pelipatan ulang
secara in vitro.
SIMPULAN DAN SARAN
Simpulan
Gen spmoB berhasil diklon dan diekpresikan secara berlebih di E. coli BL21 (DE3) tanpa menyebabkan toksisitas. Ekspresi ini dapat dilakukan pada suhu 27
oC dan 37 oC dengan konsentrasi IPTG 0.1 mM, 0.5 mM, dan 1.0 mM yang
ditambahkan ke dalam kultur. Protein spmoB rekombinan yang dihasilkan berukuran 38.9 kDa. Aktivitas oksidasi metan bakteri rekombinan adalah sebesar 0.114 mmol/mL kultur/jam.
Saran
19
DAFTAR PUSTAKA
Balasubramanian R, Smith SM, Rawat S, Yatsunyk LA, Stemmler TL, Rosenzweig AC. 2010. Oxidation of methane by a biological dicopper. Nat 465: 115-121.
Benstead J, King GM, Williams HG. 1998. Methanol promotes atmospheric methane oxidation by methanotrophic cultures and soils. Appl Environ Microbiol 64: 1091-1098.
Bowman JP, Sly LI, Nichols PD, Hayward AC. 1993. Revised taxonomy of the methanotrophs: description of Methylobacter gen. nov., emendation of Methylococcus, validation of Methylosinus and Methylocystis species, and a proposal that the family Methylococcaceae includes only the group I methanotrophs. Int J Syst Bacteriol 43: 735–753.
Chistoserdova L, Vorholt JA, Lidstrom ME. 2005. A genomic view of methane oxidation by aerobic bacteria and anaerobic archaea. Genome Biol 6: 208-214.
Culpepper MA, Rosenzweig AC. 2012. Architecture and active site of particulate methane monooxygenase. Critical Rev Bioch Mol Biol 47 (6): 483-492. Dedysh SN, Liesack W, Khmelenina VN, Suzina NE, Trotsenko YA, Semrau JD,
Bares AM, Panikov NS, Tiedje JM. 2000. Methylocella palustris gen. nov., a new methane-oxidizing acidophilic bacterium from peat bogs, representing a novel subtype of serine pathway methanotrophs. Int J Syst Evol Micr 50: 955–969.
Dedysh SN, Knief C, Dunfield PF. 2005. Methylocella species are facultatively methanotrophic. J Bacteriol 187: 4665-4670.
Dumont MG, Radajewski SM, Miguez CB, McDonald R, Murrel JC. 2006. Identification of a complete methane monooxygenase operon from soil by combining stable isotope probing and metagenomic analysis. Environ Microbiol 8 (7): 1240-1250.
Gilbert B, McDonald IR, Finch R, Stafford GP, Nielsen AK, Murrell JC. 2000. Molecular analysis of the pmo (particulate methane monooxygenase) operons from two type II methanotrophs. App Environ Microb 66 (3): 966-975.
Gopal GJ, Kumar A. 2013. Strategies for the Production of recombinant protein in Escherichia coli. Prot J 32: 419-425.
Gou X, Xing HH, Luo M, jiang H, Han B, Wu H, Wang L, Zhang F. 2006. Functional expression of the particulate methane monooxygenase gene in recombinant Rhodococcus erythropolis. FEMS Microbiol 263: 136-141. Hakemian AS, Kondapalli KC, Telser J, Hoffman BM, Stemmler TL, Rosenzweig
AC. 2008. The metal centers of particulate methane monooxygenase from Methylosinus trichosporium OB3b. Biochem 47 (26): 6793-6801. Hakemian AS, Rosenzweig A. C. 2007. The biochemistry of methane oxidation.
Ann Rev Biochem 76: 223–241.
Han B et al. 2009. Diversity and activity of methanotrophs in alkaline soil from a Chinese coal mine. FEMS Microbiol Ecol 70: 40-51.
20
Jonasson P, Liljeqvist S, Nygren PA, Stahl S. 2002. Genetic design for facilitated production and recovery of recombinant proteins in Escherichia coli. Biotechnol App Biochem 35: 91 – 105.
Khlebnikov A, Keasling JD. 2002. Effect of lacY expression on homogeneity of induction from the P(tac) and P(trc) promoters by natural and synthetic inducers. Biotechnol Prog 18: 672-674.
Lee SY. 1996. High cell-density culture of Escherichia coli. Trend Biotechnol 14: 98-105.
Lelieveld J, Crutzem PJ, Bruhl C. 1993. Climate effects of atmospheric methane. Chemosphere 26:739–768.
Lieberman RL, Rosenzweig AC. 2004. Biological methane oxidation: regulation, biochemistry, and active site structure of particulare methane monooxygenase. Chritical Rev Biochem Mol Biol 39: 149-164.
Lieberman RL, Rosenzweig AC. 2005a. Crystal structure of membrane-bound mettaloenzyme that catalyses the biological oxidation of methane. Nat 434: 177-182.
Lieberman RL, Rosenzweig AC. 2005b. The quest for the particulate methane monooxygenase active site. Dalton Tran 2005: 3390–3396.
Matheson LJ, Jahnke LL, Oremland RS. 1997. Inhibition of methane oxidation by Methylococcus capsulatus with hydrochlorofluorocarbons and fluorinated methanes. Appl Environ Microbiol 63: 2952-2956.
Mcdonald IR, Murrell JC. 1997. The particulate methane monooxygenase gene pmoA and its use as functional gene probe for methanotrophs. FEMS Microbiol Lett 156: 205-210.
Moroux B, Walker JE. 1996. Over-production of protein in Escherichia coli: mutant host that allow synthesis of some membrane proteins and globular proteins at high levels. J Mol Biol 260: 289-298.
Murrel JC. 1994. Molecular genetics of methane oxidation. Biodegradation 5: 145-159.
Murrell JC, Gilbert B, and McDonald IR. 2000. Molecular biology and regulation of methane monooxygenase. Arch Microbiol 173:325–332.
Ngunyen HH, Ellieott SJ, Yip JHK, Chan SI. 1997. The particulate methan monooxygenase from Methylococcus capsulatus (Bath) is a novel copper-containing three-subunit enzyme. J Biol Chem 273 (14): 7957-7966.
Nielsen AK, Gerdes K, Murrell JC. 1997. Copper-dependent reciprocal transcriptional regulation of methane monooxygenase genes in Methylococcus capsulatus and Methylosinus trichosporium. Mol Microbiol 25: 399–409.
Palomares LA, Mondaca SE, Raminez OT. 2004. Production of recombinant proteins, challenges and solutions. Method Mol Biol 267: 15 – 51.
Patel RN, Hoare DS. 1971. Physiological studies of methane and methanol oxidizing bacteria: oxidation of C-1 compound by Methylococcus capsulatus. J Bacteriol 107 (1): 187-192.
21 Ricke P, Erkel C, Kube M, Reinhardt R, Liesack W. 2004. Comparative analysis of the conventional and novel pmo (particulate monooxygenase) operon from Methylocystic strain SC2. Appl Environ Microbiol 70: 3055-3063. Rosano GL, Ceccarelli EA. 2014. Recombinant protein expression in Escherichia
coli: advance and challenges. Front Microbiol 5: 1–17. Rosenzweig AC. 2015. Breaking methane. Nat 518 (7539): 309-310.
Rusmana I, Akhdiya A. 2009. Isolation and characterization of methanorophic bacteria from rice fields. Biotropia 16 (2): 71-78.
Semrau JD, Chistoserdov A, Lebron J, Costello A, Davagnino J, Kenna JE, Holmes AJ, Finch R, Murrell JC. 1995. Particulate methane monooxygenase genes in methanotrophs. J Bacteriol 177: 3071–3079. Sambrook J, Russell DW. 2001. Molecular Cloning, a Laboratory Manual. Cold
spring Harbor laboratory press, cold spring Harbor, New York.
Semrau JD, Dispirito AA, Yoon S. 2010. Methanotrophs and copper. FEMS Microbiol Rev 34: 496-531.
Shiloach J, Fass R. 2005. Growing E. coli to high cell density - a historical perspective on method development. Biotechnol Adv 23: 345-357.
Smith SM, Rawat S, Telser J, Hoffman BM, Stemmler TL, Rosenzweig AC. 2011a. Chrystal structure and characterization of particulate methane monooxygenase from Methylocystis species strain M. Biochem 50 (47): 10231-10240.
Smith SM, Balasubramanian R, Rosenzweig AC. 2011b. Metal reconstitution of particulate methane monooxygenase and heterologous expression of the pmoB subunit. Methods Enzymol 495: 195-210.
Stolyar S, Costello AM, Peeples TL, Lidstorm ME. 1999. Role of multiple gene copies in particulate methane monooxygenase activity in the methane-oxidizing bacterium Methylococcus capsulatus (Bath). Microbiol 145: 1235-1244.
Stolyar S, Franke M, Lidstrom ME. 2001. Expression of individual copies of Methylococcus capsulatus (Bath) particulate methane monooxygenase genes. J Bacteriol 183 (5): 1810-1812.
Tol RSJ, Heintz RJ, Lammers PEM. 2003.Methane emission reduction: an application of FUND. Climatic Change 57:71–98.
Ward N, Larsen O, Sakwa J, Bruseth L, Khouri H, Durkin S, Dimitrov G, Jiang L, Scanlan D, Kang KH, Lewis M, Nelson KE, Methe B, Wu M, Heidelberg JF, Paulsen IT, Fouts D, Ravel J, Tettelin H, Ren Q, Read T, DeBoy RT, Seshadri R, Salzberg SL, jensen HB, Birkeland NK, Nelson WC, Dodson RJ, Grindhaug SH, Holt I, Eidhammer I, Jonase I, Vanaken S, Utterback T, Feldblyum TV, Fraser CM, Lillehaug JR, Eisen JA. 2004. Genomic insights into Methanotrophy: the complete genome sequence of Methylococcus capsulatus (Bath). Plos Biology 2: 1616-1628.
Yu SSF, Chen KHC, Tseng MYH, Wang YS, Tseng CF, Chen YJ, Huang DS & Chan SI. 2003. Production of high-quality particulate methane monooxygenase in high yields from Methylococcus capsulatus (Bath) with a hollow-fiber membrane bioreactor. J Bacteriol 185: 5915–5924. Zahn JA, DiSpirito AA. 1996. Membrane-associated methane monooxygenase
22
LAMPIRAN
Lampiran 1 Pembuatan sel kompeten dengan perlakuan CaCl2 (Sambrook &
Russel 2001)
Sebanyak 2 mL biakan E.coli DH5α umur 12 jam dikulturkan ke dalam 40 mL media LB. Kultur diinkubasi selama 2-3 jam pada suhu 37 °C sampai nilai Optical Density (OD600) mencapai 0.45-0.5. Selanjutnya kultur diambil sebanyak 1,5 mL dan disentrifugasi pada kecepatan 5000 rpm selama 15 menit. Pelet diresuspensi dengan 1 mL bufer transformasi. Sampel diinkubasi di dalam es selama 15 sampai 30 menit, kemudian disentrifugasi pada 5000 rpm selama 5 menit pada suhu 4 °C. Pelet diresuspensi kembali dengan 250 µL bufer
10% amonium persulfat (APS) 0.20 0.05
23
Lampiran 5 Komposisi reagen Sodium Nitroprusside (SNP)
Sebanyak 100 mL sodium nitroprusside 10% (m v-1), 100 mL kalium
ferisianida 10% (m v-1), dan 100 mL NaOH 5% (m v-1) dicampurkan dalam
wadah. Campuran tersebut kemudian dilarutkan dengan akuades hingga 600 mL. Konsentrasi sodium nitroprusside adalah 5.6 × 10-2 M dan NaOH 0.21 M.
Sebelum digunakan, reagen SNP disimpan pada suhu 4 oC dan dijauhkan dari
cahaya langsung.
Lampiran 6 Prosedur pengukuran kadar metanol dengan reagen SNP
Sebanyak 1 mL sampel ditambahkan 6 mL reagen SNP dalam tabung berukuran 12.5 mL. Campuran diencerkan hingga mencapai volume 12.5 mL dengan akuades dan diaduk hingga homogen. Larutan kemudian diinkubasi pada suhu 27 oC selama 15 menit. Nilai absorbansi larutan diukur pada panjang
gelombang 481 nm.
Lampiran 7 Urutan nukleotida gen spmoB M. capsulatus (Bath)
24
T R T V D V T A S D A A W E V 766 TACCGTCTGTCCGACATCATCTACGATCCGGACAGCCGTTTCGCC Y R L S D I I Y D P D S R F A 811 GGTCTGCTGTTCTTCTTCGACGCCACTGGCAACCGCCAGGTCGTC G L L F F F D A T G N R Q V V 856 CAGATCGACGCACCGCTGATCCCGTCGTTCATGTAA 891
Q I D A P L I P S F M *
Lampiran 8 Perhitungan berat molekul sampel Jarak migrasi
sampel (cm) Nilai Rf sampel Log BM (kDa) BM
25
RIWAYAT HIDUP
Penulis dilahirkan di Cianjur pada tanggal 14 Januari 1990 dari ayah Abdul Madjid dan ibu Ai Djubaedah (Alm). Penulis merupakan anak keempat dari empat bersaudara.