PEMODELAN DAN SIMULASI DINAMIKA MOLEKUL
HUMAN MATRIX
METALLOPROTEINASE
9
(hMMP9) UTUH SEBAGAI TARGET VIRTUAL
PENEMUAN LIGAN PADA
CATALYTIC SITE
MAUPUN
HEMOPEXIN-LIKE
DOMAIN
TESIS
Diajukan untuk Memenuhi Salah Satu Syarat
Memperoleh Gelar Magister Farmasi (M.Farm.)
Program Studi Magister Farmasi
Diajukan oleh:
Roy Gunawan Wicaksono, S.Farm. Nomor Induk Mahasiswa:
188122101
FAKULTAS FARMASI UNIVERSITAS SANATA DHARMA
YOGYAKARTA 2020
PEMODELAN DAN SIMULASI DINAMIKA MOLEKUL
HUMAN MATRIX
METALLOPROTEINASE
9
(hMMP9) UTUH SEBAGAI TARGET VIRTUAL
PENEMUAN LIGAN PADA
CATALYTIC SITE
MAUPUN
HEMOPEXIN-LIKE
DOMAIN
TESIS
Diajukan untuk Memenuhi Salah Satu Syarat
Memperoleh Gelar Magister Farmasi (M.Farm.)
Program Studi Magister Farmasi
Diajukan oleh:
Roy Gunawan Wicaksono, S.Farm. Nomor Induk Mahasiswa:
188122101
FAKULTAS FARMASI UNIVERSITAS SANATA DHARMA
YOGYAKARTA 2020
}ERSETUJUAN PEMBIMBING
PEMODELAN DAN SIMULASI
DINAMIKA
MIOLF]KTJLHUMAN MATRD(
METALLOPROTEINASE 9 (hMNTPg)IITUH
SEBAGAT TARGET VTRTUALPENEMUAN LIGAN PADA CATALYTIC
SITEiMAI|PIITNHEMOPEXIN
DOfuLAINTesis yang diajukan oleh:
Roy Crunawan Wicaksono, S.Farm. (f 88122101)
T€lah disetujui tar.ggdZr 19 ot"t,
Dosen ing Utarna
Enade Ph.D., Apt.
PENGESAIIAN TESIS BERJUDT]L
PEMODELAIT DAN
SIMULASI DINAMIKA MOLEKIJL
HUMANMATRD(
METALLOPROTEINASE 9 (htvfrvfp9) UTUH SEBAGAI TARGETVIRTUAL
PENEMUAN LIGA}I PADA CITALYTIC SITEMAUI{JN HEMOPHfiN-LIEE
DOMAIN
oleh:
Roy Gunawan Wicaksono, S.Farm. (188122101)
Dipertahankan di hadapan panitia penguji tesis Fakultas Farmasi
Universitas Sanata Dharma
Pada tanggal 23 lanuai 2020
Mengetahui Fakultas Farmasi Universitas Sanata Dharma
Hartini, Apt.
Panitia Penguji:
l. Enade Perdana
Istyastono, Ph.D., Apt2. Dr. Rini Dwiastuti, Apt. 3. Maywan Hariono, Ph.D., Apt.
tr!?
,: utl
>-_ | {'2-_
HALAMAN PERSEMBAHAN
Tesis ini dipersembahkan untuk Tuhan Yesus Kristus, Ibu, Bapak,
Kakak , Adik, Novia Yosin dan teman-teman sehingga dapat
PERNYATAAN
KEASLLA.NKARYA
Saya merryatakan dengan sesungguhnya bahrva tesis yang saya
tulis iui
tidak memuatkarya atau bagian karya orang lain, kecuali yang
telah disebutkan
dalamkutipan dan daftar pustaka, sebagaimaru layaknya karya ilmiah.
Apabila di
kemudian hari ditemukan indikasi plagiarisme dalam naskah ini, maka saya bersedia menanggung segala sanksi sesuai peraturan perundang-undangan yang berlaku.s/.10
Y ogyal(arta,
..../1.
.LEMBAR PERNYATAAN
PERSETUJUANPUBLIKASI
IGRYA ILML{H
UNTUK KEPENTINGAN AKADEIUIS
Yang bertanda tangan di bawah
ini, saya
mahasiswa Universitas Sanata Dharma: Nama : Roy Gunawan Wicaksono. S.Farm.Nomor
Mahasiswa :
188122101Demi
pengembanganilmu
pengekhuan,saya memberikan kepada
perpustakaanUniversitas Sanata Dharma karya ilmiah saya yang
be{udul "PEMODELAN DAN
.SIMULASI DINAMIKA MOLEKUL ITUMAN
MATRIX
'METALLOhROTEINASE
9
(hMMpg)
UTUH SEBAGAI
TARGET
I'IRTUAL
PENEMUAN
LIG,{N
PADA
CATALYTIC
SITE
MAUPUN
HEMOPEXIN
DOMAIN" beserta perangkat
yang diperlukan
(bila
ada). Dengan demikian sayamemberikan kepada Perpustakaan Universitas Sanata Dharma hak untuk menyimpan,
mengalihkan
dalam bentuk
lain,
mengelolanyadalam bentuk pangkalan
data, mendish'ibusikan secara terbatas, dan mempubli kasikannyadi intemet
atau media lainuntuk kepentingan akademis tanpa
perlu
memintaizin
saya atau memberi royal4t
kepada saya selama tetap mencantumkan nama saya sebagai penulis.
Demikian pernyataan
ini
saya buat dengan sebenar-benamya.Dibuat di Yogyakarta Pada tang!;al
,/t, .19
Yang menyatakan,)tt
ftM-t
€el
PRAKATA
Puji dan syukur penulis haturkan ke hadirat Tuhan Yang Maha Esa karena atas
berkat dan rahmat kasih-Nya penulis dapat menyelesaikan tesis yang berjudul
“
PEMODELAN DAN SIMULASI DINAMIKA MOLEKUL
HUMAN MATRIX
METALLOPROTEINASE
9
(hMMP9) UTUH SEBAGAI TARGET VIRTUAL
PENEMUAN LIGAN PADA
CATALYTIC SITE
MAUPUN
HEMOPEXIN-LIKE
DOMAIN
”
dengan baik dan lancar. Tesis ini disusun sebagai salah satu syarat
memperoleh gelar Magister Farmasi (M.Farm.) Program Studi Magister Farmasi.
Penulis berharap agar tesis ini dapat berguna bagi para pembaca, menjadi sumber
pengetahuan tentang dinamika molekul pada MMP-9 dan menjadi inspirasi untuk
melakukan penelitian yang lebih berkembang nantinya.
Penulis tesis ini tidak lepas dari banyak bantuan, dukungan, semangat, dan saran
dari berbagai pihak. Oleh karena itu, penulis ingin mengucapkan terima kasih kepada:
1.
Ibu Dr. Yustina Sri Hartini, Apt. selaku Dekan Fakultas Farmasi Universitas
Santa Dharma.
2.
Ibu Aris Widayati, M.Si., Ph.D., Apt. selaku Kaprodi Magister Farmasi Fakultas
Farmasi Universitas Sanata Dharma.
3.
Bapak Enade Perdana Istyastono, Ph.D., Apt. selaku dosen pembimbing yang
telah memberikan ilmu, saran dan bimbingan dengan sabar dalam penelitian ini.
4.
Ibu Dr. Rini Dwiastuti, Apt. selaku dosen penguji yang telah bersedia
memberikan saran bagi penelitian ini.
5.
Bapak Maywan Hariono, Ph.D., Apt. selaku dosen penguji yang telah bersedia
memberikan saran bagi penelitian ini.
6.
Keluarga penulis yang telah memberikan doa, semangat dan dukungan.
7.
Mas F.A Ottok yang telah membantu kelancaran dalam penelitian tesis.
8.
Teman-teman Magister Farmasi yang telah memberika ilmu dan saran.
9.
Novia Yosin Parura yang selalu memberi dukungan dan semangat kepada
penulis selama kuliah dan selalu mendampingin saat penulis membutuhkan
bantuan.
membangun
dari para
pembaca.Akhir
kata, semoga tesisini
dapat bermanfaat dan selamat membaca..
9/
c.o YogJlakarta,/r
.
. Penulis,4+
Roy Gunawan Wicaksono. S.Farm.DAFTAR ISI
PERSETUJUAN PEMBIMBING...i
HALAMAN PENGESAHAN...ii
HALAMAN PERSEMBAHAN...iii
PERNYATAAN KEASLIAN KARYA...iv
LEMBAR PERNYATAAN PUBLIKASI...v
PRAKATA...vi
DAFTAR ISI...viii
DAFTAR GAMBAR...x
DAFTAR LAMPIRAN...xi
INTISARI...xii
ABSTRACT
...xiii
1. PENDAHULUAN...1
2. TINJAUAN PUSTAKA...2
2.1
Matrix Metalloproteinase 9
...2
2.2
Catalytic Site
dan
Hemopexin Domain
...3
2.3 MMP-9 Sebagai Target Molekul Dalam Penyembuh Luka...4
2.4 MMP-9 Sebagai Target Molekul Dalam Terapi Kanker...5
3. METODE PENELITIAN...7
3.1 Bahan dan Instrumen...7
3.1.1 Bahan...7
3.1.2 Instrumen...7
3.2 Tahapan Simulasi...7
3.2.1 Preparasi...7
3.3.2 Analisis Hasil...8
4. HASIL DAN PEMBAHASAN...8
4.1 Kualitas Hasil Pemodelan Homologi...9
4.2 Stabilitas MMP-9 Terhadapat Ligan...10
4.3
The Free Energy of Binding
(
ΔG bind)
...
11
5. KESIMPULAN DAN SARAN...12
6. DAFTAR PUSTAKA...13
7. BIOGRAFI PENULIS...18
DAFTAR GAMBAR
Gambar 1. Struktur
Matrix Metalloproteinase
9
...2
Gambar 2. Sruktur
Hemopexin-like domain...
3
Gambar 3. Visualisasi
Catalytic site
interaksi sistein dengan Zn yang menjaga enzim
dalam keadaan tidak aktif...4
Gambar 4. Struktur N-2-(biphenyl-4-ylsulfonyl)-N-2(isopropyloxy)-acetohydroxamic
(CC27)...5
Gambar 5. Visualisasi
Catalytic site
dengan ligan CC27 (cyan), HIS
401, HIS
405, HIS
411(biru), GLU
402(merah) dan Zn (ungu)...5
Gambar 6. Dimer
hemopexin-like domain...6
Gambar 7. Struktur senyawa Duf-02 sebagai inhibitor PEX...6
Gambar 8. Visualisasi
Hemopexin-like domain
dengan SO
4(kuning), GLN
666(hijau),
ARG
618(biru), GLU
572dan GLU
526(merah)...7
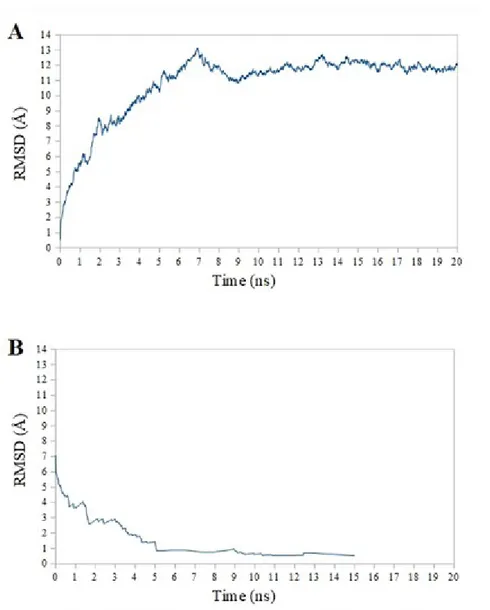
Gambar 9. Nilai RMSD
backbone
MMP-9 (kiri) dan RMSD tiap 5ns (kanan); (A)
MMP-9 CC27, (B) MMP-9 Duf-02 dan (C) MMP-9 tanpa ligan secara
berurutan...10
Gambar 10.
The free energy of binding
CC27 dengan MMP-9 selama 5,05 ns hingga
10.05 ns dengan nilai terendah -12,75 kcal/mol pada 6,40 ns...11
Gambar 11. Visualisasi 1000 pose ligan hasil penambatan molekul...12
DAFTAR LAMPIRAN
Lampiran 8.1. Status makalah ilmiah dan makalah ilmiah...18
Lampiran 8.2.
md_run_20ns_20ss.mcr...
28
Lampiran 8.3.
becalculation.mcr
...36
INTISARI
Enzim
Matrix Metalloproteinase 9
(
MMP-9) berpotensi sebagai target ligan
untuk penemuan obat kanker ataupun penyembuh luka pada
Diabetic Foot Ulcer
(DFU)
.
Saat ini, belum terdapat kristal struktur dari MMP-9 secara utuh, sehingga
simulasi pemodelan MMP-9 dalam penemuan ligan hanya menggunakan fragmen dari
MMP-9 baik
catalytic site
maupun
hemopexin-like domain
(PEX). Struktur MMP-9
secara utuh dapat dibuat model 3 dimensi menggunakan pemodelan homologi. Model
MMP-9 secara utuh dapat digunakan dalam penemuan ligan baik pada
catalytic site
maupun PEX.
Tujuan dari penelitian ini untuk membuat target virtual MMP-9 secara utuh
dalam penemuan ligan baik pada
catalytic site
maupun PEX. Simulasi pemodelan
molekul model MMP-9 dilakukan menggunakan YASARA-structure
dengan
AMBER14
force field.
Simulasi pemodelan molekul dilakukan hingga 20 ns dengan
snapshot
10 ps. Hasil simulasi dianalisis secara semi kuantitatif dengan melihat: (1)
Kualitas model homologi (i) kebenaran dari enantiomer; (ii) tidak adanya ikatan
cis
-peptida; (iii) kesesuaian penamaan; (iv) panjang ikatan; (v) sudut ikatan; (vi) sudut
dihedral; (vii) gugus planar. (2)
Stabilitas MMP-9 berdasarkan nilai RMSD backbone
tiap 5 ns, apabila RMSD backbone selama 5 ns dibawah 1Å maka MMP-9 dengan ligan
dikatakan stabil. (3) The free energy of binding (ΔG bind) masing-masing simulasi
dihitung menggunakan macro YASARA-structure. (4)
Nilai RMSD hasil penambatan
ulang dihitung untuk melihat hasil penambatan yang reprodusibel dengan nilai
≤
2
Å
.
Hasil pemodelan molekul MMP-9 dengan ligan Duf-02 dan tanpa ligan tidak
stabil. Hanya MMP-9 dengan ligan CC27 yang stabil dengan mencapai
equilibrium
pada 5,05 ns, MMP-9 telah mencapai
equilibrium
apabila RMSD
backbone
tiap 5ns
dibawah 1
Å.
The free energy of binding
terendah pada pose waktu 6,40 ns dengan nilai
-12,75 kcal/mol. Hasil penambatan ulang ligan pada pose waktu 6,40 ns memiliki hasil
yang reprodusibel dengan RMSD penambatan ulang 1000 kali berada di bawah 2
Å.
Kesimpulan dari hasil penelitian yang dilakukan bahwa model yang dibuat dapat
digunakan sebagai target penapisan virtual pada
catalytic site.
Kata kunci:
MMP-9,
catalytic site
,
hemopexin-like domain
, pemodelan molekul,
YASARA-structure
ABSTRACT
Matrix Metalloproteinase 9 (MMP-9) enzyme has the potential as a
ligand-target for the drug discovery of cancer or wounds healing in Diabetic Foot Ulcer
(DFU). At present, there is no complete crystal structure of MMP-9, so the MMP-9
modeling simulation in the ligand drug discovery only uses fragment from MMP-9 both
the catalytic site and hemopexin-like domain (PEX). The whole MMP-9 structure can
be made in the 3-dimensional model using homology modeling. The whole MMP-9
model can be used in ligand drug discovery at the catalytic site and PEX.
This study aims to create a full model MMP-9 as a virtual target in ligand drug
discovery at the catalytic site and PEX. Molecular dynamics simulation of MMP-9
models using YASARA-structure with an AMBER14 force field. Molecular dynamics
simulations running to 20 ns with a 10 ps snapshot. The results of simulations are
analyzed semi-quantitatively by looking at: (1) The quality of the homology model (i)
the truth of the enantiomer; (ii) absence of cis-peptide bonds; (iii) appropriateness of
naming; (iv) bond length; (v) bond angles; (vi) dihedral angle; (vii) planar groups. (2)
The stability of MMP-9 based on the RMSD backbone value every 5 ns, if the RMSD
backbone in 5 ns is below 1Å then MMP-9 with ligands is stable. (3) The free energy of
binding (ΔG bind) of each simulation is calculated using the YASARA-structure macro.
(4) The RMSD result from redocking is calculated to see the reproducible of docking,
reproducible if the RMSD redocking below ≤ 2Å.
The results of molecular modeling of MMP-9 with Duf-02 ligand and without
ligand are unstable. Only MMP-9 with CC27 ligand was stable by reaching equilibrium
at 5.05 ns, MMP-9 has reached equilibrium if the RMSD backbone every 5ns is below
1Å. The lowest free energy of binding at time 6.40 ns with a value of -12.75 kcal/mol.
Ligand redocking at 6.40 ns is reproducible, with 1000 times of RMSD redocking being
under 2Å. The conclusion from the results of this research that the model can be used as
a target virtual screening on a catalytic site.
Keywords:
MMP-9, catalytic site, hemopexin-like domain, molecular modeling,
YASARA-structure
1.PENDAHULUAN
1.1 Latar Belakang
Human matrix metalloproteinases
(hMMPs) merupakan enzim yang berperan
dalam proses
remodeling
extracellular matrix
(ECM) dan membran dasar
1. Enzim
MMPs tersusun dari
regulatory
,
catalytic site
dan
hemoxpexin-like domain
2.
Berdasarkan substrat enzim MMPs dikategorikan menjadi
collagenase
,
gelatinase
,
stromelysis
,
matrilysins
dan
membrane
-
types
3. Enzim MMP-9 yang termasuk
gelatinase
merupakan suatu protein yang berfungsi mendegradasi
extracellular matrix
dan ikut
berperan dalam proses penyembuhan luka. Aktivitas MMP-9 yang tidak terkontrol akan
menyebabkan proses penyembuhan luka menjadi lambat pada komplikasi penyakit
kronis seperti
Diabetic Foot Ulcer
(DFU)
4. Enzim MMP-9 berpotensi sebagai target
protein dalam penemuan obat penyembuh luka pada DFU
5. Penyembuhan luka
ditargetkan pada
catalytic site
dengan menghambat sisi
catalytic site
untuk
mendegradasi ECM, sehingga membantu proses penyembuhan luka pada penderita
diabetes. Selain sebagai target penyembuh luka MMP-9 juga digunakan sebagai target
dalam anti kanker
2. Diketahui bahwa ekspresi berlebih dari MMP-9 memiliki korelasi
terhadap proliferasi dan peningkatan ukuran sel kanker
6.
Hemopexin-like domain
(PEX)
pada MMP-9 akan berinteraksi dengan reseptor
clusters of differentiation
44 (CD44)
dan memicu proliferasi sel
7. Suatu ligan inhibitor PEX
akan menghambat pembentukan
dimer, sehingga tidak dapat berinteraksi dengan reseptor CD44 dan proliferasi sel tidak
terjadi
8.
Selama ini penghambatan MMP-9 secara umum ditargetkan pada
catalytic site.
Namun, adanya homologi yang tinggi pada
catalytic site
pada semua MMP
menyebabkan target menjadi tidak selektif
35. Semua kerja MMP menjadi dihambat,
padahal MMP juga dibutuhkan dalam memetabolisme senyawa di dalam tubuh seperti
inflamasi
11. Ketidak selektifan ini berdampak pada gagalnya hampir semua MMP
inhibitor pada fase klinik. Ke gagalan pada fase klinik dikarenakan munculnya efek
samping yaitu nyeri otot. Karena itu, dikembangkan target lain dari MMP9 yang
mempunyai homologi cukup rendah.
Hemopexin-like domain
pada
MMP-9
hanya
memiliki 25% kemiripan dengan MMP yang lain. Sehingga
hemopexin-like domain
menjadi target yang berpotensi sebagai inhibitor MMP-9
17.
Pemodelan molekul dalam penemuan ligan dengan target MMP-9 tidak
menggunakan struktur MMP-9 secara utuh, dikarenakan hingga saat ini belum terdapat
kristal struktur MMP-9 secara utuh
9. Sehingga pemodelan molekul hanya menggunakan
fragmen dari MMP-9 baik
catalytic site
maupun PEX. Simulasi pemodelan molekul
yang dilakukan secara terpisah tersebut menyebabkan tidak dapat diketahui afinitas
ligan tersebut berada pada
catalytic site
atau PEX
10.
Penelitian pemodelan molekul penemuan ligan pada MMP-9 dilakukan dengan
ligan ditambatkan pada masing-masing sub unit pada MMP-9 utuh. Tujuan dari
penelitian ini untuk memodelkan target virtual MMP-9 secara utuh sebagai target
penemuan ligan baik pada
catalytic site
maupun PEX. Hasil pemodelan ini diharapkan
dapat digunakan sebagai target virtual pemodelan molekul untuk penemuan ligan yang
spesifik pada
catalytic site
maupun PEX. Luaran yang diharapkan dari penelitian ini
adalah publikasi artikel ilmiah, setidaknya di jurnal nasional terakreditasi.
2. TINJAUAN PUSTAKA
2.1
Matrix Metalloproteinase 9
Enzim
matrix metalloproteinase 9
(MMP-9) merupakan proteinase yang
berfungsi mendegradasi
extracellular matrix
(ECM)
11. Enzim MMP-9 memiliki struktur
terdiri dari
propeptide
,
catalytic site
,
fibronectin,
linker
dan
hemopexin-like domain
(PEX) (Gambar 1)
5. Enzim MMP-9 dibentuk dalam keadan
zymogen
(tidak aktif)
12.
Propeptide
menjaga MMP-9 dalam keadaan tidak aktif dengan adanya interaksi sistein
dengan Zn
3. Ketika ada substrat yang akan masuk, interaksi sistein dengan Zn akan
putus dan MMP-9 menjadi aktif.
Catalytic site
merupakan bagian dari sisi aktif yang
memiliki Zn dan berperan dalam proses enzimatiknya
11.
Gambar 1. Struktur Matrix Metalloproteinase9
Enzim MMP-9 secara normal dalam proses fisiologis memiliki peran sebagai perbaikan
sel, proliferasi, migrasi dan angiogenesis bahkan penyembuhanan luka
13. Dalam
Propeptide Catalytic site Linker Hemopexin domain
penyakit kanker terdapat peningkatan ekspresi MMP-9 yang berpengaruh dalam
penyebaran sel kanker
14dan peningkatan ekspresi MMP-9 memiliki korelasi terhadap
proliferasi dan peningkatan ukuran sel kanker
15. Inhibitor MMP-9 menghambat migrasi
dan proliferasi sel dengan cara mengganggu pembentukan dimer PEX
sehingga sinyal
kepada reseptor
clusters of differentiation
44 untuk mengalami migrasi dan proliferasi
tidak terjadi
13.
2.2
Catalytic Site
dan
Hemopexin-like Domain
Hemopexin-like domain
(PEX) berupa
4
beta-propeller
dihubungkan dengan
catalytic site
oleh
hinge
/
linker region
yang fleksibel
16(Gambar 2).
Sub unit
PEX akan
membentuk dimer dan berinteraksi dengan reseptor
clusters of differentiation
44 pada
permukaan membran sel. Interaksi yang tejadi akan memicu sel mengalami proliferasi
sehingga sel kanker dapat tumbuh dan menyebar
17. Suatu senyawa inhibitor PEX akan
menghambat pembentukan dimer PEX. Penurunan kadar MMP-9 menunjukkan
penurunan ukuran dan metastasis dari sel kanker, oleh karena itu penemuan obat kanker
didesain sebagai inhibitor PEX
18.
Gambar 2. Struktur Hemopexin domain19
Catalytic site
merupakan sisi aktif dari MMP-9 yang berperan mendegradasi
extracellular matrix
11dan memiliki ion Zn sebagai kofaktor
3.
Catalytic site
biasa
digunakan sebagai target penyembuh luka dengan menghambat proses enzimatiknya
20.
Interaksi sistein dengan Zn menjaga enzim tidak aktif, ketika interaksi tersbut terputus
kantung ikatan pada
catalytic site
seolah-olah terbuka dan MMP-9 menjadi aktif
(Gambar 3). Substrat yang masuk ke dalam
catalytic site
akan dihidrolisis dengan
adanya molekul air yang berinteraksi dengan asam glutamat dengan bantuan Zn
7.
Secara
normal mekanisme kerja enzim MMPs akan dihambat oleh endogen spesifik seperti
tissue inhibitor of metalloproteinases
(TIMPs)
1.
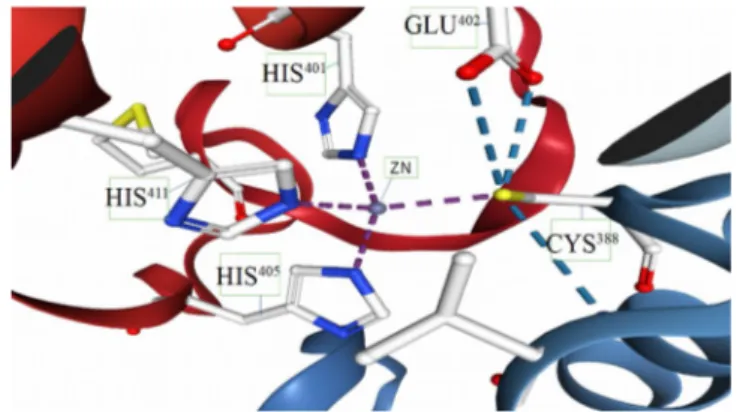
Gambar 3. Visualisasi Catalytic site interaksi sistein dengan Zn yang menjaga enzim dalam keadaan tidak aktif21
2.3 MMP-9 Sebagai Target Molekul Dalam Penyembuh Luka
Pada hari pertama luka MMP-9 disekresikan oleh sel inflamasi dan
membersihkan luka dari
extracellular matrix
(ECM) yang rusak
6. Ketika MMP-9
mendegradasi ECM, respon inflamasi akan terjadi hal ini karena mediator inflamasi
seperti prostalglandin dan bradikinin terlepas saat degradasi ECM
11. Selain
mendegradasi ECM, enzim MMP-9 juga berperan dalam pembentukan dari membran
sel
22. Selama perbaikan
sel MMP-9 juga berperan dalam mendegradasi matrik yang
tidak beraturan. Aktivitas MMP-9 yang tidak terkontrol akan mempengaruhi
keseimbangan penghancuran dan pembentukan membran sel. Penghancuran membran
sel yang tidak dihambat akan menurukan proses penyembuhan
luka
23.
Penemuan ligan sebagai inhibitor MMP-9 dalam penyembuh luka ditargetkan
pada
catalytic site
24dengan membuat enzim menjadi tidak aktif. Asam amino penting
pada pada
catalytic site
adalah HIS
401, HIS
405dan HIS
411dan GLU
402. Interaksi yang
penting sebagai inhibitor MMP-9 pada
catalytic site
adalah interaksi phi-phi antara
gugus aromatis pada ligan dengan histidin, interaksi ion dengan Zn dan interaksi
hidrogen dengan glutamat
25.
Salah satu ligan yang diketahui sebagai inhibitor MMP-9 pada
catalytic site
adalah
N-2-(biphenyl-4-ylsulfonyl)-N-2(isopropyloxy)-acetohydroxamic
yang memiliki
IC
50200nM (Gambar 4)
.
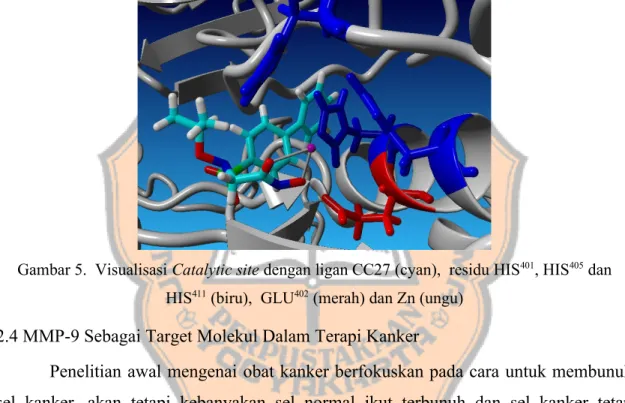
Ligan tersebut memiliki gugus aromatis yang berinteraksi
(Gambar 5). Salah satu ciri ligan inhibitor
catalytic site
pada MMP-9 yaitu memiliki
gugus hidroxamat. Ligan yang berada pada
catalytic site
akan bersifat kompetitif
menyebabkan substrat tidak dapat masuk
26.
Gambar 4.Struktur N-2-(biphenyl-4-ylsulfonyl)-N-2(isopropyloxy)-acetohydroxamic (CC27)
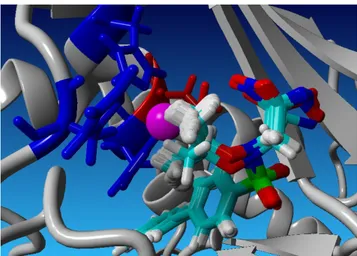
Gambar 5. Visualisasi Catalytic site dengan ligan CC27 (cyan), residu HIS401, HIS405 dan HIS411 (biru), GLU402 (merah) dan Zn (ungu)
2.4 MMP-9 Sebagai Target Molekul Dalam Terapi Kanker
Penelitian awal mengenai obat kanker berfokuskan pada cara untuk membunuh
sel kanker, akan tetapi kebanyakan sel normal ikut terbunuh dan sel kanker tetap
resisten
6. Pada tahun 90-an, MMP-9 telah teridentifikasi dan berpotensi sebagai target
penemuan ligan untuk obat kanker
25. Saat ini, penemuan obat kanker didesain sebagai
inhibitor
hemopexin-like domain
(PEX) untuk menghambat aktivitas MMP-9
27.
Sub unit
PEX akan membentuk dimer (Gambar 6) dan berinteraksi dengan
reseptor
clusters of differentiation
44 (CD44) pada permukaan sel
28. Interaksi antara
PEX dengan reseptor CD44 akan mengaktivasi
epidermal growth factor receptor
(EGFR) menyebabkan sel mengalami proliferasi
6. Ekspresi MMP-9 yang tidak
terkontrol akan menyebabkan sel kanker tumbuh tidak terkendali
12.
Gugus hidroxamat Cincin
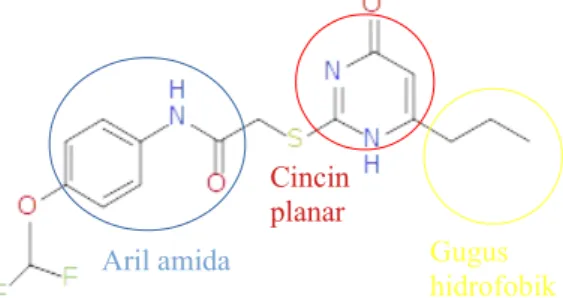
Salah satu ligan yang diketahui sebagai inhibitor MMP-9 dan telah diuji adalah
senyawa sintesis oleh Dufour
et al.
(Gambar 7). Struktur senyawa Duf-02 memiliki
gugus aril amida dan hidrofobik yang mampu berikatan pada kantung ikatan
hemopexin-like domain.
Gugus aril amida akan berada didalam kantung ikatan diantara
4 bilah dikarenakan memiliki gugus amida yang menyerupai substrat. Gugus -NH dari
aril amida akan berinteraksi hidrogen dengan GLU
618. Selain itu terdapat atom polar
seperti O maupun N pada cincin planar yang mampu berinteraksi dengan ARG
618. Ligan
tersebut bekerja dengan mencegah pembentukan dimer yang berinteraksi secara
hidrofobik sehingga PEX tidak dapat berinteraksi dengan reseptor CD44
7. Bagian
penting
hemopexin-like domain
terletak pada
blade
4 dengan ARG
618yang berinteraksi
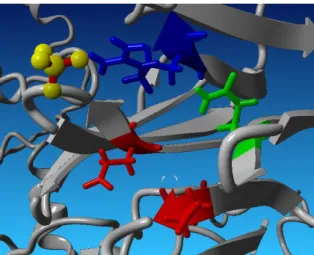
dengan SO
4.. Asam amino yang diduga penting sebagai target ligan adalah GLN
666,
ARG
618, GLU
572dan GLU
526. Inhibitor PEX untuk obat kanker mencegah agar
proliferasi sel kanker tidak terjadi dan mencegah terjadinya pertumbuhan serta
metastasis dari sel kanker
18.
Gambar 6. Dimer hemopexin-like domain
Gambar 7. Struktur senyawa Duf-02 sebagai inhibitor PEX
Aril amida
Cincin planar
Gugus hidrofobik
Gambar 8.Visualisasi Hemopexin-like domain dengan SO4 (kuning) dan residu GLN666 (hijau), ARG618 (biru), GLU572 dan GLU526 (merah)
3. METODE PENELITIAN
3.1 Bahan dan Instrumen
3.1.1 Bahan
File
pdb
berisi struktur model MMP-9 utuh hasil pemodelan homologi
menggunakan YASARA-structure
32yang diambil dari doi
:10.17632/xj7yt48jwb.1
33.
Ligan yang digunakan berupa ligan CC27 (Gambar 4) dari kristal struktur
4H3X.pdb
yang diambil dari Protein Data Bank (PDB; https://www.rcsb.org/) dan senyawa
(Gambar 7) yang diuji oleh Dufour
et al
20.
3.1.2 Instrumen
Simulasi pemodelan molekul menggunakan perangkat lunak YASARA-structure
versi 19.1.27.
Force field
yang digunakan dalam simulasi menggunakan AMBER14.
Server Fakultas Farmasi Universitas Sanata Dharma (IP 172.23.1.176) dengan
spesifikasi sistem operasi Linux dengan 16
core
RAM 16 GB dan komputer lab kimia
komputasi dengan
processor
Intel® Core
TMi5-7500, 8 GB RAM dengan sistem operasi
Windows 10 professional 64-bit.
3.2 Tahapan Simulasi
3.2.1 Preparasi
Masing-masing ligan CC27 dan Duf-02 ditambatkan pada
catalytic site
dan
hemopexin-like domain
model MMP-9. Ruang penambatan molekul pada
catalytic site
ditentukan 5
Å
dari ligan CC27 yang telah ada pada model MMP-9 sedangkan
hemopexin-like domain
5 A dari asam amino ARG
574. Pemilihan ARG
574sebagai center
dikarenakan ARG
574berada di antara asam amino GLN
666, ARG
618, GLU
572dan GLU
526.
Sehingga terdapat 4 sistem yang dilakukan dinamika molekul selama 20 ns
menggunakan macro
md_run_20ns_ss10.mcr
(Lampiran 8.2)
29. Hasil simulasi dinamika
molekul
selanjutnya dianalisis menggunakan
md_analyze.mcr
standar bawaan
YASARA-structure
untuk melihat RMSD. Hasil dinamika molekul selama 5 ns setelah
mencapai
equilibrium
diambil untuk dianalisis
The free energy of binding (ΔG)
34.
Pose
waktu model MMP-9 yang memiliki energi terendah kemudian diambil dan digunakan
sebagai target penambatan molekul ligan sebanyak 1000 kali menggunakan macro
dock_run_1000.mcr
(Lampiran 8.4).
3.3.2 Analisis Hasil
Analisis secara semi kuantitatif dilakukan dengan melihat: (1) Kualitas model
homologi yang telah dilakukan dinamika molekul selama 10 ns; (i) kebenaran dari
enantiomer; (ii) tidak adanya ikatan
cis
-peptida; (iii) kesesuaian penamaan; (iv) panjang
ikatan; (v) sudut ikatan; (vi) sudut dihedral; (vii) gugus planar
32. (2) Stabilitas MMP-9
dianalisis dengan menghitung nilai
root mean square deviation
(RMSD)
backbone
tiap
5 ns, jika RMSD backbone ≤ 2Å selama 5 ns maka dianggap MMP-9 telah stabil
30.
Apabila RMSD backbone tidak berada di bawah 2Å selama 5 ns, maka MMP-9
dikatakan tidak stabil dan tidak dilakukan analisis binding energi. (3) The binding free
energy (ΔG) dihitung menggunakan becalculation.mcr (Lampiran 8.3). (4)
Reprodusibel penambatan
ligan dilihat dengan menghitung RMSD penambatan ulang
dibanding pose ligan pada pose binding energi terendah (ligan referensi) menggunakan
dock_run_1000.mcr
(Lampiran 8.4), dikatakan reprodusibel jika nilai RMSD
≤ 2Å
31.
4. HASIL DAN PEMBAHASAN
Penelitian ini bertujuan untuk membuat target virtual yang dapat digunakan
sebagai penapisan virtual MMP-9. Target yang dibuat berupa model MMP-9 secara
utuh sebagai target penapisan virtual baik pada
catalytic site
maupun
hemopexin-like
domain
. Ligan CC27 dan senyawa Duf-02 ditambatkan pada masing-masing sub unit.
Selanjutnya MMP-9 dengan ligan yang berada pada masing-masing kantung ikatan
dilakukan dinamika molekul selama 20 ns. Simulasi tanpa adanya ligan ditambahkan
sebagai pembanding pengaruh adanya ligan terhadap stabilitas MMP-9.
Dinamika molekul MMP-9 dengan ligan CC27 maupun Duf-02 pada
hemopexin-like domain
tidak dapat dianalisis. Tidak adanya ligan referensi pada
kantung ikatan
hemopexin-like domain
menyebabkan tidak diketahuinya letak kantung
secara spesifik. Sehingga penambatan molekul pada
hemopexin-like domain
yang
dilakukan berdasarkan prediksi pada ARG
574sebagai pusat. Ligan yang di tambatkan
pada sisi yang kurang tepat kemungkinan menyebabkan terjadinya interaksi yang tidak
dapat diprediksi oleh YASARA-structure. Dinamika molekul dengan kedua ligan yang
ditambatkan pada
hemopexin-like domain
tersebut tidak dapat dianalisis oleh
YASARA-structure
.
4.1 Kualitas Hasil Pemodelan Homologi.
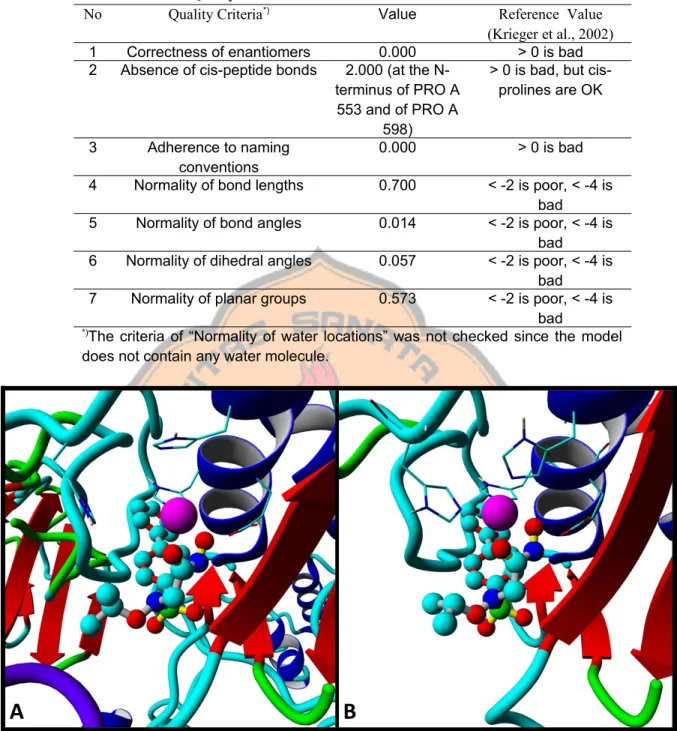
Model yang yang telah dibuat tidak terdapat kesalahan penamaan. Panjang
ikatan, sudut ikatan, sudut dihedral dan gugus planar tidak lebih kecil dari -2 (Tabel 1).
Terdapat 2 ikatan
cis-
peptida pada model MMP-9 yang dibuat
.
Adanya ikatan
cis
-peptida terdapat pada PRO
553dan PRO
598. Dua ikatan
cis-
peptida
tersebut berada pada
hemopexin-like domain
. Ikatan
cis-
peptida prolin masih dapat diterima. Secara
keseluruhan kualitas model MMP-9 utuh yang dibuat cukup baik.
Tabel 1. Hasil analisis kualitas model pemodelan homologi
Kualitas Model Homologi
32Parameter
Nilai
Kebenaran enantiomer 0 (> 0 bad)
Adanya ikatan cis-peptida 2 cis-peptida prolin (> 0 bad ) Kesesuaian penamaan 0 (> 0 bad)
Panjang ikatan 0,727 (< -2 poor, < -4 bad) Sudut ikatan 0,183 (< -2 poor, < -4 bad) Sudut dihedral 0,176 (< -2 poor, < -4 bad) Gugus planar 0,505 (< -2 poor, < -4 bad)
4.2 Stabilitas MMP-9 Terhadap Ligan
Simulasi dinamika molekul MMP-9 dikatakan stabil apabila selama 5 ns nilai
RMSD
backbone
tiap 5 ns di bawah 1
Å
30. Nilai RMSD
backbone
tiap 5 ns pada
MMP-9
dengan ligan Duf-02 dan tanpa ligan tidak ada yang di bawah 1
Å selama 5 ns.
Oleh
karena itu MMP-9 dengan ligan Duf-02 dan tanpa ligan dinyatakan tidak stabil.
Sedangkan RMSD MMP-9 dengan ligan CC27 setelah 5,05 ns berada dibawah 1
Å
hingga 20 ns. Pada 0 ns hingga 5,05 ns dianggap merupakan proses equilibrium dan
setelah 5,05 ns MMP-9 dengan ligan CC27 telah stabil
(Gambar 9). Adanya ligan CC27
pada
catalytic site
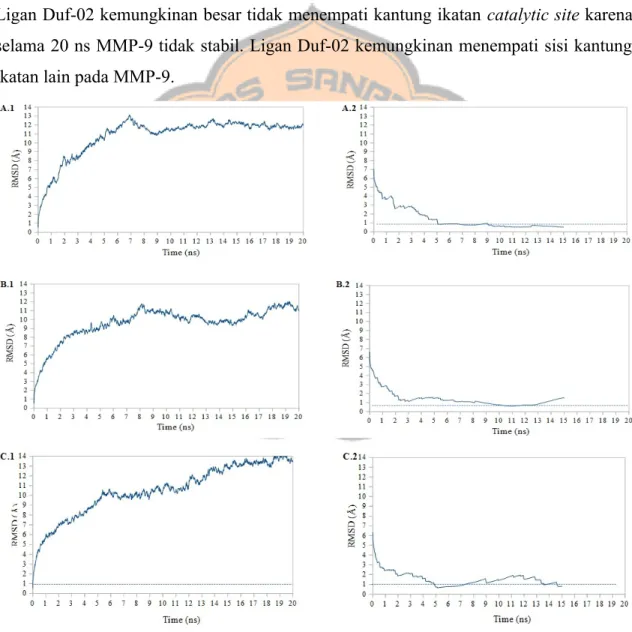
meningkatkan stabilitas dari MMP-9 dibanding MMP-9 tanpa ligan.
Ligan Duf-02 kemungkinan besar tidak menempati kantung ikatan
catalytic site
karena
selama 20 ns MMP-9 tidak stabil. Ligan Duf-02 kemungkinan menempati sisi kantung
ikatan lain pada MMP-9.
Gambar 9.Nilai RMSD backbone (kiri) dan RMSD tiap 5ns (kanan); (A) MMP-9 ligan CC27, (B) MMP-9 ligan Duf-02 dan (C) MMP-9 tanpa ligan secara berurutan
4.3
The Binding Free Energy
(
ΔG bind)
Simulasi dinamika molekul MMP-9 dengan ligan CC27 selanjutnya dilakukan
analisis
the binding free energy
. Model MMP-9 dengan ligan Duf-02 tidak dilakukan
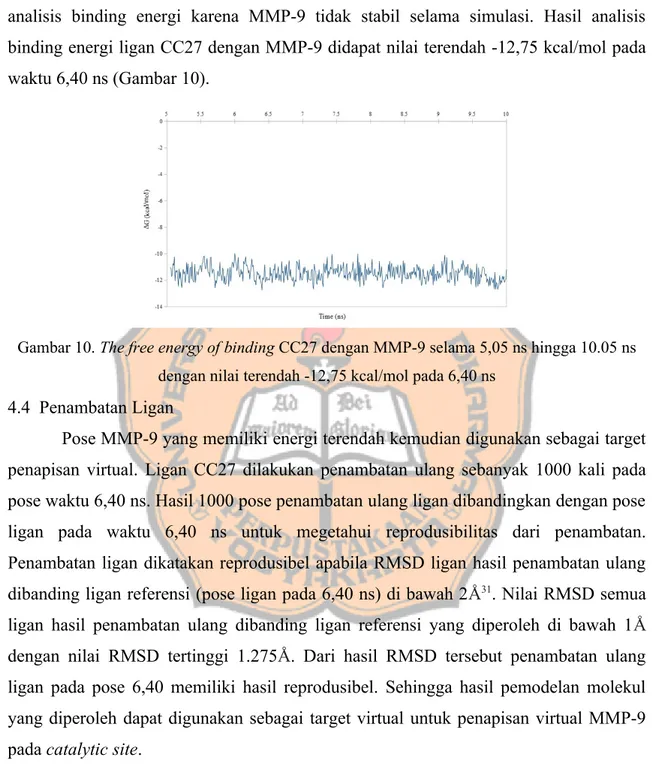
analisis binding energi karena MMP-9 tidak stabil selama simulasi. Hasil analisis
binding energi ligan CC27 dengan MMP-9 didapat nilai terendah -12,75 kcal/mol pada
waktu 6,40 ns (Gambar 10).
Gambar 10. The free energy of binding CC27 dengan MMP-9 selama 5,05 ns hingga 10.05 ns dengan nilai terendah -12,75 kcal/mol pada 6,40 ns
4.4 Penambatan Ligan
Pose MMP-9 yang memiliki energi terendah kemudian digunakan sebagai target
penapisan virtual. Ligan CC27 dilakukan penambatan ulang sebanyak 1000 kali pada
pose waktu 6,40 ns. Hasil 1000 pose penambatan ulang ligan dibandingkan dengan pose
ligan pada waktu 6,40 ns untuk megetahui reprodusibilitas dari penambatan.
Penambatan ligan dikatakan reprodusibel apabila RMSD ligan hasil penambatan ulang
dibanding ligan referensi (pose ligan pada 6,40 ns) di bawah 2
Å
31. Nilai RMSD semua
ligan hasil penambatan ulang dibanding ligan referensi yang diperoleh di bawah 1
Å
dengan nilai RMSD tertinggi 1.275
Å
. Dari hasil RMSD tersebut penambatan ulang
ligan pada pose 6,40 memiliki hasil reprodusibel. Sehingga hasil pemodelan molekul
yang diperoleh dapat digunakan sebagai target virtual untuk penapisan virtual MMP-9
pada
catalytic site
.
Gambar 11. Visualisasi 1000 pose ligan (cyan) hasil penambatan molekul
5. KESIMPULAN DAN SARAN
Berdasarkan protokol yang telah dilakukan hasil model MMP-9 dapat digunakan
sebagai target penapisan virtual pada
catalytic site
, akan tetapi tidak untuk
hemopexin-like domain
. Perlu dilakukan analisis pada
hemopexin-like domain
menggunakan
tools
lain seperti
plugin
trajectory pada VMD dan/atau dilakukan pemodelan homologi
dengan
hemopexin-like domain
yang telah ditambah ligan.
Hemopexin domain
dengan
adanya ligan pada kantung ikatan akan memastikan ligan yang ditambatkan tepat pada
tempatnya.
6. DAFTAR PUSTAKA
111
1.
Keeling J, Herrera GA. Human matrix metalloproteinases: Characteristics and
pathologic role in altering mesangial homeostasis.
Microsc Res Tech
.
2008;71(5):371-379. doi:10.1002/jemt.20565
2.
Roeb E, Schleinkofer K, Kernebeck T, Pötsch S, Jansen B, Behrmann I, Matern
S, Grotzinger J. The matrix metalloproteinase 9 (MMP-9) hemopexin domain is a
novel gelatin binding domain and acts as an antagonist.
J Biol Chem
.
2002;277(52):50326-50332. doi:10.1074/jbc.M207446200
3.
Hidalgo M, Eckhardt SG. Development of matrix metalloproteinase inhibitors in
cancer therapy.
J Natl Cancer Inst
. 2001;93(3):178-193.
doi:10.1093/jnci/93.3.178
4.
Ayuk SM, Abrahamse H, Houreld NN. The role of matrix metalloproteinases in
diabetic wound healing in relation to photobiomodulation.
J Diabetes Res
.
2016;2016:1-9. doi:10.1155/2016/2897656
5.
Vandooren J, Knoops S, Buzzo JLA, Boon L, Martens E, Opdenakker G,
Kolaczkowska E. Differential inhibition of activity, activation and gene
expression of MMP-9 in THP-1 cells by azithromycin and minocycline versus
bortezomib: a comparative study.
PLoS One
. 2017;12(4):1-19.
doi:10.1371/journal.pone.0174853
6.
Farina AR, Mackay AR. Gelatinase B/MMP-9 in tumour pathogenesis and
progression.
Cancers (Basel)
. 2014;6(1):240-296. doi:10.3390/cancers6010240
7.
Ezhilarasan R, Jadhav U, Mohanam I, Rao JS, Gujrati M, Mohanam S. The
hemopexin domain of MMP-9 inhibits angiogenesis and retards the growth of
intracranial glioblastoma xenograft in nude mice.
Int J Cancer
.
2009;124(2):306-315. doi:10.1002/ijc.23951
8.
Gentile E, Liuzzi GM. Marine pharmacology: therapeutic targeting of matrix
metalloproteinases in neuroinflammation.
Drug Discov Today
. 2017;00(00):1-15.
doi:10.1016/j.drudis.2016.09.023
9.
Hou J, Wang Y, Gao W, Yao W, Yao Q, Zhang J. Screening for the selective
inhibitors of MMP-9 from natural products based on pharmacophore modeling
and molecular docking in combination with bioassay experiment, hybrid
QM/MM calculation, and MD simulation.
J Biomol Struct Dyn
. 2018;0(0):1-15.
doi:10.1080/07391102.2018.1509019
10.
Kaczanowski S, Zielenkiewicz P. Why similar protein sequences encode similar
three-dimensional structures?
Theor Chem Acc
. 2010;125(3-6):643-650.
doi:10.1007/s00214-009-0656-3
11.
Van Lint P, Libert C. Chemokine and cytokine processing by matrix
metalloproteinases and its effect on leukocyte migration and inflammation.
J
Leukoc Biol
. 2007;82(6):1375-1381. doi:10.1189/jlb.0607338
12.
Mali AV, Joshi AA, Hedge MV, Kadam SS. Enterolactone suppresses
proliferation, migration and metastasis of MDA-MB-231 breast cancer cells
through inhibition of uPA induced plasmin activation and MMPs-mediated ECM
remodeling.
Asian Pacific J Cancer Prev
. 2017;18(4):905-915.
doi:10.22034/APJCP.2017.18.4.905
13.
Vu TH, Werb Z. Matrix metalloproteinases: effectors of development and normal
physiology.
Genes Dev
. 2000;14:2123-2133. doi:10.1101/gad.815400.GENES
14.
Merchant N, Nagaraju GP, Rajitha B, Lammata S, Jella KK, Buchwald ZS,
Lakka SS, Ali AN. Matrix metalloproteinases: their functional role in lung
cancer.
Carcinogenesis
. 2017;38(8):766-780. doi:10.1093/carcin/bgx063
15.
Bhowmick NA, Neilson EG, Moses HL. Stromal fibroblasts in cancer initiation
and
progression
Neil.
Nature
.
2011;432(7015):332-337.
doi:10.1038/nature03096.Stromal
16.
Glasheen MB, Kabra TA, Page-McCaw A. Distinc functions for the catalytic and
hemopexin domains of drosophila matrix metalloproteinase.
PNAS
.
2009;302(6784):1053-1055. doi:10.1073/pnas.0804171106
17.
Dufour A, Cao J, Kuscu C, Dufour A, Zucker S, Sampson NS. Role of matrix
metalloproteinase-9 dimers in cell migration.
J Biol Chem
.
2010;285(46):35944-35956. doi:10.1074/jbc.m109.091769
18.
Alford VM, Kamath A, Ren X, Kumar K, Gan Q, Awwa M, Tong M, Seeliger
MA, Cao J, Ojima I, Sampson NS. Targeting the hemopexin-like domain of
latent matrix metalloproteinase-9 (proMMP-9) with a small molecule inhibitor
prevents the formation of focal adhesion junctions.
ACS Chem Biol
.
2017;12:2788-2803. doi:10.1021/acschembio.7b00758
19.
Cha H, Kopetzki E, Huber R, Lanzendörfer M, Brandstetter H. Structural basis of
the adaptive molecular recognition by MMP9.
J Mol Biol
.
2002;320(5):1065-1079. doi:10.1016/S0022-2836(02)00558-2
20.
Hingorani DV, Lippert CN, Crisp JL, Savariar EN, Hasseimann JPC, Kuo C,
Nguyen QT, Tsien RY, Whitney MA, Ellies LG. Impact of MMP-2 and MMP-9
enzyme activity on wound healing , tumor growth and RACPP cleavage.
PLoS
One
. 2018;13(9):1-17. doi:10.1371/journal.pone.0198464
21.
Elkins PA, Yen SH, Smith WW, Janson CA, Alessio KJD, McQueney MS,
Cummings MD, Romanic AM. Structure of the C-terminally truncated human
ProMMP9, a gelatin-binding matrix metalloproteinase.
Acta Crystallogr Sect D
Biol Crystallogr
. 2002;58(7):1182-1192. doi:10.1107/S0907444902007849
22.
Lebert DC, Squirrell JM, Rindy J, Broadbridge E, Lui Y, Zakrzewska A, Elicelri
KW, Meijer AH, Huttenlocher A. Matrix metalloproteinase 9 modulates collagen
matrices and wound repair.
Co Biol
. 2015;142:2136-2146.
doi:10.1242/dev.121160
23.
Hariono M, Yuliani SH, Istyastono EP, Riswanto FDO, Adhipandito CF. Matrix
metalloproteinase 9 (MMP9) in wound healing of diabetic foot ulcer: molecular
target and structure-based drug design.
Biochem Pharmacol
. 2018;9:1-47.
doi:10.1016/j.wndm.2018.05.003
24.
Sabino F, Keller U auf dem K. Matrix metalloproteinases in impaired wound
healing.
Met Med
. 2015;2:1–8. doi:https//dx.doi.org/10.2147/MNM.S68420
25.
Winer A, Adams S, Mignatti P. Matrix metalloproteinase inhibitors in cancer
therapy: turning past failures into future successes.
Mol Cancer Ther
.
2018;17(6):1147-1155. doi:10.1158/1535-7163.MCT-17-0646
26.
García-Pardo A, Opdenakker G. Nonproteolytic functions of matrix
metalloproteinases in pathology and insights for the development of novel
therapeutic inhibitors.
Met Med
. 2015;2:19-28. doi:10.2147/MNM.S63629
27.
Cathcart J, Pulkoski-Gross A, Cao J. Targeting matrix metalloproteinases in
cancer: Bringing new life to old ideas.
Genes Dis
. 2015;2(1):26-34.
doi:10.1016/j.gendis.2014.12.002
28.
Grass GD, Tolliver LB, Bratoeva M, Toole BP. CD147 , CD44 and the epidermal
growth factor receptor (EGFR) signaling pathway cooperate to regulate breast
epithelial cell invasiveness.
J Biol Chem
. 2013;288(36):26089-26104.
doi:10.1074/jbc.M113.497685
29.
Krieger E, Vriend G. New ways to boost molecular dynamics simulations.
J
Comput Chem
. 2015;36(13):996-1007. doi:10.1002/jcc.23899
30.
Liu K, Watanabe E, Kokubo H. Exploring the stability of ligand binding modes
to proteins by molecular dynamics simulations.
J Comput Aided Mol Des
.
2017;31(2):201-211. doi:10.1007/s10822-016-0005-2
31.
Liu K, Kokubo H. Exploring the stability of ligand binding modes to proteins by
molecular dynamics simulations: a cross-docking study.
J Chem Inf Model
.
2017;57(10):2514-2522. doi:10.1021/acs.jcim.7b00412
32.
Krieger E, Koraimann G, Vriend G. Increasing the precision of comparative
models with YASARA NOVA - A self-parameterizing force field.
Proteins
Struct Funct Genet
. 2002;47(3):393-402. doi:10.1002/prot.10104
33.
Istyastono EP. Model of full human matrix metalloproteinase 9 with ligands from
PDB:4H3X and PDB:1ITV.
Mendeley Data
. 2019. doi:10.17632/xj&yt48jwb.1
34. Prasasty VD, Istyastono EP. Structure-based design and molecular dynamics
simulations of pentapeptide AEYTR as a potential acetylcholinesterase inhibitor.
Indones.
J. Chem
. 2019;1–7.
https://doi.org/10.22146/ijc.46329
35. Adhipandito CF, Ludji DPKS, Aprilianto E, Jenie RI, Al-Najjar B, Hariono M.
Matrix Metalloproteinase9 as the protein target in anti-breast cancer drug
discovery: an approach by targeting hemopexin domain.
Futur J Pharm Sci
.2019;5(1):1-15.
7. BIOGRAFI PENULIS
Roy Gunawan Wicaksono, S.Farm. Lahir di
Sleman pada tanggal 28 Februari 1996. Riwayat
pendidikan formal yang telah ditempuh oleh penulis adalah
SD N 1 Nanggulan (2002-2008). SMP N 7 Yogyakarta
(2008-2011). SMA N 1 Nanggulan (2011-2014). Sarjana
Farmasi di Universitas Sanata Dharma Yogyakarta
(2014-2018). Setamat dari pendidikan Sarjana Farmasi, penulis
melanjutkan pendidikan Magister Farmasi di Fakultas
Farmasi Universitas Sanata Dharma Yogyakarta. Selama
menempuh pendidikan Magister Farmasi penulis menjadi dosen luar biasa (DLB)
praktikum biokimia di Universitas Sanata Dharma Yogyakarta.
8.
LAMPIRAN
8.1 Status makalah ilmiah dan makalah ilmiah
JURNAL FARMASI SAINS DAN KOMUNITAS, month year, pp Vol. X No. X p-ISSN 1693-5683; e-ISSN 2527-7146
doi: